2. 130021 长春,吉林大学:药学院生物医学工程系
2. Department of Biomedical Engineering, School of Pharmacy, Jilin University, Changchun, Jilin Province, 130021, China
室管膜瘤是常见的小儿脑肿瘤,占儿童神经上皮性肿瘤的2%~9%,占所有颅内肿瘤的6%~12%[1-2]。室管膜瘤可以沿着神经轴发展,尤其是靠后颅窝、幕上和脊髓。幕下室管膜瘤在儿童中更常见,成人通常患脊髓室管膜瘤[3]。目前室管膜瘤的治疗方法主要是手术切除、术后辅助放化疗[4]。然而,当术中监测电位降到50%以下或肿瘤牵涉3个以上的脊髓节段水平时,治疗结果并不乐观。近年来关于室管膜瘤的研究日益增多,然而,室管膜瘤的高风险信号通路、室管膜瘤发生发展的核心致病基因以及针对其靶向治疗却很少被提及。生物信息学的发展,采用一系列高通量大数据的研究手段,特别是结合基因表达微阵列技术,为研究疾病的分子机制提供了新的思路[5]。本研究通过对大量临床样本数据的挖掘,针对室管膜瘤,进行致病高风险因子即核心驱动基因的筛选研究,并探究室管膜瘤致病的高风险信号通路,为临床诊治提出潜在新的靶标。
1 材料与方法 1.1 一般资料 1.1.1 微阵列芯片数据的获取从GEO数据库下载室管膜瘤样本和正常脑组织样本mRNA表达芯片数据集。其中GSE86574包括39个EPN样本和10个正常样本;GSE74195包括14个EPN样本和5个正常样本;GSE66354包括83个EPN样本和13个正常样本;GSE50161包括46个EPN样本和13个正常样本。
1.1.2 细胞系的获取人正常胶质细胞(HEB)和室管膜瘤细胞系(BXD-1425 EPN,DKFZ-EP1NS,R254)均购买于美国ATCC细胞库(https://www.atcc.org/)。细胞培养于Dulbecco改良的Eagle培养基(DMEM,Hyclone,USA),该培养基含有10%胎牛血清。37 ℃,5%CO2和95%空气条件下培养细胞。
1.1.3 临床患者数据的收集从中国胶质瘤基因组图谱(CGGA)(http://www.cgga.org.cn)数据库下载临床患者样本数据,其中共包括325例患者的临床数据(男性203例,女性122例,年龄8~81岁)。根据核心驱动基因TOP2A、CDK1、PCNA和ACTA2的表达,将患者分为相对高表达组和相对低表达组进行生存分析,无进展生存率(progression free survival, PFS)和整体生存率(overall survival, OS)作为患者的预后指标。
1.2 实验方法 1.2.1 差异表达基因的鉴别使用GeneSpring软件(美国Aglient 11.5版)对样本基因表达谱数据进行分析处理,针对4个样本数据集,分别进行疾病组和对照组的对比研究,采用层次聚类分析和主成分分析(PCA)对GeneSpring探针进行质量控制,强度值低于20%的探针选择“Filter Proeset by Expression”进行过滤,采用t检验进行统计学处理。最后使用了在线网络工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)对4组差异表达的基因进行维恩图的绘制。
1.2.2 差异表达基因的GO、KEGG和GSEA分析使用David(http://david.abcc.ncifcrf.gov/)基因功能注释在线数据库工具,对鉴别出的差异表达基因进行分析,探究差异表达基因的主要功能及富集的信号通路,探究差异表达基因背后的生物学意义。GO分析研究差异表达基因富集的具体生物学过程、分子功能和细胞成分。KEGG分析探究差异表达基因引起哪些具体信号通路的改变。GSEA进一步分析探究差异表达基因主要作用的细胞信号通路。
1.2.3 PPI蛋白互作网络构建利用基因相互作用的在线检索工具String(http://string.embl.de/)以及CYTOCAPE软件,构建差异表达基因的PPI网络。随后进行分子复合体检测(MCODE),筛选出MCODE评分>62,节点数>9的3个蛋白互作模块,并对共表达关系紧密的模块进行GO、KEGG分析,以了解整体模块基因主要参与的细胞功能及富集的信号通路。
1.2.4 实时定量逆转录PCR基于CFX 96实时系统(Bio-Rad),使用FastStart通用SYBR Green Master(ROX)进行qRT-PCR,根据仪器使用说明书,利用GAPDH作为基因表达水平的参照,以探究核心驱动基因在室管膜瘤以及正常脑细胞中的表达情况。利用Ct法,进行RT-qPCR数据的分析。
1.3 统计学分析实验获得的数据均输入SPSS 22.0软件进行统计学分析,采用独立样本t检验。检验水准:α=0.05。
2 结果 2.1 差异表达基因的鉴别GSE86574数据集中检出12 392个差异表达基因,其中5 983个表达水平下调,6 409个上调。GSE74195中共有3 350个差异表达基因,其中1 909个下调,1 441个上调。GSE66354中共有5 581个差异表达基因,2 918个下调,2 663个上调。GSE50161中共有5 721个差异表达基因,3 021个下调,2 700个上调。
2.2 差异表达基因的GO、KEGG以及GSEA分析分别将4个微阵列芯片数据共同上调或者下调的基因导入DAVID工具,进行GO和KEGG计算分析。在室管膜瘤肿瘤中,有多种细胞功能和信号通路受到影响。GO分析结果表明,上调的基因富集于神经元细胞化学突触传递、神经递质分泌、胞吐调节和视网膜及神经系统发育等方面;参与离子通道信号传递、GABA-A受体活性、钙离子结合影响钙调素等分子功能;在细胞连接、突触后膜中发挥促进作用。下调的基因在细胞外基质、纤毛形态发生和成纤维细胞分化、纤毛发生以及胶原纤维组织的分化中具有重要作用;发挥调节MHCⅡ类受体活性,调节细胞表面整合素的作用;主要参与构成细胞外小体、细胞外基质、黏着斑。使肿瘤细胞间彼此依赖性减弱,易于肿瘤细胞迁移。此外,上调的基因主要促进逆行神经内信号传导、谷氨酸能突触、GABA能突触、吗啡成瘾等信号通路的功能;而下调的差异表达基因在ECM受体相互作用、病毒性心肌炎、移植物抗宿主病、同种异体排斥反应和Hippo信号通路起到负性调节作用。具体分析结果见图 1A、表 1。同时,GSEA分析结果表明,尤其在Hedgehog信号、Notch信号和错配修复通路中,差异表达基因起到明显作用,对室管膜瘤的发生发展产生重要的影响(图 1B)。
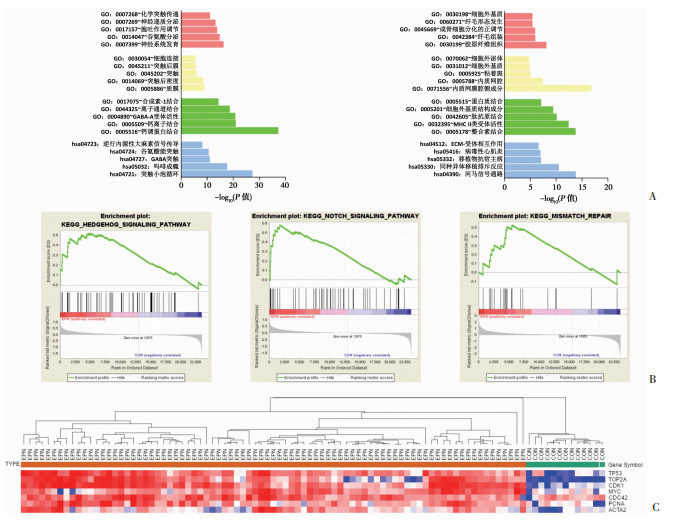
|
| A:差异表达基因富集的信号通路左:上调基因富集的信号通路;右:下调基因富集的信号通路;B:高风险通路的GSEA分析结果左:Hedgehog信号通路;中:Notch信号通路;右:错配修复通路;C:核心驱动基因表达情况热图 图 1 核心驱动基因表达情况及高风险通路的筛选 |
| 表达 | 类型 | 类目 | 基因数 | 百分比(%) | P值 |
| 表达上调 | GOTERM_BP_DIRECT | GO:0007268~化学突触传递 | 53 | 8.03 | 3.77E-28 |
| GOTERM_BP_DIRECT | GO:0007269~神经递质分泌 | 22 | 3.33 | 2.06E-18 | |
| GOTERM_BP_DIRECT | GO:0017157~胞吐作用调节 | 13 | 1.97 | 8.79E-12 | |
| GOTERM_BP_DIRECT | GO:0014047~谷氨酸分泌 | 13 | 1.97 | 2.61E-11 | |
| GOTERM_BP_DIRECT | GO:0007399~神经系统发育 | 32 | 4.85 | 6.07E-09 | |
| GOTERM_CC_DIRECT | GO:0030054~细胞连接 | 83 | 12.58 | 6.09E-38 | |
| GOTERM_CC_DIRECT | GO:0045211~突触后膜 | 43 | 6.52 | 1.14E-21 | |
| GOTERM_CC_DIRECT | GO:0045202~突触 | 40 | 6.06 | 1.66E-21 | |
| GOTERM_CC_DIRECT | GO:0014069~突触后密度 | 38 | 5.76 | 2.17E-19 | |
| GOTERM_CC_DIRECT | GO:0005886~质膜 | 216 | 32.73 | 4.96E-15 | |
| GOTERM_MF_DIRECT | GO:0017075~合成素-1结合 | 10 | 1.52 | 1.19E-09 | |
| GOTERM_MF_DIRECT | GO:0044325~离子通道结合 | 20 | 3.03 | 2.79E-09 | |
| GOTERM_MF_DIRECT | GO:0004890~GABA-A受体活性 | 8 | 1.21 | 1.16E-06 | |
| GOTERM_MF_DIRECT | GO:0005509~钙离子结合 | 48 | 7.27 | 2.31E-06 | |
| GOTERM_MF_DIRECT | GO:0005516~钙调蛋白结合 | 21 | 3.18 | 2.71E-06 | |
| KEGG_PATHWAY | hsa04723:逆行内源性大麻素信号传导 | 27 | 4.09 | 5.15E-17 | |
| KEGG_PATHWAY | hsa04724:谷氨酸能突触 | 27 | 4.09 | 1.37E-15 | |
| KEGG_PATHWAY | hsa04727:GABA突触 | 23 | 3.48 | 1.42E-14 | |
| KEGG_PATHWAY | hsa05032:吗啡成瘾 | 23 | 3.48 | 6.62E-14 | |
| KEGG_PATHWAY | hsa04721:突触小泡循环 | 18 | 2.73 | 7.63E-12 | |
| 表达下调 | GOTERM_BP_DIRECT | GO:0030198~细胞外基质 | 37 | 4.75 | 1.53E-14 |
| GOTERM_BP_DIRECT | GO:0060271~纤毛形态发生 | 27 | 3.47 | 2.81E-11 | |
| GOTERM_BP_DIRECT | GO:0045669~成骨细胞分化的正调节 | 15 | 1.93 | 7.86E-08 | |
| GOTERM_BP_DIRECT | GO:0042384~纤毛组装 | 21 | 2.70 | 1.09E-07 | |
| GOTERM_BP_DIRECT | GO:0030199~胶原纤维组织 | 12 | 1.54 | 2.45E-07 | |
| GOTERM_CC_DIRECT | GO:0070062~细胞外泌体 | 188 | 24.13 | 1.77E-14 | |
| GOTERM_CC_DIRECT | GO:0031012~细胞外基质 | 43 | 5.52 | 3.82E-13 | |
| GOTERM_CC_DIRECT | GO:0005925~黏着斑 | 46 | 5.91 | 8.36E-11 | |
| GOTERM_CC_DIRECT | GO:0005788~内质网腔 | 30 | 3.85 | 3.91E-10 | |
| GOTERM_CC_DIRECT | GO:0071556~内质网膜腔侧成分 | 11 | 1.41 | 7.84E-08 | |
| GOTERM_MF_DIRECT | GO:0005515~蛋白质结合 | 452 | 58.02 | 1.41E-17 | |
| GOTERM_MF_DIRECT | GO:0005201~细胞外基质结构成分 | 16 | 2.05 | 3.76E-08 | |
| GOTERM_MF_DIRECT | GO:0042605~肽抗原结合 | 9 | 1.16 | 8.58E-06 | |
| GOTERM_MF_DIRECT | GO:0032395~MHCⅡ类受体活性 | 7 | 0.90 | 1.35E-05 | |
| GOTERM_MF_DIRECT | GO:0005178~整合素结合 | 16 | 2.05 | 1.51E-05 | |
| KEGG_PATHWAY | hsa04512:ECM-受体相互作用 | 20 | 2.57 | 7.17E-09 | |
| KEGG_PATHWAY | hsa05416:病毒性心肌炎 | 14 | 1.80 | 1.06E-06 | |
| KEGG_PATHWAY | hsa05332:移植物抗宿主病 | 11 | 1.41 | 1.15E-06 | |
| KEGG_PATHWAY | hsa05330:同种异体移植排斥反应 | 11 | 1.41 | 3.68E-06 | |
| KEGG_PATHWAY | hsa04390:河马信号通路 | 22 | 2.82 | 3.74E-06 |
2.3 核心驱动基因的筛选及基因共表达模块的构建
通过构建蛋白互作网络,可以筛选处于核心地位的致病高风险因子以及联系紧密的基因共表达模块。共选出10个核心驱动基因,TP53、TOP2A、CDK1、PCNA和ACTA2等在室管膜瘤发生、发展中处于核心地位(图 1C、表 2)。此外,使用MCODE筛选出最重要的3个基因共表达模块,模块内基因相互联系紧密,共同发挥促瘤作用(图 2A)。分别对各个模块进行具体功能分析,结果表明,这些基因主要在细胞周期、胞吞作用、损伤修复、谷氨酸突触、GABA能突触、蛋白剪接体、粘着斑和胶原纤维的形成等方面产生重要影响,赋予细胞成瘤特性,从而导致室管膜瘤的发生(表 3)。
| 基因名 | 权重 | 中介中心性 |
| TP53 | 158 | 0.111 474 |
| TOP2A | 123 | 0.055 364 |
| CDK1 | 103 | 0.037 421 |
| PCNA | 99 | 0.042 523 |
| ACTA2 | 98 | 0.049 124 |
| MYC | 88 | 0.024 017 |
| UMPS | 82 | 0.030 001 |
| CDC42 | 81 | 0.041 230 |
| NOTCH1 | 75 | 0.025 682 |
| CDK2 | 74 | 0.013 782 |

|
| A:蛋白质-蛋白质相互作用网络模块;B:RT-qPCR验证核心驱动基因TOP2A、CDK1、PCNA以及ACTA2在不同细胞系中的表达a: P < 0.05,与HEB比较 图 2 蛋白互作核心驱动基因筛选以及核心基因表达量的验证 |
| 模块 | 类目 | 基因数 | P值 | FDR | 基因 |
| 1 | cfa04110:细胞周期 | 6 | 3.72E-05 | 0.034633614 | CDK1, MAD2L1, PCNA, BUB1B, TTK, CDC20 |
| cfa04144:胞吞作用 | 7 | 1.07E-04 | 0.099771701 | SH3GL3, DNM3, ARRB1, DNAJC6, DNM1, SH3GL2, AMPH | |
| GO:0019985~跨膜损伤修复 | 3 | 3.65E-04 | 0.486721579 | DTL, KIAA0101, PCNA | |
| 2 | cfa04724:谷氨酸能突触 | 11 | 1.43E-10 | 1.53E-07 | GRM4, ADCY1, GRM3, PLCB4, GNG13, GNB5, GNG11, GNG3, GNG12, GNG5, GRM1 |
| cfa04727: GABA突触 | 9 | 9.40E-09 | 1.01E-05 | ADCY1, GABBR1, GNG13, GNB5, GNG11, GABBR2, GNG3, GNG12, GNG5 | |
| cfa03040:剪接体 | 10 | 1.15E-08 | 1.23E-05 | EIF4A3, SNRPD3, LSM7, ALYREF, LSM5, HNRNPC, SNRPF, SNRPE, PUF60, SNRPG | |
| 3 | cfa04974:蛋白质消化和吸收 | 11 | 2.54E-12 | 2.86E-09 | COL4A2, COL9A2, COL4A1, COL3A1, COL6A2, COL1A2, COL6A1, COL1A1, COL5A2, COL5A1, COL4A5 |
| cfa04510:黏着斑 | 14 | 2.86E-12 | 3.22E-09 | CDC42, CCND1, COL4A2, COL4A1, PIK3CB, ERBB2, COL3A1, COL1A2, COL6A2, COL6A1, COL1A1, COL5A2, COL5A1, COL4A5 | |
| GO:0030199~胶原纤维组织 | 8 | 9.21E-12 | 1.30E-08 | P4HA1, PLOD3, COL3A1, COL1A2, COL1A1, COL5A2, SERPINH1, COL5A1 |
2.4 RT-qPCR对核心驱动基因表达量的验证
采用RT-qPCR方法,分别测定人正常胶质细胞(HEB)和室管膜瘤细胞系(BXD-1425 EPN、DKFZ-EP1NS、R254)中TOP2A、CDK1、PCNA和ACTA2基因的表达量。结果表明,TOP2A、CDK1、PCNA和ACTA2基因在正常胶质细胞中的表达均显著低于室管膜瘤细胞株(P < 0.05);在室管膜瘤细胞系中,TOP2A、CDK1、PCNA和ACTA2基因的表达水平略有不同(图 2B)。
2.5 临床样本生存曲线分析为了明确4个核心基因的表达水平与室管膜瘤患者预后的关系,生存曲线分析结果显示,TOP2A患者的生存率更高,TOP2A、CDK1、PCNA、ACTA2相对低表达患者预后良好,生存率高,生存期更长(P < 0.05)。生存曲线见图 3。
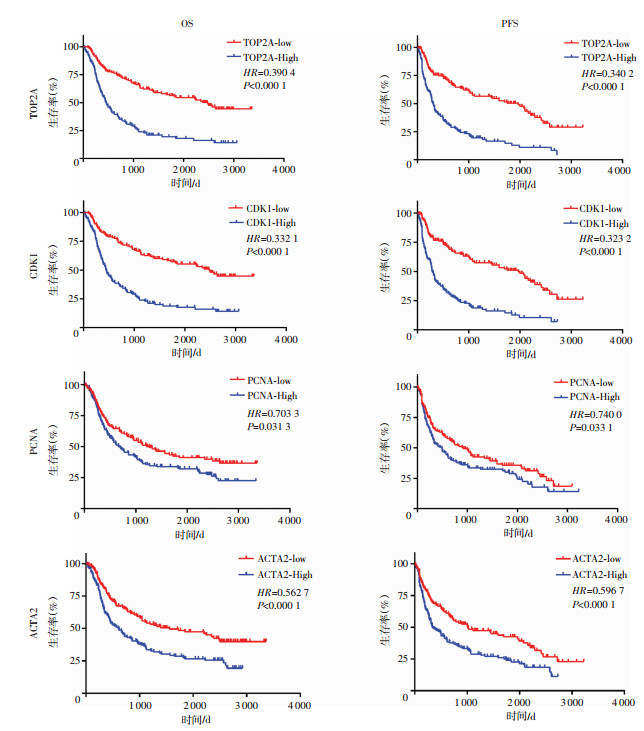
|
| 图 3 核心驱动基因在室管膜瘤患者预后情况的生存曲线分析 |
3 讨论
室管膜瘤占胶质瘤的6.8%,儿童患病率高于成人[6-7]。常以手术方法切除,术后进行放射化疗辅助,然而术后肿瘤复发率高,疗效十分有限。因此,本研究针对室管膜瘤患者的临床大样本数据集,采用标准化实验方法,针对室管膜瘤患者基因芯片数据集的mRNA表达情况进行差异表达基因的筛选,并构建蛋白表达互作网络,加之以基因权重分析,筛选出室管膜瘤形成的致病核心驱动基因。同时分析差异表达基因富集的高风险信号通路,能够为临床对室管膜瘤的预防、诊断、治疗及预后提供靶标,具有重要意义。
肿瘤不是单基因突变的结果,而是由多基因突变累积所造成。TP53是细胞抑癌基因,编码一种转录因子,控制着细胞周期的启动。其突变早期促进室管膜瘤的发生,晚期促进室管膜瘤的恶性转化[8-10]。TOP2A编码DNA拓扑异构酶,在DNA解链过程中发挥作用,通过形成瞬时双链DNA断裂来改变DNA拓扑结构,打开DNA超螺旋状态。研究表明,TOP2A在原发肿瘤中的过度表达赋予肿瘤侵袭性,同时,TOP2A与组蛋白甲基转移酶EZH2表达存在显著的正相关。EZH2在中枢神经系统中起着重要的作用,其调控异常进一步促进室管膜瘤的发生发展[11-12]。CDK是细胞周期依赖性激酶,细胞周期进程的重要调节因子。CDK1,又称CDC2,与cyclin B共同构成细胞周期检查点(MPF),促进真核细胞周期G1/S和G2/M期的转变。活性受本身磷酸化/去磷酸化化学修饰调控,其异常表达,对于室管膜瘤的形成具有重要影响[13-14]。ACTA2编码的肌动蛋白,维持骨骼肌结构和功能的完整性,维持细胞的机械张力及细胞变形运动,肌动蛋白的异常表达导致质膜流动性的改变和细胞骨架紊乱。ACTA2突变可导致主动脉瘤,影响肺腺癌细胞侵袭性,在室管膜瘤组织中ACTA2的表达量极高,具有关键作用地位[15]。PCNA编码DNA聚合酶δ的辅助因子,在DNA复制过程中提高先导链合成的效率,同时有助于提高SY的加工率,参与DNA修复。研究发现,PCNA编码的蛋白通过与染色质结合发挥作用,其参与调节的细胞过程,是癌细胞生长和繁殖的基础[16-17]。因此,通过药物靶向抑制PCNA从而抑制肿瘤生长,可能是治疗室管膜瘤新的有效方法[18]。
对室管膜瘤差异表达基因的功能注释进信号通路分析发现,室管膜瘤在染色体活化、微管组装以及细胞周期调控进而影响细胞有丝分裂等方面发生重要改变,促进室管膜瘤细胞过度增殖。同时室管膜瘤细胞表面蛋白质含量下降,减弱本身抗原性,逃避免疫系统的杀伤。室管膜瘤细胞间神经递质传递功能改变异常。这很可能是室管膜瘤并发抽筋或癫痫的病因。据报道内源性大麻素信号转导系统和吗啡成瘾与多种癌症密切相关[19],在脑内,内源性大麻素和CB1大麻素轴突终末受体联合共同调节离子通道活性及神经递质释放[20]。与正常人相比,吗啡滥用者大麻素受体数量及IL-4、IgG、IgM均上调,受体的表达和免疫功能的紊乱促进室管膜瘤的发生[21]。Hedgehog信号、Notch信号和错配修复系统与室管膜瘤密切相关。Hedgehog信号通路在胚胎发生、组织稳态和肿瘤发生过程中,调节细胞的生长状态和细胞分化,具有重要影响。Notch信号失调导致细胞恶性转化,促进包括室管膜瘤在内的多种肿瘤的发生发展[22]。综上所述,Hedgehog信号、Notch信号转导和错配修复信号异常是室管膜瘤发生的重要分子基础,可作为临床诊断性生物标志物、治疗靶点和预后指标。
本研究利用生物信息学方法,结合基因表达微阵列芯片技术,基于临床大数据样本,阐明了室管膜瘤的核心致病驱动基因是TOP2A、CDK1、PCNA和ACTA2,明确了驱动基因与患者预后的关系,并通过核心基因以及信号通路的研究进一步阐述了室管膜瘤发生发展的作用机制,为临床分子诊断以及肿瘤基因治疗提供新的靶点和思路,具有重要意义。本研究虽基于临床大样本数据进行设计,但因缺乏具体细胞、动物、分子机制上的实验验证,尚且存在着不足之处,还需要未来进一步体内外实验的证明。
| [1] | THORP N, GANDOLA L. Management of ependymoma in children, adolescents and young adults[J]. Clin Oncol (R Coll Radiol), 2019, 31(3): 162–170. DOI:10.1016/j.clon.2018.12.001 |
| [2] | TSANG D S, MURRAY L, RAMASWAMY V, et al. Craniospinal irradiation as part of Re-irradiation for children with recurrent intracranial ependymoma[J]. Neuro-oncology, 2018. [Epub ahead of print]. DOI: 10.1093/neuonc/noy191. |
| [3] | HOSOYA T, KAMBE A, NISHIMURA Y, et al. Pediatric case of Li-fraumeni syndrome complicated with supratentorial anaplastic ependymoma[J]. World Neurosurg, 2018, 120: 125–128. DOI:10.1016/j.wneu.2018.08.203 |
| [4] | RYU S M, LEE S H, KIM E S, et al. Predicting survival of patients with spinal ependymoma using machine learning algorithms with the SEER database[J]. World Neurosurg, 2018. [Epub ahead of print]. DOI: 10.1016/j.wneu.2018.12.091. |
| [5] | ZHANG C Y, PENG L, ZHANG Y Q, et al. The identification of key genes and pathways in hepatocellular carcinoma by bioinformatics analysis of high-throughput data[J]. Med Oncol, 2017, 34(6): 101. DOI:10.1007/s12032-017-0963-9 |
| [6] | DOMAZET I, PAŠALIĆ I, NEMIR J, et al. Predictors of functional outcome after spinal ependymoma resection[J]. J Neurosci Rural Pract, 2018, 9(3): 354–358. DOI:10.4103/jnrp.jnrp_56_18 |
| [7] | DONSON A M, AMANI V, WARNER E A, et al. Identification of FDA-approved oncology drugs with selective potency in high-risk childhood ependymoma[J]. Mol Cancer Ther, 2018, 17(9): 1984–1994. DOI:10.1158/1535-7163.mct-17-1185 |
| [8] | MENG X B, YANG S J, LI Y J, et al. Combination of proteasome and histone deacetylase inhibitors overcomes the impact of gain-of-function p53 mutations[J]. Dis Markers, 2018, 2018: 1–7. DOI:10.1155/2018/3810108 |
| [9] | QU H J, SU Y, YU L Z, et al. Wild-type p53 regulates OTOP2 transcription through DNA loop alteration of the promoter in colorectal cancer[J]. FEBS Open Bio, 2019, 9(1): 26–34. DOI:10.1002/2211-5463.12554 |
| [10] | GARIBALDI F, FALCONE E, TRISCIUOGLIO D, et al. Mutant p53 inhibits miRNA biogenesis by interfering with the microprocessor complex[J]. Oncogene, 2016, 35(29): 3760–3770. DOI:10.1038/onc.2016.51 |
| [11] | HANLI C, CHEN Y C. Targeting EZH2 for cancer therapy: progress and perspective[J]. Curr Protein Pept Sci, 2015, 16(6): 559–570. DOI:10.2174/1389203716666150409100233 |
| [12] | KIRK J S, SCHAARSCHUCH K, DALIMOV Z, et al. Top2a identifies and provides epigenetic rationale for novel combination therapeutic strategies for aggressive prostate cancer[J]. Oncotarget, 2015, 6(5): 3136–3146. DOI:10.18632/oncotarget.3077 |
| [13] | LIU R, FAN M, CANDAS D, et al. CDK1-mediated SIRT3 activation enhances mitochondrial function and tumor radioresistance[J]. Mol Cancer Ther, 2015, 14(9): 2090–2102. DOI:10.1158/1535-7163.MCT-15-0017 |
| [14] | BEDNAREK K, KIWERSKA K, SZAUMKESSEL M, et al. Recurrent CDK1 overexpression in laryngeal squamous cell carcinoma[J]. Tumour Biol, 2016, 37(8): 11115–11126. DOI:10.1007/s13277-016-4991-4 |
| [15] | LEE H W, PARK Y M, LEE S J, et al. Alpha-smooth muscle actin (ACTA2) is required for metastatic potential of human lung adenocarcinoma[J]. Clin Cancer Res, 2013, 19(21): 5879–5889. DOI:10.1158/1078-0432.CCR-13-1181 |
| [16] | STRZALKA W, ZIEMIENOWICZ A. Proliferating cell nuclear antigen (PCNA): A key factor in DNA replication and cell cycle regulation[J]. Ann Bot, 2011, 107(7): 1127–1140. DOI:10.1093/aob/mcq243 |
| [17] | WANG G, CAO X, LAI S, et al. PI3K stimulates DNA synthesis and cell-cycle progression via its p55PIK regulatory subunit interaction with PCNA[J]. Mol Cancer Ther, 2013, 12(10): 2100–2109. DOI:10.1158/1535-7163.mct-12-0920 |
| [18] | GU L, SMITH S, LI C, et al. A PCNA-derived cell permeable peptide selectively inhibits neuroblastoma cell growth[J]. PLoS ONE, 2014, 9(4): e94773. DOI:10.1371/journal.pone.0094773 |
| [19] | HERMANSON D J, MARNETT L J. Cannabinoids, endo-cannabinoids, and cancer[J]. Cancer Metastasis Rev, 2011, 30(3/4): 599–612. DOI:10.1007/s10555-011-9318-8 |
| [20] | PIOMELLI D. The molecular logic of endocannabinoid signalling[J]. Nat Rev Neurosci, 2003, 4(11): 873–884. DOI:10.1038/nrn1247 |
| [21] | ZHANG Q Y, ZHANG M, CAO Y. Exposure to morphine affects the expression of endocannabinoid receptors and immune functions[J]. J Neuroimmunol, 2012, 247(1/2): 52–58. DOI:10.1016/j.jneuroim.2012.04.003 |
| [22] | ABIRIA S A, WILLIAMS T V, MUNDEN A L, et al. Expression of Hedgehog ligand and signal transduction components in mutually distinct isocitrate dehydrogenase mutant glioma cells supports a role for paracrine signaling[J]. J Neurooncol, 2014, 119(2): 243–251. DOI:10.1007/s11060-014-1481-7 |



