2. 400715 重庆,西南大学生命科学院分子发育生物学实验室
2. Laboratory of Molecular Developmental Biology, School of Life Sciences, Southwest University, Chongqing 400715, China
二代测序技术发展迅速,为基因表达调控和基因功能研究带来了革命性改变[1]。而RNA-seq作为转录组研究的重要方法,加深了我们对细胞亚群和基因功能的认识,有利于发现细胞中差异性表达基因,阐明疾病的发生机制[2-3]。虽然现已发展多种单细胞RNA-Seq技术,如Drop-seq[4]、MARS-seq[5]及CEL-seq[6-7]等,但在临床诊断和个体化医疗中,常常需要对稀有组织或少量细胞样本进行RNA-seq分析。目前, 少量细胞RNA-seq技术仍面临样本RNA含量过少、构库效率较低的难题。少量细胞RNA-seq文库构建常借助Tn5转座酶,在快速片段化cDNA的同时,将测序接头连接在cDNA片段两端,有效缩短建库周期[8]。
但研究表明,Tn5转座体在少量细胞RNA-seq建库过程中,容易引入大量ME(Mosaic End)序列[9],它们在最后PCR进行文库富集时可产生非特异扩增,形成大量的无效测序Reads,降低测序文库中转录本信息含量,严重影响RNA-seq数据质量。为解决少量细胞RNA-seq中ME序列的非特异性扩增问题,我们优化Tn5转座酶表达、亲和纯化过程,进一步提高Tn5转座酶的产量,同时调整Tn5转座体的组装方式,采取超滤纯化的手段来降低组装体系中ME序列含量,并对少量RAW264.7细胞进行RNA-seq建库分析。本研究旨在建立一种高效的少量细胞RNA-seq策略,以有效减少非特异性扩增,显著提高测序质量,为进一步研究Tn5转座酶在测序中应用奠定基础。
1 材料与方法 1.1 材料和试剂 1.1.1 菌株、质粒和细胞Tn5质粒由金唯智公司合成,Codon Plus BL21(DE3)感受态细胞、TOP10感受态细胞,RAW264.7细胞均由本实验室保存,pET28a-6His-SUMO(pSUMO)载体由西南大学生命科学院分子发育生物学实验室惠赠。
1.1.2 主要试剂RNA提取试剂盒购自Zymo research公司,质粒提取试剂盒购自Megan公司, DNA第二链合成试剂盒购自NEB公司,Seamless Cloning试剂盒购自碧云天公司,文库扩增PCR酶、DNA文库纯化磁珠购自诺唯赞公司,TRIzol试剂、30 K蛋白浓缩管购自Invitrogen公司, Ni纯化珠购自Qiagen公司,所用引物由苏州金唯智公司合成。
1.2 实验方法 1.2.1 pSUMO-Tn5重组质粒构建设计两对引物扩增Tn5基因的上下游引物F1、R1;扩增载体的上下游引物F2、R2(表 1),产物同源重组后得重组质粒pSUMO-Tn5。转化后,挑单克隆过夜培养取菌液用T7引物PCR鉴定,阳性克隆送至苏州金唯智生物科技有限公司做测序鉴定。
| 引物名称 | 序列(5′-3′) |
| F1 | GGATCCGAATTCGAGCTCCGTATGATTACCAGTGCACTGC |
| R1 | GCTCGAGTGCGGCCGCAAGCTTGTCGTTATTTTAATGCCCTGCGCCATC |
| F2 | CGACAAGCTTGCGGCCGCACTCGAGC |
| R2 | ACGGAGCTCGAATTCGGATCC |
| Tn5ME-A | [phos]TCGTCGGCAGCGTCAGATGTGTATAAGAGACAG |
| Tn5ME-B | [phos]GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAG |
| Tn5MErev | [phos]CTGTCTCTTATACACATCT |
| P5 | AATGATACGGCGACCACCGAGATCTACAC[XXXXXXXX]TCGTCGGCAGCGTC |
| P7 | CAAGCAGAAGACGGCATACGAGAT[XXXXXXXX]GTCTCGTGGGCTCGG |
| [XXXXXXXX]:8 bp的测序标签;[Phos]:5′磷酸化修饰 | |
1.2.2 重组质粒表达与蛋白纯化
将重组质粒转化进入Codon Plus BL21(DE3)细胞,振荡培养过夜,将菌液转入到1L LB培养基中继续37 ℃培养至D(600)=0.6,加入0.1 mmol/L IPTG进行诱导表达,25 ℃培养8 h后收菌。菌液超声破碎,加入DNase和RNase,离心后取上清上样Ni柱,充分洗涤后分别用200 mmol/L咪唑和500 mmol/L咪唑缓冲液进行洗脱。洗脱蛋白超滤浓缩后,加入甘油于-80 ℃保存。
1.2.3 Tn5转座体的组装和纯化Tn5ME-A、Tn5ME-B分别与Tn5MErev(表 1)逐步退火形成双链,与组装缓冲液(100 mmol/L HEPES-KOH at pH 7.2, 200 mmol/L NaCl, 0.2 mmol/L EDTA, 2 mmol/L DTT, 0.2% Triton X-100, 20%甘油),Tn5转座酶蛋白,甘油充分混合,室温孵育1 h,完成组装后转移到纯化缓冲液(甘油,组装缓冲液,超纯水按照4 :4 :2体积比混匀配制)中充分混匀,并转移至30 K蛋白超滤浓缩管中,4 ℃条件下4 500×g离心。
1.2.4 RNA-seq文库构建BD Jazz流式细胞仪分选100个RAW264.7细胞至预先装有TRIzol的流式管中。用RNA提取试剂盒提取RNA后逆转录。逆转录产物用NEB第2链合成试剂盒形成双链DNA。采用超滤纯化前后的Tn5转座体(纯化组和未纯化组)和商业化Tn5试剂(对照组)酶切双链DNA,P5、P7引物PCR扩增构建测序文库,送至北京泛生子基因科技有限公司测序。
1.3 统计学分析采用GraphPad Prism 7.0软件进行单因素方差分析,比较不同Tn5转座体的无效Reads和基因数,并绘制柱形图。数据以x±s表示,多组间比较采用单因素方差分析,两组间比较采用t检验。检验水准:α=0.05。
2 结果 2.1 pSUMO-Tn5重组质粒构建扩增Tn5基因和线性载体,同源重组获得Tn5转座酶的表达质粒pSUMO-Tn5(图 1A)。挑取转化的单克隆菌落,培养后提取质粒,采用T7引物PCR验证重组质粒(图 1B)。pSUMO-Tn5质粒的PCR产物约2 000 bp,而pSUMO载体扩增产物约600 bp,鉴定正确,重组质粒送测序验证正确。
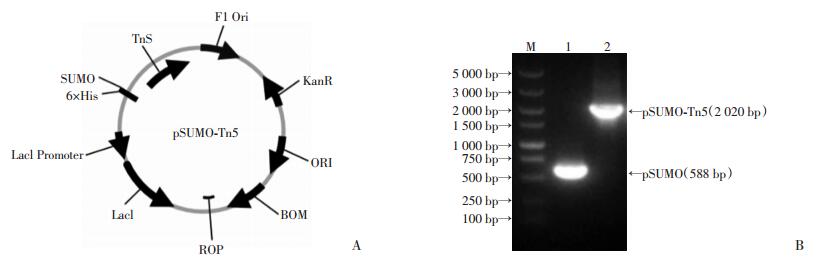
|
| A:pSUMO-Tn5重组质粒图谱;B:重组质粒的PCR鉴定 M:DNA标准;1:pSUMO载体;2:pSUMO-Tn5 图 1 质粒图谱与重组质粒的T7引物鉴定 |
2.2 Tn5转座酶的表达纯化
pSUMO-Tn5质粒转化感受态细胞,挑取单克隆菌落过夜诱导培养,采用Ni-NTA柱亲和纯化超声裂解后的细菌上清,高浓度咪唑洗脱得到SUMO-Tn5蛋白(图 2)。10% SDS-PAGE胶分析细菌裂解液、穿出液、洗涤液和洗脱液可见细菌裂解液中有明显的SUMO-Tn5蛋白条带,用不同浓度的咪唑洗脱后,获得纯度较高的SUMO-Tn5转座酶蛋白。
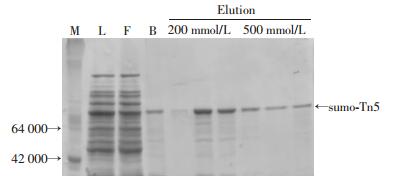
|
| M:标准;L:裂解液;F:穿出液;B:洗涤液;Elution:不同浓度咪唑洗脱产物 图 2 Tn5蛋白的表达和纯化收集 |
2.3 Tn5转座体对少量细胞转录组测序
Tn5转座酶与Tn5ME组装形成稳定的Tn5转座体,超滤纯化Tn5转座体,分选100个RAW264.7细胞构建RNA-seq文库,进行片段分析(图 3A),结果显示对照组与未纯化组中仍存在明显的非特异性扩增,而纯化组中非特异扩增大幅减少(图 4)。对纯化前后的Tn5转座体进行电泳分析,结果显示游离的Tn5ME显著减少(图 3B),从而降低Tn5ME在文库构建过程中的干扰。
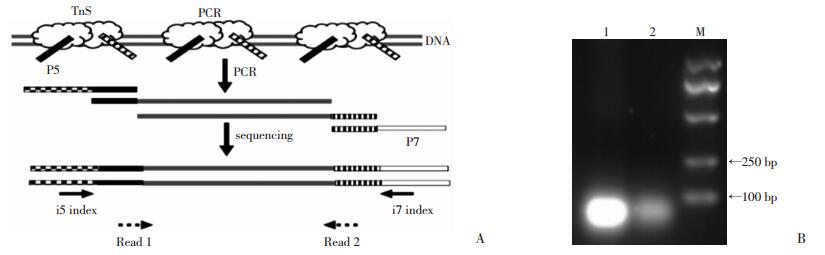
|
| A:Tn5建库流程示意图;B:纯化前后Tn5复合体的电泳分析 1:纯化前;2:纯化后;M:DNA标准 图 3 Tn5建库流程与Tn5复合体纯化前后效果比较 |
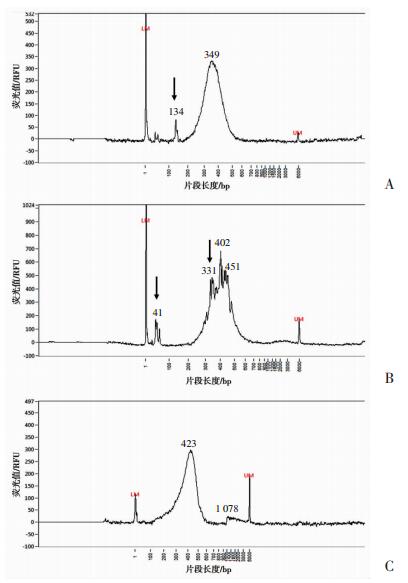
|
| A:对照组;B:未纯化组;C:纯化组;LM、UM为检测片段范围,分别为1 bp和6 000 bp;蓝色数值表示对应的片段长度;↑:示非特异性扩增 图 4 各组文库片段分布 |
2.4 测序结果
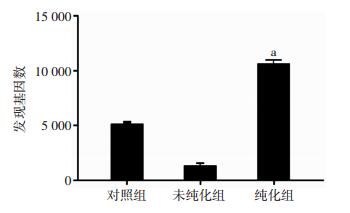
测序原始数据中过滤接头序列,未纯化组数据损失较大,剩余有效Reads数仅占35.09%,对照组中有效Reads达82.42%,而纯化组中有效Reads可以达到99.64%(表 2)。将两组数据测序标准化后,对有效Reads进行基因比对,纯化组的发现基因数为(10 806± 303),显著高于对照组(P < 0.05,图 5),说明Tn5组装后超滤纯化效果显著,可以有效提高少量细胞RNA-seq文库质量。
| 组别 | 原始Reads数 | 过滤后Reads数 | 有效Reads占比 |
| 未纯化组 | 91411849 | 32074311 | 35.09% |
| 纯化组 | 23741038 | 23655348 | 99.64% |
| 对照组 | 19437641 | 16020917 | 82.42% |
|
| a: P < 0.05, 与对照组比较 图 5 测序分析结果 |
3 讨论
Tn5转座酶属于RNaseH样蛋白超家族(RNHL)[10],在DNA转座过程中发挥重要作用[11]。转座发生时,Tn5转座酶以二聚体形式与转座序列形成转座体,促使转座子序列整合进入新的DNA位点。借助此特性,Tn5转座酶已被发展为重要的二代测序文库工具[13]。这在病毒复制和抗体V(D)J重排过程中具有重大意义[12]。而Tn5转座酶作为二代测序文库的重要工具,在少量细胞RNA-seq建库过程中容易引入过量ME序列,导致非特异性扩增,严重影响测序质量[9]。
有研究表明,融合表达Tn5-CBD(chitin-bindingdomain)产量较低、纯化难度大[14-15]。为高效获得表达的Tn5蛋白,本研究在Tn5转座酶N末端添加SUMO标签,有效提高可溶性蛋白表达量。蛋白亲和纯化前采用DNase和RNase除去体系中的DNA和RNA,充分洗涤以去除杂蛋白以及残留的DNase和RNase,从而获得较高纯度的SUMO-Tn5转座酶蛋白。
在少量细胞RNA-seq建库过程中,使用Tn5转座体易引入过量ME序列,导致非特异性扩增,严重影响测序质量[9]。我们采用蛋白浓缩管超滤纯化Tn5组装体系,选择性留下大分子量的SUMO-Tn5蛋白复合体,过滤去除小分子量的Tn5ME,从而降低体系中ME干扰。对100个RAW264.7细胞进行RNA-Seq构库,测序结果显示,对照组数据损失较大,过滤后有效Reads占82.42%,而纯化组有效Reads可达到99.64%,对有效Reads进行基因比对,纯化组基因数显著高于对照组(P < 0.05),表明超滤纯化后的Tn5转座体可显著降低少量细胞RNA-Seq的无效数据。
综上所述,本研究构建的少量细胞RNA-seq文库,以快捷的Tn5转座酶为基础,通过优化Tn5转座酶的表达和组装,降低Tn5ME在少量细胞RNA-seq文库构建中干扰,提升少量细胞测序文库质量,建立了一种高效的少量细胞RNA-Seq策略。
| [1] | BUERMANS H P, DEN DUNNEN J T. Next generation sequencing technology: Advances and applications[J]. Biochim Biophys Acta, 2014, 1842(10): 1932–1941. DOI:10.1016/j.bbadis.2014.06.015 |
| [2] | WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nat Rev Genet, 2009, 10(1): 57–63. DOI:10.1038/nrg2484 |
| [3] | OZSOLAK F, MILOS P M. RNA sequencing: advances, challenges and opportunities[J]. Nat Rev Genet, 2011, 12(2): 87–98. DOI:10.1038/nrg2934 |
| [4] | RAMSKÖLD D, LUO S J, WANG Y C, et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells[J]. Nat Biotechnol, 2012, 30(8): 777–782. DOI:10.1038/nbt.2282 |
| [5] | JAITIN D A, KENIGSBERG E, KEREN-SHAUL H, et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types[J]. Science, 2014, 343(6172): 776–779. DOI:10.1126/science.1247651 |
| [6] | PICELLI S, BJÖRKLUND Å K, FARIDANI O R, et al. Smart-seq2 for sensitive full-lengthtranscriptome profiling in single cells[J]. Nat Methods, 2013, 10(11): 1096–1098. DOI:10.1038/nmeth.2639 |
| [7] | ZIEGENHAIN C, VIETH B, PAREKH S, et al. Comparative analysis of single-cell RNA sequencing methods[J]. Mol Cell, 2017, 65(4): 631–643. DOI:10.1016/j.molcel.2017.01.023 |
| [8] | ADEY A, MORRISON H G, AS AN, et al. Rapid, low-input, low-bias construction of shotgun fragment libraries by high-density in vitro transposition[J]. Genome Biol, 2010, 11(12): R119. DOI:10.1186/gb-2010-11-12-r119 |
| [9] | CHEN C Y, XING D, TAN L Z, et al. Single-cell whole-genome analyses by linear amplification via transposon insertion (LIANTI)[J]. Science, 2017, 356(6334): 189–194. DOI:10.1126/science.aak9787 |
| [10] | MAJOREK K A, DUNIN-HORKAWICZ S, STECZKIEWICZ K, et al. The RNase H-like superfamily: new members, comparative structural analysis and evolutionary classification[J]. Nucleic Acids Res, 2014, 42(7): 4160–4179. DOI:10.1093/nar/gkt1414 |
| [11] | GERTZ J, VARLEY K E, DAVIS N S, et al. Transposase mediated construction of RNA-seq libraries[J]. Genome Res, 2012, 22(1): 134–141. DOI:10.1101/gr.127373.111 |
| [12] | REZNIKOFF W S. Tn5 as a model for understanding DNA transposition[J]. Mol Microbiol, 2003, 47(5): 1199–1206. DOI:10.1046/j.1365-2958.2003.03382.x |
| [13] | KIA A, GLOECKNER C, OSOTHPRAROP T, et al. Improved genome sequencing using an engineered transposase[J]. BMC Biotechnology, 2017, 17(1): 6. DOI:10.1186/s12896-016-0326-1 |
| [14] | HENNIG B P, VELTEN L, RACKE I, et al. Large-scale low-cost NGS library preparation using a robust Tn5 purification and tagmentation protocol[J]. G3 (Bethesda), 2018, 8(1): 79–89. DOI:10.1534/g3.117.300257 |
| [15] | PICELLI S, BJÖRKLUND A K, REINIUS B, et al. Tn5 transposase and tagmentation procedures for massively scaled sequencing projects[J]. Genome Res, 2014, 24(12): 2033–2040. DOI:10.1101/gr.177881.114 |



