2. 162650 内蒙古 呼伦贝尔,呼伦贝尔市传染病医院检验科
2. Department of Clinical Laboratory, Hulun Buir Infectious Disease Hospital, Hulun, Buir, Inner Mongolia Autonomous Region, 063210, Hebei Province, China
一线抗结核药物异烟肼、利福平、吡嗪酰胺和乙胺丁醇的联用仍是治疗肺结核最有效的方法,而四药联用引起的肝毒性是导致肺结核治疗间断及耐药发生的主要原因之一[1]。联合用药引起肝毒性作用机制的复杂性,导致损伤机制至今不明。研究证实抗结核药物性肝损伤(anti -tuberculosis drug induced liver injury,ADLI)除了与环境因素有关外,遗传因素也起重要作用[2]。DNA甲基化是一种重要的表观遗传学DNA修饰机制,其能控制多种细胞和发育过程,包括胚胎发育、X灭活、染色体稳定性和基因印记等。DNA甲基化的表观遗传学机制包括在DNA复制后将甲基从S-腺苷-L-甲硫氨酸转移到CpG二核苷酸的胞嘧啶[3]。目前,DNA异常甲基化的作用和重要性已经在多种疾病中得到证实,但在ADLI尚未探索。在本研究中,我们采用全基因组甲基化芯片技术建立ADLI全基因组DNA甲基化模式,通过生物信息学手段筛选出差异甲基化位点,对筛选出的差异位点扩大样本利用MSP法进行验证,旨在为进一步阐述ADLI发生、发展的分子机制提供理论依据。
1 资料与方法 1.1 研究对象 1.1.1 临床资料2016年1月至2017年7月间在唐山市第四医院住院且接受标准短程化疗的肺结核初治患者137例,男性65例,女性72例。纳入标准:符合《肺结核诊断和治疗指南》[4]和《肺外结核的诊断和治疗》[5]的诊断标准。符合ADLI诊断标准,采用国际共识会议标准[6]:ALT > 2倍的正常值上限(ULN)(2×ULN),或AST和TBIL联合升高,且其中之一升高 > 2×ULN,并排除其他原因引起的肝功能异常。排除标准:基线肝功能异常者、患有可引起肝功异常的其他疾病者(如病毒性肝炎、自身免疫型肝炎、酒精性肝病、脂肪肝、菌血症、充血性心力衰竭等)、服用其他可引起肝功异常的药物者。依据患者住院病历,收集患者的人口学资料、疾病相关资料、用药资料及肝损伤情况,并收集肝功能检测结果。
肺结核患者的用药剂量参照《肺结核诊断和治疗指南》[4]:由于本研究收集的是初治肺结核患者,所有患者均为成年人,依据标准化疗方案(较普遍使用,适应于大多数初治结核病患者),体质量50 kg以上成人用药剂量为:异烟肼0.3 g,利福平0.45 g,吡嗪酰胺1.5 g,乙胺丁醇0.75 g,均每天1次,强化治疗2个月,后继续用异烟肼0.3 g,利福平0.45 g,均每天1次,维持治疗4个月。但低体质量(<50 kg)、高年龄(>60岁)或弱体质患者会根据实际情况调整用药剂量。
按照病例组和对照组年龄、性别匹配的原则,共收集到符合标准的肝损伤病例65例(男性:30例,女性:35例),非肝损伤72例(男性:35例,女性:37例),从中筛选出7例非肝损伤患者(男性:4例,女性:3例)和7例肝损伤患者(男性:4例,女性:4例),其全血用于基因芯片检测,MSP法验证剩余患者(包含58例病例和65例对照)。研究得到华北理工大学医学伦理委员会批准(审批号:16011)。
1.1.2 试剂分别有:全血DNA提取试剂盒(美国Qiagen公司),全血RNA提取试剂盒(美国Qiagen公司),DNA甲基化修饰试剂盒EZ DNA Methylation-GoldTM Kit(美国ZYMO公司),逆转录试剂PrimeScriptTM RT Master Mix和SYBR® Primix Ex TaqTM购买自日本TaKaRa公司,琼脂糖(西班牙),2×Power Taq PCR Master Mix(北京艾德莱公司)。
1.2 方法 1.2.1 基因组DNA的提取与保存采集研究对象经乙二胺四乙酸二钠(EDTA)抗凝的静脉血3 mL,-80 ℃保存。采用全血DNA提取试剂盒提取DNA(Qiagen公司)。使用Nanodrop 2000(ThermoScietific)估计DNA的纯度和浓度,将质检合格的DNA于-20 ℃冰箱保存待用。
1.2.2 850k甲基化芯片全基因组扫描检测使用EZ DNA甲基化试剂盒对基因组DNA进行重亚硫酸氢盐转化。转化后经过碱变性-全基因组的扩增、段化、沉淀、重悬,在Illumina Methylation 850K芯片上经过杂交、洗涤、延伸、染色、扫描过程,获得DNA甲基化信号。应用Genomestudio软件标准化处理芯片扫描的数据结果。
1.2.3 差异甲基化基因的功能聚类分析对芯片筛选出的差异基因进行gene ontology(GO)和Kyoto Encyclopedia of Genes and Genomes(KEGG)通路分析。
1.2.4 候选基因筛选及验证依据病例组和对照组人全血DNA样本的甲基化差异值,选择组间差异值较大的高甲基化和低甲基化基因,采用MSP方法对候选基因进行验证,经850K甲基化芯片筛选发现,病例组与对照组差异值较大的高甲基化位点在DUSP22位点上,差异值较大的低甲基化位点在HLA-C位点上。DUSP22基因甲基化上、下游引物序列分别为:5′-TTT-TTCGGTATAGGTTTCGG-3′,5′-GCAATATCAACAAAT-CAACGAC-3′,产物长度为145 bp;非甲基化上、下游引物序列分别为:5′-GGAGGTGTTTTTGTTTTTATTTTATTT-3′,5′-CATTCAAAACTCAACTAACCTCTCA-3′,产物长度为153 bp。HLA-C基因甲基化上、下游引物序列分别为:5′-TGGGGAGGATTAGATTTAGGATATC-3′,5′-TAAAAAACTCTTACAACCCCTCGTA-3′,产物长度为152 bp;非甲基化上、下游引物序列分别为:5′-GGG-AGGATTAGATTTAGGATATTGA-3′,5′-TAAAAAACTC-TTACAACCCCTCATA-3′,产物长度为150 bp。MSP反应体系:2×Taq PCR Master Mix 10 μL,上、下游引物各1 μL,DNA模板3 μL,纯水5 μL。MSP反应条件均为95 ℃预变性5 min;95 ℃变性30 s,57 ℃退火30 s,72 ℃延伸30 s,40个循环;72 ℃延伸5 min,4 ℃保存。扩增产物采用经含有EB的2%琼脂糖凝胶电泳、紫外观察、凝胶成像系统扫描成像后拍照,检测扩增效果。
1.2.5 甲基化程度判定只有甲基化特异引物扩增出条带的样本记为完全甲基化(M),只有非甲基化特异引物扩增出条带的样本记为完全未甲基化(U),若甲基化特异引物和非甲基化特异引物均扩增出条带的样本记为半甲基化,也判断为甲基化。
1.2.6 DUSP22、HLA-C基因mRNA表达的检测严格按照试剂盒说明书进行全血总RNA的提取,测定RNA浓度和纯度后进行逆转录反应得到cDNA,进行PCR反应。以GAPDH为内参,引物序列为:正义链:5′-GAAGGTCGGAGTCAACGGATT-3′,反义链:5′-CCTGGAAGATGGTGATGGGAT-3′:DUSP22正义链:5′-AG-TTTAGGCTGGTGCTGC-3′,反义链:5′-CAACTACACTG-GGCTCGTC-3′。HLA-C正义链:5′-AGAACGGGAAGG- AGACGC-3′,反义链:5′ -CCAGAAGGCACCACCACA-3′。PCR条件为:95 ℃预热2 min,而后95 ℃ 15 s和60 ℃ 30 s反应40个循环。用2-△△Ct 值进行结果分析,△Ct =Ct目的基因-CtGAPDH,△△Ct = △Ct病例组-△Ct对照组。
1.3 统计学分析使用R语言中的ChAMP包分析阵列数据以获得甲基化水平。所有探针的甲基化水平用βeta (β)值表示。采用pooled t test法筛选组间差异甲基化位点,定义P < 0.01,beta. Difference > 0.10和 < -0.10分别作为组间差异基因位点高、低甲基化的评判标准。计量资料两组间比较采用t检验,计数资料采用χ2检验。检验水准:α=0.05。
2 结果 2.1 两组患者的基本资料比较芯片患者的基本信息:ADLI患者7例,男性4例,女性3例,年龄25~55(41.00±13.41)岁,作为病例组。7例非肝损伤患者,男性4例,女性3例,年龄27~57(41.43±12.33)岁,作为对照组;病例组与对照组年龄、性别差异均无统计学意义(P > 0.05)。
MSP验证组患者的基本信息:病例组,ADLI患者58例,男性26例,女性32例,17~87(48.83±18.67)岁;对照组:非肝损伤患者65例,男性31例,女性34例,年龄18~80(46.72±18.35)岁。病例组和对照组由于匹配设计的要求,两组中性别和年龄的分布是不存在差异的。文化程度、职业、吸烟、饮酒等因素在病例组和对照组中差异均无统计学意义(P > 0.05,表 1)。
| 组别 | 文化程度 | 职业 | 吸烟 | 饮酒 | |||||||||
| 小学及以下 | 初中 | 高中及以上 | 农民 | 工人 | 其他 | 是 | 否 | 是 | 否 | ||||
| 病例组 | 28 | 20 | 10 | 30 | 10 | 18 | 30 | 28 | 35 | 23 | |||
| 对照组 | 30 | 21 | 14 | 35 | 15 | 15 | 43 | 22 | 43 | 22 | |||
| χ2 | 0.363 | 1.263 | 2.645 | 0.446 | |||||||||
| P | 0.834 | 0.532 | 0.104 | 0.504 | |||||||||
2.2 ADLI与非肝损伤患者差异甲基化位点的分布特征
芯片所检测的853 307个CpG位点,根据P < 0.01和甲基化β值差值筛选出病例组和对照组差异甲基化位点共1 012个,包括254个高甲基化位点和758个低甲基化位点,低甲基化位点多于高甲基化位点。差异性高、低甲基化前10位位点分别见表 2、3。
| 位点编码 | P值 | β差值 | 染色体位置 | 基因名称 |
| cg16976594 | 5.24×10-5 | 0.241001411 | 18 | PQLC1 |
| cg26987645 | 0.000190408 | 0.253295901 | 1 | FMOD |
| cg10075506 | 0.001146974 | 0.253736725 | 2 | MYT1L |
| cg01516881 | 0.001936583 | 0.255353191 | 6 | DUSP22 |
| cg22082780 | 0.004386945 | 0.269045147 | 1 | |
| cg15648040 | 0.004580519 | 0.239220775 | 22 | PHF21B |
| cg18110333 | 0.006246024 | 0.296060381 | 6 | DUSP22 |
| cg21548813 | 0.00652814 | 0.245461196 | 6 | DUSP22 |
| cg05064044 | 0.006567219 | 0.249118328 | 6 | DUSP22 |
| cg06201514 | 0.007817602 | 0.254487217 | 2 | MYT1L |
| 位点编码 | P值 | β差值 | 染色体位置 | 基因名称 |
| cg09556042 | 2.88×10-5 | -0.417004393 | 6 | HLA-C |
| cg23970458 | 0.000235667 | -0.305437656 | 1 | |
| cg09035930 | 0.000295571 | -0.350247905 | 12 | SLC15A4 |
| cg22597981 | 0.000792329 | -0.3198943 | 2 | CPS1 |
| cg08444004 | 0.000971131 | -0.306093769 | 2 | HDAC4 |
| cg17638213 | 0.001014441 | -0.290847918 | 13 | TMCO3 |
| cg20485733 | 0.00117854 | -0.295506624 | 17 | RAP1GAP2 |
| cg17429870 | 0.001358879 | -0.306187247 | 11 | BRSK2 |
| cg14064268 | 0.001564938 | -0.314579227 | 11 | BRSK2 |
| cg17416644 | 0.009087289 | -0.297353143 | 11 | BRSK2 |
芯片所检出的高、低甲基化位点在各区域的分布情况见表 4,高、低甲基化位点主要位于基因体区(Genebody)。
| 甲基化情况 | 个数 | TSS1500 | TSS200 | 5’UTR | 1stExon | Genebody | 3’UTR | CpG岛 | 岛外CpG位点 |
| 高甲基化 | 254 | 19(7.5) | 19(7.5) | 23(9.0) | 14(5.5) | 90(35.4) | 7(2.8) | 28(11.0) | 54(21.3) |
| 低甲基化 | 758 | 100(13.2) | 27(3.6) | 73(9.6) | 16(2.1) | 269(35.5) | 23(3.0) | 29(3.8) | 221(29.2) |
2.3 差异甲基化位点的聚类分析
聚类分析显示,ADLI患者呈低甲基化状态,对照组呈高甲基化状态,见图 1。
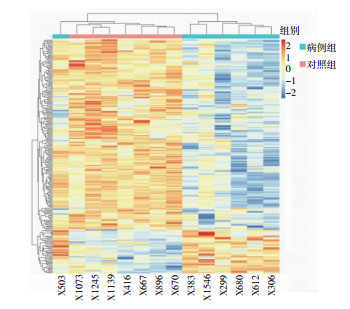
|
| X1546、X299、X306、X680、X383、X612、X503:为病例组;X1245、X416、X1139、X670、X667、X896、X1073:为对照组 图 1 病例组和对照组的聚类分析结果 |
对筛选出的差异甲基化位点分别行功能注释和代谢通路分析,富集得到840个GO功能注释和16条通路。840个GO功能注释涵盖584个基因的生物学过程、97个细胞组分和159个分子功能,见表 5。
| GO编码 | GO条目名称 | GO分类名 | P值 |
| GO:0007156 | 通过质膜粘附分子的嗜同性细胞粘附 | 生物学过程 | 1.10E-26 |
| GO:0098742 | 胞间黏附通过质膜黏附分子 | 生物学过程 | 9.20E-22 |
| GO:0007399 | 神经系统发育 | 生物学过程 | 2.90E-16 |
| GO:0005887 | 质膜的组成部分 | 细胞组分 | 6.50E-11 |
| GO:0031226 | 质膜的固有成分 | 细胞组分 | 1.80E-10 |
| GO:0044459 | 质膜部分 | 细胞组分 | 2.30E-08 |
| GO:0005509 | 钙离子结合 | 分子功能 | 1.10E-14 |
| GO:0043169 | 阳离子结合 | 分子功能 | 1.30E-05 |
| GO:0043167 | 离子结合 | 分子功能 | 2.20E-05 |
通过异常甲基化基因的生物学通路分析可知,差异甲基化位点参与多条生物学通路,如非小细胞肺癌、甲状旁腺激素的合成,分泌和作用、人巨细胞病毒感染等,见表 6。
| 通路编码 | 通路名称 | P值 |
| hsa05223 | 非小细胞肺癌 | 0.001147034 |
| hsa04928 | 甲状旁腺激素的合成,分泌和作用 | 0.001186872 |
| hsa05163 | 人巨细胞病毒感染 | 0.001321744 |
| hsa04727 | γ-氨基丁酸能突触 | 0.001428038 |
| hsa05032 | 吗啡成瘾 | 0.001773221 |
| hsa05222 | 小细胞肺癌 | 0.002037814 |
| hsa05202 | 癌症中的转录失调 | 0.002148485 |
| hsa05146 | 阿米巴病 | 0.002492003 |
| hsa04370 | VEGF信号通路 | 0.003202367 |
| hsa04080 | 神经活性配体-受体相互作用 | 0.003499154 |
2.4 MSP检测DUSP22、HLA-C甲基化状态
根据芯片结果中的基化β值差值,分别筛选差异值最显著的高甲基化位点DUSP22和低甲基化位点HLA-C,应用MSP在其他标本中做进一步验证。依据琼脂糖凝胶电泳结果中条带位置及数目判断DUSP22、HLA-C基因的甲基化情况,甲基化条带结果见图 2。
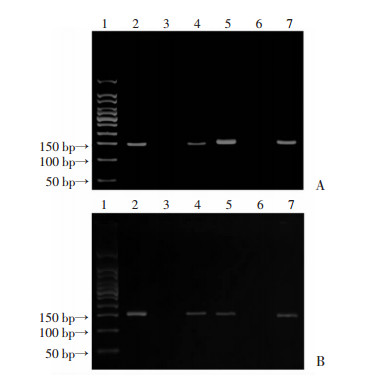
|
| A: DUSP22基因 1:DNA标准;2、3:DUSP22基因完全甲基化;4、5:DUSP22基因部分甲基化;6、7:DUSP22基因非甲基化;B:HLA-C基因 1:DNA标准; 2、3:HLA-C基因完全甲基化;4、5:HLA-C基因部分甲基化;6、7:HLA-C基因非甲基化 图 2 DUSP22和HLA-C基因MSP扩增产物电泳结果 |
DUSP22基因在病例组中甲基化率为65.5%,对照组中甲基化率为36.9%,差异具有统计学意义(P < 0.05)。对于HLA-C基因,病例组中甲基化率仅为37.9%,而非肝损伤患者的甲基化率为58.5%,差异具有统计学意义(P < 0.05,表 7)。
| 基因名称 | 组别 | 甲基化(+) | 非甲基化(-) | χ2值 | P值 |
| DUSP22 | 病例组 | 38(65.6) | 20(34.4) | 10.025 | 0.002 |
| 对照组 | 24(36.9) | 41(63.1) | |||
| HLA-C | 病例组 | 22(37.9) | 36(62.1) | 5.171 | 0.023 |
| 对照组 | 38(58.5) | 27(41.5) |
2.5 RT-PCR检测DUSP22、HLA-C mRNA表达水平
DUSP22基因mRNA的表达水平在病例组低于对照组,两组比较差异具有统计学意义(t=-15.152,P < 0.001,图 3A)。病例组HLA-C基因mRNA的表达水平高于对照组,两组比较差异具有统计学意义(t=10.681,P < 0.001,图 3B)。
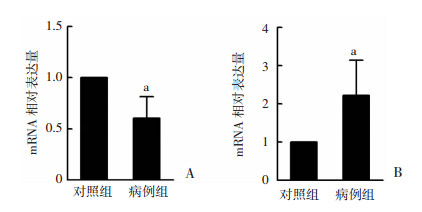
|
| A:DUSP22基因;B:HLA-C基因 a:P < 0.05,与对照组比较 图 3 DUSP22、HLA-C基因在两组中的mRNA表达水平 |
3 讨论
本研究首先利用甲基化芯片初筛抗结核药物性肝损伤与非肝损伤患者全血中差异甲基化位点。对两组人群全基因组所有甲基化位点分析、总结,我们发现,在DNA甲基化出现差异性改变的所有位点中,非损伤患者中大部分位点甲基化水平高于肝损伤患者,即肝损伤患者呈现DNA低甲基状态(图 2)。这说明DNA低甲基化在ADLI中可能发挥作用。DNA甲基化在疾病中的作用已成为近些年研究的热点,在肿瘤的研究学领域,学者提出肿瘤的发生涉及DNA甲基化模式改变,包括基因组广泛低甲基化和关键基因启动子区(如抑癌基因、DNA修复、黏附分子、转移抑制基因)的高甲基化[7]。过去研究者一直致力于这些关键基因的高甲基化研究,对于DNA低甲基化的关注尚少。有研究表明基因组广泛低甲基化也是肿瘤组织的特征之一,如重复序列、癌基因和转移相关基因的甲基化程度均可降低[8]。另外,发现了DNA低甲基化有导致染色体的杂合性缺失[9]、激活致癌基因等作用[10],这些结果提示我们,DNA低甲基化在疾病中的重要性不可忽视,有待于进一步深入研究。
本研究发现,检出差异的高甲基化和低甲基化位点分布主要位于基因体。众所周知,DNA甲基化在不同基因组区域中的作用不同,在基因间区域中,DNA甲基化的主要作用之一是抑制潜在有害遗传元件的表达[11]。在CpG岛中,CpG岛的甲基化可损害转录因子结合,募集抑制性甲基结合蛋白,并稳定沉默基因表达。针对基因体区DNA的甲基化,基因体是转录起始点(transcription start site, TSS)下游一直到转录终止位点附近区域,CpG位点数相较启动子而言更少,但它们被广泛地甲基化,中间包含有许多重复序列或者转座子。关于body区甲基化的作用目前还不是完全清楚。有证据表明基因体的DNA甲基化与分裂细胞中更高水平的基因表达相关[12],基因体内非CpG位点的甲基化与基因表达呈负相关[13]。另有研究表明,肿瘤药物地西他滨能够影响基因体的甲基化,可以将基因体甲基化作为靶标,开发新的肿瘤治疗药物[14]。这些研究提示基因体DNA甲基化的作用机制是未来研究的方向之一。
本研究通过对芯片技术筛出的2个差异倍数较大的甲基化基因DUSP22、HLA-C进一步扩大样本在人群中进行验证,发现DUSP22基因在肝损伤组甲基率高于非肝损伤组,而该基因的mRNA表达水平在肝损伤组低于非肝损伤组。最近发现的双特异性磷酸酶(dual specificity phosphatase,DUSP)是蛋白质酪氨酸磷酸酶(protein tyrosine phosphatases,PTPs)超家族的一个亚家族,因其对酪氨酸和丝/苏氨酸具有双特异性而引起重视。DUSP研究尚少,但现有的研究表明,大多数DUSP家族成员均作为丝裂原活化蛋白激酶(mitogen-activated protein kinase phosphatases,MAPK)的负向调节剂参与细胞增殖、分化、代谢、基因转录、免疫应答、肿瘤形成等过程[15]。DUSP22是DUSP家族成员之一,在c-Jun N末端激酶(JNK)信号传导中充当不同的支架蛋白。研究表明,DUSP22增加了JNK活化环中的磷酸化,此外,DUSP22选择性地与凋亡信号调节激酶1(ASK1),MAPK激酶7(MKK7)和JNK1/2相关,DUSP22通过作为ASK1-MKK7-JNK信号转导途径的支架蛋白而不依赖于其磷酸酶活性来调节细胞死亡[16]。一项关于阿尔兹海默症(alzheimer’s disease,AD)的研究中发现,在对照和患者的人海马的DNA甲基化谱研究中,鉴定出AD中DUSP22基因的启动子高甲基化的存在和其表达缺失,并进一步证明,该酶作为PKA抑制剂可能与TAU磷酸化和CREB信号传导相关[17]。本研究证明了DUSP22在肝损伤组的DNA甲基化高于非肝损伤组,且其mRNA表达水平在损伤组低于非损伤组,说明在抗结核药物性肝损伤中存在DUSP22高甲基化和其表达的缺失,但具体的生物学机制还有待下一步研究。
HLA-C是HLA类抗原的成员之一,研究表明,HLA与药物性肝损伤的发生具有强相关,但研究集中于HLA基因多态性研究。其机制可能是药物及其代谢物可与细胞内蛋白共价结合,形成复合物,HLA可将复合物呈递给T细胞,引起T细胞发生不良应答,进而导致肝细胞损伤。或者药物直接与HLA作用,无须形成共价复合物,造成T细胞发生不良应答,促使肝细胞损伤[18]。关于HLA基因甲基化与药物性肝损伤的研究尚少见报道。本研究发现人类白细胞抗原HLA-C基因在肝损伤组甲基化率低于非肝损伤组,而该基因的mRNA表达水平在肝损伤组是高于非肝损伤组。研究结果提示我们HLA-C基因表达上调可能在ADLI中发挥重要作用,而HLA-C基因DNA低甲基化改变可能是HLA-C表达上调的重要机制。
不足之处:由于本研究检测的是ADLI患者与非ADLI患者外周血白细胞中差异DNA甲基化,而不是更为直接的检测肝组织标本中差异DNA甲基化情况。另外,外周血白细胞包含了大量不同种类的细胞群体,无法解决细胞异质性难题,精准性尚存在不足。在现今单细胞测序时代,可通过对单个细胞进行甲基化测序弥补不足。
综上所述,本研究在芯片的结果中筛查出ADLI患者与非ADLI患者中差异甲基化基因DUSP22和HLA-C,证明DUSP22在ADLI中呈高甲基化状态,抑制其mRNA的表达,而HLA-C在ADLI中呈低甲基化状,其mRNA呈高表达水平,提示外周血中DUSP22、HLA-C基因甲基化与ADLI的发生相关。
| [1] | CAO J, MI Y, SHI C, et al. First-line anti-tuberculosis drugs induce hepatotoxicity: A novel mechanism based on a urinary metabolomics platform[J]. Biochem Biophys Res Commun, 2018, 497(2): 485–491. DOI:10.1016/j.bbrc.2018.02.030 |
| [2] | RUSSMANN S, JETTER A, KULLAK-UBLICK G A. Pharmacogenetics of drug-induced liver injury[J]. Hepatology, 2010, 52(2): 748–761. DOI:10.1002/hep.23720 |
| [3] | KUMAR S, CHENG X, KLIMASAUSKAS S, et al. The DNA (cytosine-5) methyltransferases[J]. Nucleic Acids Res, 1994, 22(1): 1–10. DOI:10.1093/nar/22.1.1 |
| [4] |
中华医学会结核病学分会. 肺结核诊断和治疗指南[J].
中华结核和呼吸杂志, 2001, 24(2): 70–74.
TB Society of Chinese Medical Association. Guidelines for diagnosis and treatment of tuberculosis?[J]. Chin J Tuber Respir Dis, 2001, 24(2): 70–74. DOI:10.3760/j:issn:1001-0939.2001.02.002 |
| [5] |
许绍发, 端木宏谨. 肺外结核的诊断和治疗[J].
中华结核和呼吸杂志, 1997, 20(3): 131–132.
XU S F, DUANMU H J. Diagnosis and treatment of extra-pulmonary tuberculosis[J]. Chin J Tuber Respir Dis, 1997, 20(3): 131–132. |
| [6] | BéNICHOU C. Criteria of drug-induced liver disorders. Report of an international consensus meeting[J]. J Hepatol, 1990, 11(2): 272–276. DOI:10.1016/0168-8278(90)90124-A |
| [7] | JELTSCH A, JURKOWSKA R Z. New concepts in DNA methylation[J]. Trends Biochem Sci, 2014, 39(7): 310–318. DOI:10.1016/j.tibs.2014.05.002 |
| [8] | QU J, HODGES E, MOLARO A, et al. Evolutionary expansion of DNA hypomethylation in the mammalian germline genome[J]. Genome Res, 2018, 28(2): 145–158. DOI:10.1101/gr.225896.117 |
| [9] | NAKAGAWA T, KANAI Y, USHIJIMA S, et al. DNA hypomethylation on pericentromeric satellite regions significantly correlates with loss of heterozygosity on chromosome 9 in urothelial carcinomas[J]. J Urol, 2005, 173(1): 243–246. DOI:10.1097/01.ju.0000141577.98902.49 |
| [10] | VAN TONGELEN A, LORIOT A, DE SMET C. Oncogenic roles of DNA hypomethylation through the activation of cancer-germline genes[J]. Cancer Lett, 2017, 396: 130–137. DOI:10.1016/j.canlet.2017.03.029 |
| [11] | RAUSCHER G H, KRESOVICH J K, POULIN M, et al. Exploring DNA methylation changes in promoter, intragenic, and intergenic regions as early and late events in breast cancer formation[J]. BMC Cancer, 2015, 15(1). DOI:10.1186/s12885-015-1777-9 |
| [12] | ARAN D, TOPEROFF G, ROSENBERG M, et al. Replication timing-related and gene body-specific methylation of active human genes[J]. Hum Mol Genet, 2011, 20(4): 670–680. DOI:10.1093/hmg/ddq513 |
| [13] | XIE W, BARR C L, KIM A, et al. Base-resolution analyses of sequence and parent-of-origin dependent DNA methylation in the mouse genome[J]. Cell, 2012, 148(4): 816–831. DOI:10.1016/j.cell.2011.12.035 |
| [14] | YANG X, HAN H, DE CARVALHO D D, et al. Gene body methylation can alter gene expression and is a therapeutic target in cancer[J]. Cancer Cell, 2014, 26(4): 577–590. DOI:10.1016/j.ccr.2014.07.028 |
| [15] | HUANG C Y, TAN T H. DUSPs, to MAP kinases and beyond[J]. Cell Biosci, 2012, 2(1): 24. DOI:10.1186/2045-3701-2-24 |
| [16] | JU A, CHO Y C, KIM B R, et al. Scaffold role of DUSP22 in ASK1-MKK7-JNK signaling pathway[J]. PLoS ONE, 2016, 11(10): e0164259. DOI:10.1371/journal.pone.0164259 |
| [17] | SANCHEZ-MUT J V, ASO E, HEYN H, et al. Promoter hypermethylation of the phosphatase DUSP22 mediates PKA-dependent TAU phosphorylation and CREB activation in alzheimer's disease[J]. Hippocampus, 2014, 24(4): 363–368. DOI:10.1002/hipo.22245 |
| [18] |
王书杰, 王沛, 李晓天, 等. 物性肝损伤的代谢、遗传学机制[J].
中国药理学通报, 2016, 32(7): 898–902.
WANG S J, WANG P, LI X T, et al. Drug-induced liver injury:metabolic and genetic basis[J]. Chin Pharmacol Bull, 2016, 32(7): 898–902. DOI:10.3969/j.issn.1001-1978.2016.07.003 |



