神经胶质瘤起源于神经胶质细胞,是最常见的脑部肿瘤。根据2016 WHO标准[1],胶质瘤被分为Ⅰ~Ⅳ级。级别越高患者预后越差,尤其是胶质母细胞瘤,中位生存期只有14个月[2]。被诊断为原发性胶质瘤WHOⅣ级的患者中约90%无低级别胶质瘤病史[3]。当前公认有效的治疗方案仍然以手术治疗为主,辅助以放疗、化疗[4-5]。虽然这些治疗技术也取得了新的进展[6],但胶质瘤整体预后并不乐观。胶质瘤细胞的异质性使得它们对化疗和放疗敏感度较差[7-8]。因此,研究胶质瘤发生、发展的分子机制对寻找潜在药物靶点和促进治疗进展有重大意义。
长链非编码RNA(long noncoding RNA, lncRNA)传统的定义是长度大于200碱基的RNA链,并且无蛋白质编码能力。LncRNA可通过多种机制发挥作用,如调节靶基因转录因子[9]、调控翻译和剪切、作为miRNA前体[10]、ceRNA[11]等。研究表明lncRNA在胶质瘤的发生、发展中具有重要作用。为了发现新的lncRNA,本研究分析了TCGA数据库,发现LINC00475有深入研究价值。本实验以人脑胶质瘤细胞系LN382、U87MG为研究对象,利用siRNA和RTCA技术初步探讨LINC00475对胶质瘤细胞生物学行为的影响,为寻找胶质瘤潜在的药物治疗靶点和预后标记物提供参考。
1 材料与方法 1.1 公开数据库和在线分析网站生物信息学分析使用的胶质瘤基因表达数据来自癌症和肿瘤基因图谱(cancer genome atlas,TCGA)。使用R语言TCGAbiolinks包下载胶质瘤RNA测序的FPKM表达数据,该数据使用Ensembl gene ID作为基因ID,包含174例样本,其中5例正常,13例复发胶质母细胞瘤,156例原发胶质母细胞瘤。利用HGNC数据库(www.genenames.org)提供的Complete HGNC dataset文件对TCGA数据集的Ensembl gene ID进行注释。该数据的locus_type字段描述了基因转录出的RNA是mRNA还是lncRNA。根据locus_type字段筛选出明确为lncRNA的Ensembl gene ID供进一步分析。使用limma包分析差异基因,差异标准设定为|log2FC|>1, adjust P-value < 0.001。利用Oncolnc网站(www.oncolnc.org)进行在线生存分析,String网站(string-db.org)进行基因富集分析。
1.2 样本来源10例正常组织样本和48例胶质瘤样本来自于2013年1月至2018年2月皖南医学院弋矶山医院。所采集样本均得到患者及其家属同意。10例正常样本来源于外伤患者,年龄为(54.20±13.28)岁。48例胶质瘤样本来源于肿瘤手术患者,纳入标准为:年龄40~90岁,无严重慢性病,无其他肿瘤病史,无特殊污染物接触史(放射污染、致癌化工污染)。48例胶质瘤患者中包括14例WHO Ⅱ级胶质瘤[年龄(44.07±8.26)岁]、14例WHO Ⅲ级[年龄(57.00±18.72)岁]、20例WHO Ⅳ级[年龄(53.40±14.14)岁]。样本采集后放于液氮中,TRIzol法提取的RNA储存于-80 ℃。样本根据WHO病理分级进行分组。由于大部分样本采集自额叶和颞叶,其他区域的样本量较少,根据采集区域进行分组后统计功效较差,因而未讨论采集区域与表达的关系。
1.3 细胞系人类胶质瘤细胞系(LN382、U87MG)购自中科院上海细胞库。2个细胞系均使用含10%血清的DMEM高糖培养基培养(美国HyClone公司),培养液不含青霉素和链霉素,在5%CO2的37 ℃培养箱中孵育。
1.4 qRT-PCR检测总RNA使用TRIzol reagent(美国Invitrogen公司)从组织或细胞中提取。总RNA逆转录反应选用RevertAid Fist Strand cDNA Synthesis Kit #K1622 (美国Thermo公司),取1 μg总RNA作为逆转录模板,在Icycling GT9612 PCR仪进行逆转录。逆转录后使用QuantiNovaTM SYBR ® Green PCR kit在QuantStudio3仪器进行荧光定量PCR,使用GAPDH作为内参。引物GAPDH上游:5′-GTGGTCTCCTCTGACTTCAACA-3′, 下游:5′-CCACCACCCTGTTGCTGTAG-3′,产物长度123 bp; LINC00475上游:5′-AGGGTTCACAAACGAGCCT-3′, 下游:5′-CCCAGGCTCCTAACCATGTC-3′,产物长度169 bp。逆转录和qPCR反应步骤遵循试剂盒说明书。
1.5 细胞转染由于LINC00475在TCGA和临床样本中显著上调,实验选用siRNA在胶质瘤细胞系中敲减LINC00475。LINC00475-siRNA和negative siRNA由锐博公司提供。转染试剂使用锐博公司提供的riboFectTM CP。LINC00475-siRNA序列5′-GGCAAACCTTTACAGGATTATGA-3′。按照终浓度100 nmol/L siRNA配置转染液。具体操作步骤遵循riboFectTM CP试剂盒说明书。
1.6 实时细胞分析(real time cellular analysis,RTCA)RTCA技术将微电传感器整合到迁移或侵袭板的底面,当细胞在底面生长时将改变底面的电阻。电阻变化被传感器记录并由RTCA软件自动计算成细胞指数用以反映底面生长的细胞数量。可实时、动态对细胞进行定量分析,可用于追踪细胞增殖、迁移和浸润。本实验采用xCELLigence RTCA DP(model:3×16)仪器记录以及分析数据,增殖使用E-Plate 16板,迁移侵袭使用CIM-Plate 16板。实验开始前RTCA机器整体置于5%CO2、37 ℃细胞培养箱内,待RTCA机器温度与培养箱内一致方可进行数据采集。实验为每个细胞系设2个组:NC组(转染negative siRNA)和LINC00475 silence组(转染LINC00475 siRNA)。实验用RTCA技术分别在2个细胞系上检测NC组和LINC00475 silence组在细胞增殖、迁移、侵袭功能上的变化。RTCA结果为细胞指数曲线,本研究以Mann-Kendall趋势检验P < 0.001为标准判定是否存在统计学差异。以下实验均重复3次。
1.6.1 RTCA增殖实验使用E-Plate 16板共16个孔,每个孔加入含10%血清高糖DMEM培养基50 μL取得背景读数,随后按照150 μL/孔加入转染后对数期细胞悬液,设细胞浓度5 000个/孔。接种细胞后将E-Plate 16放入RTCA机器中等待0.5 h(以待细胞沉降,CIM-Plate 16板复温到37 ℃)开始记录数据,连续观察120 h。本实验中细胞约在24 h增长加速,48 h达到对数期,100 h达到平台期。因此选择第24小时按100 nmol/L siRNA转染浓度进行转染。每个细胞系选择4个孔转染LINC00475 silence,4个孔转染NC。
1.6.2 RTCA迁移实验使用CIM-Plate 16,上室加入100 μL/孔转染后对数期细胞的无血清培养基悬液,设细胞浓度20 000/孔。下室加入含10%血清培养基后合上上下室。放入RTCA机器中等待0.5 h(以待CIM-Plate 16板复温到37 ℃)启动RTCA程序。待第24小时终止RTCA实验,导出数据并统计分析。
1.6.3 RTCA侵袭实验使用CIM-Plate 16。在种细胞前需要在每个实验孔上室内铺30 μL稀释后基质胶(BD MatrigelTM 356234,1:5稀释),其他具体操作步骤与RTCA迁移实验步骤类似。接种细胞数量改为每孔40 000细胞。
1.7 统计学分析数据以x±s表示。两组比较采用非配对Student t检验。多组比较采用单因素方差分析,组间使用Tukey HSD检验。临床样本按照LINC00475中位表达值作为阈值,将样本分为高表达组和低表达组进行生存分析。生存分析采用Kaplan-Meier法和log-rank检验。RTCA曲线的检验方法采用Mann-Kendall趋势检验:①计算差值序列:设有2条RTCA曲线A、B,Ai Bi表示时刻i曲线A或B对应的细胞指数。差值序列定义为:Dj=abs(Aj-Bj),abs表示绝对值运算;②对序列D进行Mann-Kendall趋势检验。统计分析使用R语言完成。
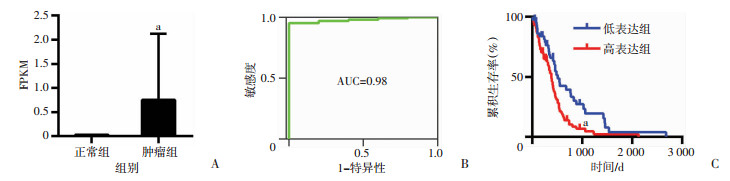
2 结果 2.1 LINC00475在胶质瘤中明显上调且与预后相关LINC00475在TCGA FPKM数据中的分布见图 1A,肿瘤组LINC00475表达较正常组显著上调(P < 0.001)。ROC曲线(图 1B)显示LINC00475的表达对胶质母细胞瘤诊断拥有较好的诊断效能,曲线下面积(area under curve, AUC)达到0.98。生存分析显示LINC00475的表达和预后有显著关联(图 1C)。高表达组预后明显差于低表达组(P < 0.01)。该数据提示LINC00475有深入研究意义。
|
| A:LINC00475在TCGA中表达差异a: P < 0.001, 与正常组比较;B: LINC00475 TCGA数据的ROC曲线;C:基于LINC00475 TCGA胶质瘤数据的生存分析a: P < 0.01, 与低表达组比较 图 1 LINC00475在TCGA胶质瘤测序数据中的分析结果 |
2.2 LINC00475在胶质瘤组织中明显上调且与分期分级显著相关
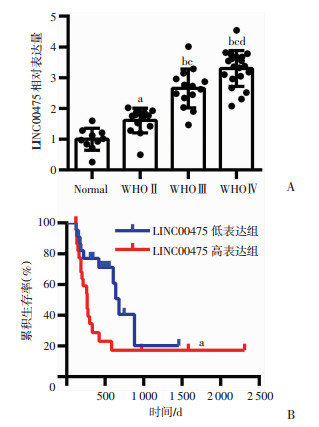
qRT-PCR检测组织样本LINC00475的表达,结果显示LINC00475表达和WHO分级呈显著相关,随着WHO分级的增加LINC00475表达显著增加(图 2A)。在组织标本中的LINC00475表达与预后显著相关(P < 0.05,图 2B),与TCGA数据相符。
|
| A:LINC00475的相对表达量与WHO分级的关系a: P < 0.05, b: P < 0.01, 与Normal比较;c: P < 0.01,与WHOⅡ比较;d: P < 0.01, 与WHOⅢ比较;B:LINC00475在临床样本中的生存分析a: P < 0.05,与低表达组比较 图 2 qRT-PCR检测LINC00475在胶质瘤组织标本中的表达 |
2.3 siRNA在LN382和U87MG细胞系中抑制LINC00475的表达
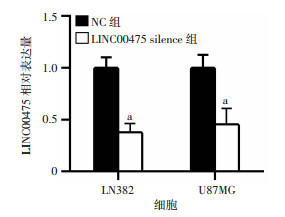
LN382细胞和U87MG细胞在6孔板内培养,转染24~48 h后用qPCR验证转染效率,结果显示应用抑制剂后LINC00475表达明显降低(图 3)。说明本实验中所使用的siRNA能有效抑制LINC00475的表达。
|
| a: P < 0.001, 与对应NC组比较 图 3 qRT-PCR检测LINC00475在转染后细胞的表达 |
2.4 LINC00475能影响胶质瘤细胞增殖、侵袭、迁移能力
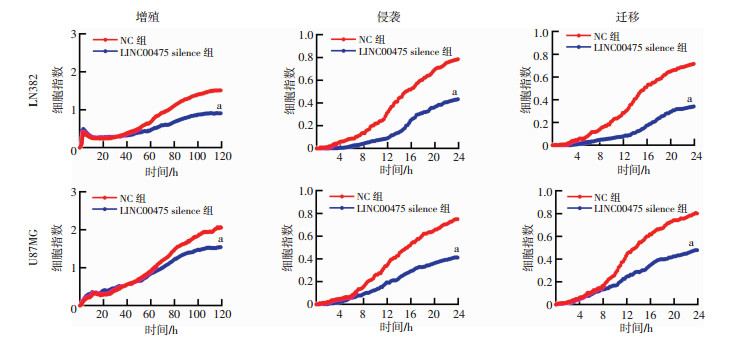
RTCA实验证实,LN382和U87MG两个细胞系siRNA组的增殖、侵袭、迁移功能被显著抑制(P < 0.001,图 4)。表明LINC00475能促进细胞增殖、侵袭和迁移。
|
| a: P < 0.001,与NC组比较 图 4 RTCA检测LINC00475对胶质瘤细胞增殖、侵袭、迁移功能的影响 |
2.5 LINC00475共表达分析
在确认LINC00475在胶质瘤中的功能后,根据TCGA数据,利用“Spearman相关”构建了共表达网络。在其中找到与LINC00475 Spearman相关系数绝对值大于0.5的基因,共计110个,对这些基因进行GO富集分析发现ARHGEF7、ADGRB1、CADM2、CD58、CLDN7、CSTA、DSCAM、FAP、FES、FLRT1、HSPB1、IL15、LPXN、NLGN3、PODXL2、PSTPIP1、RAB27A、TNFSF4显著富集在细胞黏附通路(图 5)。
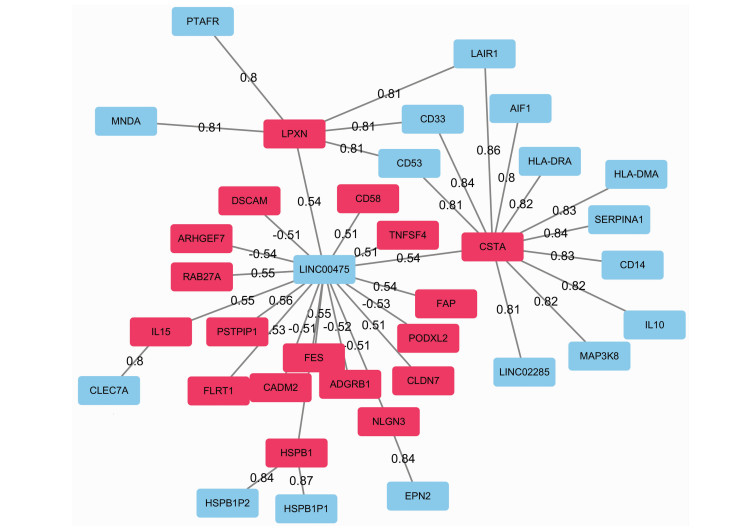
|
| 图中只显示了富集在细胞黏附通路的基因, 及其部分相邻基因;富集到细胞黏附通路的基因用红色表示,其他基因用浅蓝色表示;连线上的数字为Spearman相关系数 图 5 LINC00475的共表达网络 |
3 讨论
随着胶质瘤发病率的逐年增加,胶质瘤已威胁全球健康。现阶段以手术治疗疗效明确,不同等级胶质瘤中以WHOⅣ级酵母细胞瘤预后较差,是否复发仍无有效手段评估,故而对其发生、发展的分子机制研究显得尤为重要。随着研究的深入,人们发现lncRNA参与动脉粥样硬化[12]、肿瘤、糖尿病[13]等疾病的发生、发展,lncRNA现已成为研究热点。虽然不断有新的lncRNA的报道,但大部分lncRNA功能仍然是未知的。
为了寻找新的与胶质瘤发生、发展相关的功能lncRNA。本研究通过挖掘TCGA数据库筛选出有潜在研究价值的基因。发现LINC00475在肿瘤组织中明显上调,且生存分析中高表达组预后显著差于低表达组(P < 0.01),并在临床组织样本中重现了这一结果。在48个胶质瘤样本和10个正常样本的qPCR数据中,LINC00475的表达与WHO高低分级显著正相关,高表达组预后显著差于低表达组(P < 0.05),该结果与TCGA数据库相符合。在TCGA数据中的ROC曲线下面积达到0.98,但这里的ROC分析存在缺陷,TCGA数据中脑组织正常样本量不够丰富。在功能实验中,本研究利用siRNA技术在胶质瘤细胞系LN382、U87MG中敲减了LINC00475的表达,并检测细胞增殖、
美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)为LINC00475分配的Gene Accession为NR_027341, 根据hg38基因组注释,定位于chr9: 92141467-92159608+。为进一步揭示LINC00475的可能机制,本研究对TCGA进行共表达分析。筛选出与LINC00475存在共表达的基因共计110个,其中有18个基因在GO富集分析中显著富集到细胞黏附通路。我们的实验结果证实LINC00475对胶质瘤细胞侵袭迁移能力有影响,这和富集分析结果相符。在共表达网络(图 5)中红色标记的点是明确富集到黏附相关通路的蛋白,这些蛋白中有些起到抑制肿瘤侵袭、迁移的作用(如CADM2[14]、CLDN7[15]等),有些起到促进肿瘤迁移(如NLGN3[16])的作用。LINC00475与这些蛋白在胶质瘤中存在共表达现象,它们之间可能存在直接或者间接的相互作用。
综上所述,本研究表明LINC00475的表达与胶质瘤WHO分级呈显著正相关,并且高表达患者预示着不良的预后。提示LINC00475可能是潜在的肿瘤标记物和药物靶点。功能实验证实了LINC00475能促进胶质瘤细胞的增殖、侵袭、迁移功能,但具体分子机制尚不明确,有待进一步研究。
| [1] | WEN P Y, HUSE J T. 2016 World Health Organization Classification of central nervous system tumors[J]. Continuum (Minneap Minn), 2017, 23(6): 1531–1547. DOI:10.1212/con.0000000000000536 |
| [2] | DELGADO-L-PEZ P D, CORRALES-GARC-A E M. Survival in glioblastoma: a review on the impact of treatment modalities[J]. Clin Transl Oncol, 2016, 18(11): 1062–1071. DOI:10.1007/s12094-016-1497-x |
| [3] | OHGAKI H, KLEIHUES P. The definition of primary and secondary glioblastoma[J]. Clin Cancer Res, 2013, 19(4): 764–772. DOI:10.1158/1078-0432.CCR-12-3002 |
| [4] | HEGI M E, DISERENS A C, GODARD S, et al. Clinical trial substantiates the predictive value of O-6-methylguanine-DNA methyltransferase promoter methylation in glioblastoma patients treated with temozolomide[J]. Clin Cancer Res, 2004, 10(6): 1871–1874. DOI:10.1158/1078-0432.ccr-03-0384 |
| [5] | STUPP R, MASON W P, VAN DEN BENT M J, et al. Radiotherapy plus concomitant and adjuvant temozolomide for glioblastoma[J]. N Engl J Med, 2005, 352(10): 987–996. DOI:10.1056/NEJMoa043330 |
| [6] | MANSOURI A, MANSOURI S, HACHEM L D, et al. The role of 5-aminolevulinic acid in enhancing surgery for high-grade glioma, its current boundaries, and future perspectives: A systematic review[J]. Cancer, 2016, 122(16): 2469–2478. DOI:10.1002/cncr.30088 |
| [7] | HADJIPANAYIS C G, VAN MEIR E G. Tumor initiating cells in malignant gliomas: biology and implications for therapy[J]. J Mol Med, 2009, 87(4): 363–374. DOI:10.1007/s00109-009-0440-9 |
| [8] | GOURLAY J, MOROKOFF A P, LUWOR R B, et al. The emergent role of exosomes in glioma[J]. J Clin Neurosci, 2017, 35: 13–23. DOI:10.1016/j.jocn.2016.09.021 |
| [9] | HUNG T, WANG Y, LIN M F, et al. Extensive and coordinated transcription of noncoding RNAs within cell-cycle promoters[J]. Nat Genet, 2011, 43(7): 621–629. DOI:10.1038/ng.848 |
| [10] | LU Y, ZHAO X, LIU Q, et al. lncRNA MIR100HG-derived miR-100 and miR-125b mediate cetuximab resistance via Wnt/β-catenin signaling[J]. Nat Med, 2017, 23(11): 1331–1341. DOI:10.1038/nm.4424 |
| [11] | WANG P, LIU Y H, YAO Y L, et al. Long non-coding RNA CASC2 suppresses malignancy in human gliomas by miR-21[J]. Cell Signal, 2015, 27(2): 275–282. DOI:10.1016/j.cellsig.2014.11.011 |
| [12] | LIU Y, ZHENG L, WANG Q, et al. Emerging roles and mechanisms of long noncoding RNAs in atherosclerosis[J]. Int J Cardiol, 2017, 228: 570–582. DOI:10.1016/j.ijcard.2016.11.182 |
| [13] | ZHANG B, WANG D, JI T F, et al. Overexpression of lncRNA ANRIL up-regulates VEGF expression and promotes angiogenesis of diabetes mellitus combined with cerebral infarction by activating NF-κB signaling pathway in a rat model[J]. Oncotarget, 2017, 8(10): 17347–17359. DOI:10.18632/oncotarget.14468 |
| [14] | YANG L, ZHAO G, WANG F, et al. Hypoxia-regulated miR-146a targets cell adhesion molecule 2 to promote proliferation, migration, and invasion of clear cell renal cell carcinoma[J]. Cell Physiol Biochem, 2018, 49(3): 920–931. DOI:10.1159/000493224 |
| [15] | LU Z, DING L, HONG H, et al. Claudin-7 inhibits human lung cancer cell migration and invasion through ERK/MAPK signaling pathway[J]. Exp Cell Res, 2011, 317(13): 1935–1946. DOI:10.1016/j.yexcr.2011.05.019 |
| [16] | LIU R, QIN X P, ZHUANG Y, et al. Glioblastoma recurrence correlates with NLGN3 levels[J]. Cancer Med, 2018, 7(7): 2848–2859. DOI:10.1002/cam4.1538 |



