2. 400038 重庆,陆军军医大学(第三军医大学):全军战救训练技术与器材研发实验室;
3. 859000 西藏 阿里,解放军第957医院卫生处;
4. 844900 新疆 叶城,解放军第950医院新疆军区高山病研究所
2. Research and Development Laboratory of Military Combat Rescue Training Technology and Equipment, Army Health Service Training Base, Army Medical University (Third Military Medical University), Chongqing, 400038;
3. Health Department, No.957 Hospital of PLA, Ali, Tibet Autonomous Region, 859000;
4. Institute of Mountain Sickness of Xinjiang Military Command, No.950 Hospital of PLA, Yecheng, Xinjiang Autonomous Region, 844900, China
全基因组测序(whole genome sequencing,WGS)是指一次性确定某个体基因组全部DNA序列的过程。这个过程需要将个体全部染色体DNA进行测序,同时也包括线粒体DNA以及植物中的叶绿体DNA。WGS与生物信息分析相结合,有助于检测整个基因组水平上发生的变异,包括单核苷酸变异(single nucleotide variant,SNV)、小的缺失插入(insertion-deletion,INDEL)、染色体结构变异(structure variation,SV)、拷贝数变异(copy number variation,CNV)等[1]。WGS作为一种强大的研究工具,目前广泛应用于科学研究和临床研究,其庞大的测序数据为分析个体间差异、种群演化、临床疾病提供了较为全面的依据。由于二代高通量测序技术具有测序快、成本低、错误率低等特点,目前最为常用。本研究即采用二代高通量测序技术对中国塔吉克族人的全基因组进行测序分析。
中国塔吉克族主要分布在新疆喀什地区塔什库尔干塔吉克自治县,位于平均海拔4 000 m以上帕米尔高原的东部。由于常年居住在高原低氧高寒环境,其对高原环境有了相关的适应。在慢性高原病的调查中发现,世居帕米尔高原的塔吉克族人比世居青藏高原的藏族成年人患病率明显要高,表明两者之间的高原适应能力存在差异[2]。在塔吉克族50岁以上成年人高血压患病率调查中发现, 其患病率相对较高(39.2%),但均低于同年龄段新疆平原地区维吾尔族(49.53%)、汉族(51.93%)的高血压患病率[3-4]。这可能与常年居住在高原环境对动脉血压的影响有关[5],也表明塔吉克族人群有着较好的高原适应能力。在塔吉克族男性性激素水平调查研究中发现,世居高原的塔吉克族人的卵泡刺激素(follicle-stimulating hormone,FSH)、黄体生成素(luteotropic hormone,LH)显著高于世居平原的塔吉克族人,雌二醇(E2)、孕酮(PG)、睾酮(T)显著低于世居平原的塔吉克族人[6]。探索塔吉克族人群的高原遗传适应机制,不仅可以揭示塔吉克族人群的遗传特点,还能为这些高原慢性疾病的防治提供帮助。另外,中国塔吉克族的居住环境相对封闭,所以其种族基因相对比较保守,具有重要的遗传学研究意义。
目前,关于中国塔吉克族人群基因的研究很少,本研究对1例中国塔吉克族人进行全基因组重测序分析,以更全面地了解中国塔吉克族人的遗传背景和高原适应机制。
1 资料与方法 1.1 研究对象研究对象为中国新疆喀什地区塔什库尔干塔吉克自治县的1名健康成年男性:塔吉克族,年龄46岁,身高170 cm,体质量74 kg,血压130/80 mmHg,居住在平均海拔4 000 m以上的帕米尔高原46年。根据其在样本采集和全基因组重测序过程中编号均为153,将该例志愿者编号为T153。采取志愿者的外周静脉血后,经抗凝低温保存,从新疆喀什地区运送至重庆陆军军医大学进行遗传学研究。样本和临床信息采集均符合伦理学审查原则,并通过本校伦理委员会审查(2015年3月)。志愿者同意将本人遗传信息用于科学研究和论文发表。
1.2 DNA提取和mtDNA序列分析利用全血DNA提取试剂盒(Omega bio-tek)进行DNA提取,然后对线粒体DNA全长序列进行扩增(扩增引物[7]见表 1),扩增后的线粒体DNA序列送上海生工成都测序部进行测序(测序引物[7]见表 2)和拼接,将得到的线粒体DNA全长序列用mitotool网站(http://www.mitotool.org./)软件与修订后的剑桥标准序列(revised cambridge standard sequence,rCRS)进行比对[8],筛选出有差异的SNV位点,将这些SNV位点与线粒体DNA系统进化树进行比对,对其进行线粒体单倍群分型。最后运用MEGA4软件,将T153与rCRS、汉族(Han)以及藏族(Tibetan)的线粒体DNA全长序列进行比对,构建系统进化树。
| 序号 | 引物序列(5′→3′) | 扩增位置 | 片段长度(bp) |
| 1 | 上游:AACCAAACCCCAAAGACACC | 569~2 941 | 2 372 |
| 下游:GACTCTAGAATAGGATTGCGC | |||
| 2 | 上游:GTCCTAAACTACCAAACCTGC | 2 797~5 193 | 2 396 |
| 下游:GTGTTAGTCATGTTAGCTTG | |||
| 3 | 上游:AGCAGTTCTACCGTACAACC | 5 061~7 497 | 2 436 |
| 下游:TTTGAAAAAGTCATGGAGGCC | |||
| 4 | 上游:GATTTGAGAAGCCTTCGCTTC | 7 336~9 819 | 2 483 |
| 下游:GCCAATAATGACGTGAAGTCC | |||
| 5 | 上游:TCCCACTCCTAAACACATCC | 9 611~12 111 | 2 500 |
| 下游:AAACCCGGTAATGATGTCGG | |||
| 6 | 上游:GCCCACGGGCTTACATC | 11 727~14 159 | 2 432 |
| 下游:GATTGTTAGCGGTGTGGTCG | |||
| 7 | 上游:AATCTCCACCTCCATCATCACC | 14 046~15 774 | 1 728 |
| 下游:ACTGGTTGTCCTCCGATTCAGG | |||
| 8 | 上游:TTCGCCTACACAATTCTCCG | 15 591~626 | 1 604 |
| 下游:TTTATGGGGTGATGTGAGCC |
| 序号 | 5′端位置 | 引物序列(5′→3′) |
| 1 | 569 | 上游:AACCAAACCCCAAAGACACC |
| 2 | 626 | 上游:TTTATGGGGTGATGTGAGCC |
| 3 | 1 305 | 上游:GTAAGCGCAAGTACCCACG |
| 4 | 2 070 | 上游:AATTTGCCCACAGAACCCTC |
| 5 | 2 797 | 上游:GTCCTAAACTACCAAACCTGC |
| 6 | 3 568 | 上游:CGCTCTTCTACTATGAACCC |
| 7 | 4 322 | 上游:ATAATAGGAGCTTAAACCCCC |
| 8 | 5 061 | 上游:AGCAGTTCTACCGTACAACC |
| 9 | 5 828 | 上游:GAAAATCACCTCGGAGCTGG |
| 10 | 6 563 | 上游:ACCTCAACACCACCTTCTTC |
| 11 | 7 336 | 上游:GATTTGAGAAGCCTTCGCTTC |
| 12 | 8 080 | 上游:TCTTGCACTCATGAGCTGTC |
| 13 | 8 815 | 上游:CTCATTTACACCAACCACCC |
| 14 | 9 611 | 上游:TCCCACTCCTAAACACATCC |
| 15 | 10 380 | 上游:TCTGGCCTATGAGTGACTAC |
| 16 | 11 107 | 下游:TTCACAGCCACAGAACTAATC |
| 17 | 11 381 | 下游:AAGTGGAGTCCGTAAAGAGG |
| 18 | 12 862 | 下游:AAACCGATATCGCCGATACG |
| 19 | 13 627 | 上游:AAGCGAGGTTGACCTGTTAG |
| 20 | 14 046 | 下游:AATCTCCACCTCCATCATCACC |
| 21 | 15 774 | 上游:ACTGGTTGTCCTCCGATTCAGG |
| 22 | 15 591 | 下游:TTCGCCTACACAATTCTCCG |
1.3 建库测序和质控分析
将提取的全血核DNA送至北京博奥晶典生物技术有限公司进行全基因组测序和数据分析。样品检测合格之后进行基因组DNA片段化,选择质量合格的核DNA作为测序建库起始样品,根据不同的测序类型及测序长度,选择合适的片段化程序将核DNA片段化到目标长度范围。片段化后用Agilent 2100 Bioanalyzer进行质控,建库之后进行末端修复和3′端加A制备文库。PCR扩增后通过QUBIT对文库进行定量,之后利用Agilent 2100 Bioanalyzer检测文库插入片段大小情况。最后库检合格的样品进行Hiseq 2500 PE150测序。测序原始数据中部分reads含有测序接头,部分reads测序质量较低。这些reads将会影响到后续的数据分析,所以首先使用软件cutadapt-1.7.1[9]对原始数据进行过滤得到Clean Data。有效测序数据通过bwa-0.7.12[10]比对到参考基因组上得到SAM格式的最初的比对结果。SAM文件用Picard(http://broadinstitute.github.io/picard/)对比对结果进行排序、标记重复reads(Mark duplicate reads)并转化成为BAM文件。最后利用重复标记后的比对结果进行覆盖度、深度等的统计。
1.4 生物信息学分析将测序得到的原始reads(双端序列)进行质量评估和过滤后,得到高质量的Clean Data, 将高质量的reads比对到参考序列上,基于比对结果检测变异位点SNV和Small InDel,利用Annovar进行数据库注释。利用比对信息检测结构变异(SV)及拷贝数变化(CNV),并进行注释。将常见高原适应相关基因的SNV[11]以及与中国塔吉克族民族特点相关的CNV在注释结果中进行检索和统计分析,根据检索结果分析中国塔吉克族人的遗传特征和高原适应的相关性情况。最后,将全基因组重测序数据中所有变异利用Circos软件进行全局统览[12]。
2 结果 2.1 中国塔吉克族T153 mtDNA全长序列分析通过mitotool网站,在Phylotree 17 rcRS(线粒体DNA进化树2016版中,以修订后的剑桥标准序列:NC_012920)为标准,将T153的mtDNA序列全长与其进行比对分析,检索得到相应的SNV分布情况和线粒体单倍群分型(表 3)。
| 位点 | 碱基 | 替换 |
| 73 | A | G |
| 204 | T | C |
| 207 | G | A |
| 263 | A | G |
| 497 | C | T |
| 750 | A | G |
| 1 050 | C | G |
| 1 189 | T | C |
| 1 438 | A | G |
| 1 811 | A | G |
| 2 220 | A | G |
| 2 706 | A | G |
| 3 480 | A | G |
| 4 769 | A | G |
| 5 196 | T | C |
| 5 460 | G | A |
| 7 028 | C | T |
| 7 229 | C | T |
| 8 860 | A | G |
| 9 055 | G | A |
| 9 698 | T | C |
| 10 398 | A | G |
| 10 550 | A | G |
| 11 299 | T | C |
| 11 467 | A | G |
| 11 719 | G | A |
| 12 508 | A | G |
| 12 566 | A | G |
| 12 738 | T | G |
| 12 771 | G | A |
| 14 167 | C | T |
| 14 182 | T | C |
| 14 766 | C | T |
| 14 798 | T | C |
| 15 326 | A | G |
| 15 391 | C | G |
| 15 799 | A | G |
| 16 172 | T | C |
| 16 224 | T | C |
| 16 301 | C | T |
| 16 311 | T | C |
| 16 519 | T | C |
比对得到其单倍群属于K1a12,其基因数据库编号为JQ703323。单倍群H2a2a1代表的是rCRS,即修订后的标准的剑桥序列。运用MEGA4软件将T153与rCRS、汉族(Han)以及藏族(Tibetan)的线粒体DNA全长序列进行比对,然后构建系统进化树(图 1)。
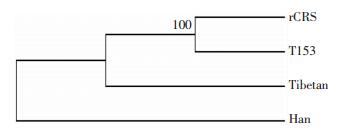
|
| 图 1 mtDNA系统进化树分布 |
2.2 T153全基因组测序比对和变异分析
对中国塔吉克族T153的全基因组测序进行汇总分析,发现原始序列(Raw Reads)共有787 706 956条,过滤后的序列(Clean Reads)为756 274 435条,比对到参考基因组hg38上的序列(Mapped Data)为753 357 812条,比对到参考基因组hg38上的序列比例(Mapping Rate)为99.61%,平均测序深度为32.17×,发现的变异包括:3 351 535个SNV,845 638个INDEL,7 829个SV,63 397个CNV。
2.3 SNV分布情况与高原适应性分析通过与hg38千人基因组数据库进行比对,在T153的全基因组测序中共发现3 351 535个SNV,所有的SNV替换类型数目如下,T-A:244 500(7.30%),T-C:1 101 779(32.87%),T-G:286 639(8.27%),C-A:277 050(8.55%),C-T:1 153 692(34.42%),C-G:287 875(8.59%)。其中T-C与C-T替换数目较多,两者相加的比例是67.29%,约为2/3,与既往研究发现转换类型即T-C或A-G替换约占整个SNV替换类型的2/3的结论相符合。
利用SIFT(得分 < 0.05)和Polyphen2_HDIV(得分>0.5)两种预测方法,对这些非同义的突变进行致病性预测,然后将T153的这些致病性SNV进行汇总,结果SIFT预测数量9 015个,得分1 593,Polyphen2_HDIV预测数量8 795个,得分1 928,两种方法共同预测数量8 424个,得分873。将这两种预测方法得到的结果绘制成散点图(图 2),并用不同颜色来区分,绿色区域表示两种方法均预测为有害突变的数量。
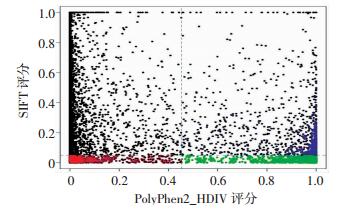
|
| 图 2 T153的SNV致病性预测分析散点图 |
世界上主要有三大高原人群,每个人群的高原适应机制不全相同,包括与藏族高原适应相关的基因脯氨酸羟化酶2(proline hydroxylase 2, PHD2,又名EGLN1)、内皮PAS蛋白1 (endothelial Per-Arnt-Sim domain protein 1, EPAS1)和过氧化物酶体增殖物激活受体ɑ(peroxisome proliferator-activated receptor A, PPARA),与埃塞俄比亚高原人群相关的碱性螺旋-环-螺旋蛋白家族e4(basic helix-loop-helix family, member e4,BHLHE4)、甲状腺素受体β(bhyroid hormone receptor β,THRB)、Vav鸟嘌呤核苷酸交换因子3(vav Guanine nucleotide exchange factor 3,VAV3)、钙结合特异性抗原1 (calcium-binding atopy-related autoantigen 1,CBARA1),以及与安第斯山脉高原人群相关的腺苷酸单磷酸活化蛋白激酶α1(protein kinase,AMP-activated,alpha 1 catalytic subunit,PRKAA1)、一氧化氮合酶2A(nitric oxide synthase 2A,NOS2A)、趋化因子CXC基序受体4[chemokine (C-X-C motif) receptor 4,CXCR4]、转化生长因子α(transforming growth factor alpha,TGFA)。将这些与高原适应相关的基因SNV位点,在全基因组测序分析得到的SNV注释列表中进行检索,得到了相关的SNV分布情况(表 4)。
| 编号 | 藏族人群 | 安第斯山脉人群 | 埃塞俄比亚人群 | ||||||||||
| EGLN1 | EPAS1 | PPARA | TGFA | CXCR4 | PRKAA1 | NOS2A | BHLHE4 | THRB | VAV3 | CBARA1 | |||
| T153 | 258 | 135 | 0 | 280 | 182 | 0 | 0 | 233 | 429 | 421 | 0 | ||
从上述结果可知与T153高原适应相关基因的SNV的数目,但这些SNV到底是呈正相关还是负相关,目前尚未可知。根据以往其他种族的高原适应性研究结果,来验证在T153中是否也存在相应的多态性位点。既往发现EGLN1的多态性位点rs479200和rs480902,以及EPAS1的多态性位点rs6756667均与藏族的高原适应密切相关,ROCK2的多态性位点与高原原发性高血压存在密切相关,将上述这些多态性位点在T153的SNV测序注释列表中检索,其相关的生物信息学分析结果见表 5。发现这7个位点中只有rs2230774的突变属于非同义突变,且发生在外显子区域,其密码子由C突变成A会导致氨基酸由苏氨酸变成天冬酰胺。其SIFT评分为0.874,Polyphen2_HDIV评分为0,根据之前提到的突变的致病性预测方法,两种预测均为无害突变。
| 基因型 | 多态性位点 | 基因 | T153测序 | 所在区域 | 1000g-ALL MAF | |
| 适应 | 适应不良 | |||||
| EGLN1 | rs479200 | C | T | T | 内含子 | 0.663 938 |
| rs480902 | T | C | C | 内含子 | 0.569 489 | |
| EPAS1 | rs6756667 | A | G | G | 内含子 | 0.723 442 |
| ROCK2 | rs978906 | A | G | G | 3′非翻译区 | 0.386 581 |
| rs6753921 | G | A | A | 3′非翻译区 | 0.386 182 | |
| rs10495582 | C | G | G | 内含子 | 0.387 580 | |
| rs2230774 | C | A | A | 外显子 | 0.399 760 | |
| 1000g-ALL MAF:1000Genome所有人群的MAF,MAF为最小等位基因频率,是指在给定人群中的不常见的等位基因发生频率 | ||||||
2.4 拷贝数变异研究
根据高原缺氧对塔吉克族男性性激素水平改变的调查情况,以及性激素水平和男性生育能力存在明显的相关性,探讨塔吉克族男性性激素水平改变的遗传因素。对代表男性生育能力相关的基因DAZ(deleted in azoospermia)、BPY2(basic protein on Y chromosome 2)和CDY(chromodomain Y-like protein)的CNV进行检索,统计其发生CNV区域的数量:DAZ为382、BPY2为168、CDY为71。这些相关基因均位于Y染色体,于是对Y染色体的CNV进行了分析,在Y染色体两端位置存在大量拷贝数变异区域,且以代表拷贝数增加的红色区域较多,蓝色区域代表拷贝数减少,而黑色表示拷贝数无变化,经检索发现DAZ、BPY2和CDY的CNV均为拷贝数减少(图 3)。
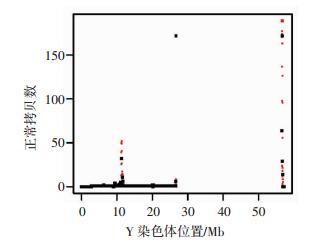
|
| 图 3 T153的Y染色体上CNV分布 |
2.5 T153变异全局统览分析
将全基因组重测序数据中所有变异利用Circos软件进行全局统览(图 4)。整个变异全局统览图从外到内分为九层:第1层代表染色体; 第2层是SNP密度图,密度计算是以每个窗口内SNP数量取log10,柱子越高表示SNP越多,颜色呈红、黄、蓝变化,越红表示SNP越多,越蓝表示SNP数量少; 第3层是Insert的密度图,密度计算是以每个窗口内Insert数量取log10,柱子越高表示Insert越多; 第4层是Delete的密度图,密度计算是以每个窗口内Delete数量取log10,柱子越高表示Delete越多; 第5层是coding区域变异位点的密度图,包括SNP和InDel, 密度计算是以每个窗口内变异位点数量取log10,柱子越高coding区的变异数量越多; 第6层是nocoding区域变异位点的密度图,包括SNP和InDel, 密度计算是以每个窗口内变异位点数量取log10,柱子越高表示nocoding区的变异数量越多; 第7层是CNV位置图,区域大小表示CNV大小; 第8层是SV的位置图,区域大小表示SV的大小,橙色表示缺失DEL,绿色表示插入INS; 第9层是SV的类型关联图,蓝色表示转换INV,红色表示染色体间易位CTX,绿色表示染色体内易ITX。通过变异全局的统览图,对各变异有一个直观和全局性的了解。
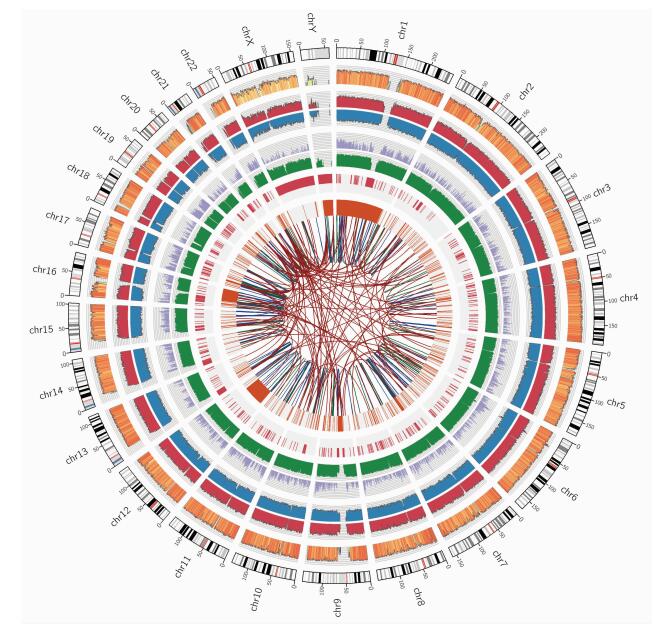
|
| 图 4 T153变异全局统览图 |
3 讨论
本研究通过对中国塔吉克族T153的线粒体DNA全长序列进行分析,经过构建生物进化树,发现T153在母系遗传上与rCRS的遗传距离较近,而与藏族和汉族在母系遗传进化上的遗传距离较远,表明其在母系遗传适应上存在差异。
在藏族人群的高原低氧适应中,缺氧诱导因子(hypoxia inducible factor, HIF)基因家族发挥了重要的作用,包括EGLN1和EPAS1[13-15]。EGLN1基因相关位点rs479200和rs480902的多态性与低氧环境的适应相关[16-17]。这可能与其基因表达水平和藏族血红蛋白浓度以及红细胞数目呈负相关有关,在中国塔吉克族T153的SNV分析中发现这两个位点的基因型与藏族人群不同。EPSA1基因的rs6756667位点A等位基因是藏族人群高原低氧适应的有利因素[18],而T153的rs6756667位点是高原低氧适应不良的基因型G。从高原遗传适应相关的常见SNV位点分析中,发现T153与藏族人群的高原遗传适应机制不同,可能存在其他机制,需要进一步探索。
既往研究发现ROCK2基因的多态性位点rs978906、rs6753921、rs10495582、rs2230774与高原原发性高血压存在显著相关,其组成的单倍群GAGA是高原原发性高血压的易感因素[19]。而T153在这4个位点的测序单倍群正好是GAGA,表明T153也存在高原原发性高血压的遗传易感因素。另外,rs2230774位点的突变属于非同义突变,且发生在外显子区域,其密码子由C突变成A会导致氨基酸由苏氨酸变成天冬酰胺。但是用SIFT和Polyphen2_HDIV两种评分[20]预测均提示该突变为无害突变。这可能是由于单核苷酸突变,虽引起了单个氨基酸的变化,但不足以引起相应蛋白结构和功能的改变,所以不会对机体造成损害,所以通过预测均为无害突变。
在T153的CNV分析当中发现,存在382个DAZ的拷贝数变异区域,且均为缺失变异,在Y染色体分布图上也得到了验证。既往研究已经发现基因DAZ的AZFc区DAZ1/DAZ2的共缺失及其缺失率与男性原发不育相关,表明DAZ拷贝数降低与男性生育能力相关[21],而男性性激素水平与男性生育能力也是存在明显相关性的[22],所以DAZ拷贝数降低很可能是中国塔吉克族族高原环境下性激素水平改变的一个重要影响因素。
总之,通过对中国塔吉克族T153的全基因组重测序分析,我们从线粒体母系遗传上发现T153与藏族和汉族都存在差异,也表明其高原遗传适应与藏族和汉族可能存在不同。而在核基因分析中发现,中国塔吉克族T153的高原遗传适应位点与既往研究[13-15]发现的常见高原遗传适应位点无关,而既往研究[3-4]发现中国塔吉克族是具有一定相关高原适应能力的,所以推测中国塔吉克族人具有其他的高原遗传适应机制。而在CNV分析当中,发现DAZ的拷贝数变异与男性性激素水平改变可能存在相关性。目前,通过对T153单个样本进行全基因组重测序,发现了与中国塔吉克族基因特点相关的SNV和CNV,很多结果只能代表其个体的遗传特点,仍需扩大样本来进一步验证这些SNV和CNV在整个塔吉克族人群中的普遍性,这也是个体重测序的不足之处。本研究所发现的这一系列遗传特点和倾向,将为今后更好地研究中国塔吉克族人相关医学问题提供参考。
| [1] |
李晓明, 杨莹, 彭辉, 等. 全基因组测序在医学应用进展[J].
基因组学与应用生物学, 2015, 34(5): 1071–1075.
LIX M, YANG Y, PENG H, et al. Whole genome sequencing progress in medical application[J]. Genomics Appl Biol, 2015, 34(5): 1071–1075. DOI:10.13417/j.gab.034.001071 |
| [2] |
高亮, 张西洲, 朱光辉, 等. 160例塔吉克族高原世居人群慢性高原病患病率调查[J].
西南国防医药, 2015, 25(4): 462–463.
GAO L, ZHANG X Z, ZHU G H, et al. Investigation on the prevalence of chronic mountainsickness in 160 permanent high altitude natives of Chinese Tajik population[J]. Med J Nat Defend Force Southwest China, 2015, 25(4): 462–463. DOI:10.3969/j.issn.1004-0188.2015.04.050 |
| [3] |
朱伟梁, 沙热甫拉孜·卡达木, 局马巴依·夏古力, 等. 200例帕米尔高原地区塔吉克族人高血压发病情况分析与治疗[J].
中国医学工程, 2015, 23(3): 71–72.
ZHU W L, KDAMU S, XIAGULI J, et al. Analysis and treatment of 200 Tajik hypertensive patients in Pamirs[J]. China Med Eng, 2015, 23(3): 71–72. |
| [4] |
玛依拉·卡米力江, 张德莲, 周玲, 等. 塔什库尔干县塔吉克族50岁及以上中老年人群高血压流行病学调查[J].
新疆医学, 2018, 48(4): 357–359.
KMILIJIANG M, ZHANG D L, ZHOU L, et al. Epidemiological survey of hypertension in the middle age and elderly people among the Tajik nationality living in Tashkurghan[J]. Xinjiang Med J, 2018, 48(4): 357–359. |
| [5] |
刘阳, 黄岚. 高原环境对动脉血压影响的研究进展[J].
医学综述, 2014, 20(6): 963–966.
LIU Y, HUANG L. Effects of high-altitude hypoxia on arterial blood pressure[J]. Med Recapitulate, 2014, 20(6): 963–966. DOI:10.3969/j.issn.1006-2084.2014.06.002 |
| [6] |
高亮, 刘燕, 李彬, 等. 帕米尔高原世居塔吉克族男性性激素水平分析[J].
国际检验医学杂志, 2018, 39(5): 615–617.
GAO L, LIU Y, LI B, et al. Analysis of male sex hormone level in permanent native Tajik in Pamirs[J]. Int J Lab Med, 2018, 39(5): 615–617. DOI:10.3969/j.issn.1673-4130.2018.05.028 |
| [7] | JIANG C H, CUI J H, LIU F Y, et al. Mitochondrial DNA 10609T promotes hypoxia-induced increase of intracellular ROS and is a risk factor of high altitude polycythemia[J]. PLoS ONE, 2014, 9(1): e87775. DOI:10.1371/journal.pone.0087775 |
| [8] | ELHEFNAWI M, JEON S, BHAK Y, et al. Whole genome sequencing and bioinformatics analysis of two Egyptian genomes[J]. Gene, 2018, 668: 129–134. DOI:10.1016/j.gene.2018.05.048 |
| [9] | MARTIN M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnetJ, 2011, 17(1): 10–12. DOI:10.14806/ej.17.1.200 |
| [10] | LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754–1760. DOI:10.1093/bioinformatics/btp324 |
| [11] |
何海金, 张瑶, 刘丽军, 等. 人类起源与进化的高原适应性遗传因素[J].
国外医学(医学地理分册), 2017, 38(1): 97–100.
HE H J, ZHANG Y, LIU L J, et al. Genetics of human origin and evolution: high-altitude adaptation[J]. Foreign Med Sci(Section Medgeogr), 2017, 38(1): 97–100. DOI:10.3969/j.issn.1001-8883.2017.01.026 |
| [12] | ZHANG Y B, LI X, ZHANG F, et al. A preliminary study of copy number variation in Tibetans[J]. PLoS ONE, 2012, 7(7): e41768. DOI:10.1371/journal.pone.0041768 |
| [13] |
陈郁, 高钰琪. 基因组学研究在藏族高原适应遗传机制研究的实践与运用[J].
国际遗传学杂志, 2012, 35(4): 213–216.
CHEN Y, GAO Y Q. Practice and application of genomics research in genetic mechanism of high-altitude adaptation in Tibetan[J]. Int J Genet, 2012, 35(4): 213–216. DOI:10.3760/enm.J.issn.1673-4386.2012.04.005 |
| [14] |
李小薇, 李翠莹. 藏、汉族中遗传差异与高原低氧适应研究[J].
中国输血杂志, 2017, 30(8): 877–879.
LI X W, LI C Y. The correlation of genetic variation and high-altitude hypoxia adaption between Tibetans and Han populations[J]. Chin J Blood Transfus, 2017, 30(8): 877–879. DOI:10.13303/j.cjbt.Issn.1004-549x.2017.08.008 |
| [15] | HUERTA-SÁNCHEZ E, JIN X, ASA N, et al. Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA[J]. Nature, 2014, 512(7513): 194–197. DOI:10.1038/nature13408 |
| [16] |
席焕久. 藏族的高原适应——西藏藏族生物人类学研究回顾[J].
人类学学报, 2013, 32(3): 247–255.
XI H J. Adaptation to high altitude hypoxia environment in Tibetans: a priliminary review of the biological anthropology of Tibetan[J]. Acta Anthropol Sin, 2013, 32(3): 247–255. DOI:10.16359/j.cnki.cn11-1963/q.2013.03.004 |
| [17] |
蒋丽, 殷维瑶, 刘建, 等. EPAS1基因rs6756667多态性与藏族运动员的有氧运动能力[J].
中国组织工程研究, 2016, 20(20): 2957–2963.
JIANG L, YIN W Y, LIU J, et al. EPAS1 gene rs6756667 polymorphism and aerobic exercise capacity of Tibetan athletes[J]. Chin J Tissue Eng Res, 2016, 20(20): 2957–2963. DOI:10.3969/j.issn.2095-4344.2016.20.010 |
| [18] |
陈郁, 蒋春华, 罗勇军, 等. EPAS1基因rs6756667及rs7583392多态性与汉族男性高原红细胞增多症的相关性研究[J].
解放军医学杂志, 2012, 37(12): 1120–1124.
CHEN Y, JIANG C H, LUO Y J, et al. Correlation of rs6756667 and rs7583392 polymorphism of EPAS1 with high altitude polycythemia in male Han population[J]. Med J Chin PLA, 2012, 37(12): 1120–1124. |
| [19] | PANDEY P, MOHAMMAD G, SINGH Y, et al. Polymorphisms and haplotype of ROCK2 associate with high altitude essential hypertension in native high altitude Ladakhi Indian population: a preliminary study[J]. Clin Exp Hypertens, 2016, 38(2): 238–244. DOI:10.3109/10641963.2015.1081231 |
| [20] |
章亮, 苏志煕. 位点致病性预测软件对错义突变的预测效用评估[J].
基因组学与应用生物学, 2016, 35(8): 1916–1925.
ZHANG L, SU Z X. Evaluation of mutation pathogenicity prediction softwares on missense variants[J]. Genomics Appl Biol, 2016, 35(8): 1916–1925. DOI:10.13417/j.gab.035.001916 |
| [21] |
杨元, 肖翠英, 张思仲, 等. 男性原发不育与DAZ基因家族部分拷贝缺失[J].
中华医学遗传学杂志, 2004, 21(5): 444–447.
YANG Y, XIAO C Y, ZHANG S Z, et al. Idiopathic male infertility and partial copy deletion of DAZ gene family[J]. Chin J Med Genetics, 2004, 21(5): 444–447. DOI:10.3760/j.issn:1003-9406.2004.05.007 |
| [22] | ALI H S, BAIG M, RANA M F, et al. Relationship of serum and seminal plasma zinc levels and serum testosterone in oligospermic and azoospermic infertile men[J]. J Coll Physicians Surg Pak, 2005, 15(11): 671–673. |



