2. 730030 兰州,甘肃省骨关节疾病研究重点实验室
2. Key Laboratory of Orthopedics of Gansu Province, Lanzhou, Gansu Province, 730030, China
强直性脊柱炎(ankylosing spondylitis,AS)是一种以骶髂关节和脊柱附着点等中轴关节为主要病变部位的慢性系统性疾病,具有进行性和独立性。AS除了引起脊柱强直和纤维化,还会造成不同程度眼、肺、肌肉、骨骼等的病变。AS常见于年轻人,发病年龄通常在26岁左右,男女比例约为2 :1, 45岁以上的患者出现率低于5%[1]。症状发作年龄越小的AS患者, 他们的预后以及晚期机体功能结局将会越差[2]。在不同国家的研究中,强直性脊柱炎的发病率在(0.5~14.0)/10万[3-4]。在特定人群中,人类白细胞抗原(human leukocyte antigen,HLA)B27的血清阳性率与该疾病的发病率和患病率之间存在一定的相关性[5]。AS常见的治疗包括非甾体抗炎药(non-steroidal anti-inflammatory drugs,NSAIDs)、缓解疾病的抗风湿药、镇痛药以及坚持针对性的定期体位训练[6-7]。近年来,TNF阻断剂在治疗强直性脊柱炎和其他脊柱关节炎中发挥着越来越重要的作用[8], 但这种效果可能是一种类效应[9-10]。因此,针对AS的特异性诊断以及有效治疗仍值得继续探究。
由于AS的发病机制目前依旧不十分清楚,经典的AS诊断主要依赖于患者血清免疫学检测、影像学及患者AS疾病症状等。因此针对AS发病及特异性诊断相关的研究显得十分重要。本研究以AS患者膝关节滑膜的高通量基因芯片为研究对象,运用生物信息学相关知识筛选出AS中有意义的DEGs并行功能、通路和PPI等分析,从而为AS的发病、诊断及预后提供新的研究视角和理论基础。
1 材料与方法 1.1 强直性脊柱炎相关基因芯片获取从美国国立生物技术信息(National Center for Biotechnology Information,NCBI)数据库中的GEO数据库(http://www.ncbi.nlm.nih.gov/geo)中下载强直性脊柱炎基因芯片数据。数据获取条件为:①数据来自人类mRNA表达芯片的原始数据;②强直性脊柱炎与对应条件下有对照的实验。以ankylosing spondylitis为检索词,获得Thomas G上传的GSE41038芯片数据。本实验采用Illumina Human Ref-8 v3.0 expression beadchip的15个样本。
1.2 基因芯片的处理及DEGs的筛选将GEO的原始数据运用GEO自带分析功能GEO2R行DEGs的筛选,其中8个属于强直性脊柱炎及分类未定脊柱炎的膝关节滑膜组织样本为实验组,7个属于骨关节炎及健康正常组织的膝关节滑膜组织样本为对照组。差异基因的筛选条件为:P < 0.05、logFC的绝对值≥1(差异表达倍数≥2),其中logFC≥1的为上调,-logFC≤1为下调。
1.3 DEG的GO富集分析和KEGG信号通路分析利用DAVID6.8(https://david-d.ncifcrf.gov/)对差异基因行Gene Ontology(GO)富集分析。将筛选出的DEGs上传至DAVID数据库,并将List和Background选取为homosapiens,选取GO中的GOTERM_BP_DIRECT,GOTERM_CC_DIRECT和GOTERM_MF_ DIRECT进行GO功能富集分析。选取PATHWAY中的KEGG_PATHWAY选项行信号通路分析。GO富集分析和KEGG信号通路分析的筛选条件均为:P < 0.05。
1.4 蛋白的相互作用网络分析相互作用基因库检索工具(Search Tool for the Retrieval of Interacting Genes,STRING)数据库能够构建已知蛋白质与预测蛋白质之间的相互作用,通过对蛋白质之间相互作用的可能性进行打分,评价相互作用可能性的大小[11]。构建差异基因编码蛋白相互作用网络(protein-protein interaction,PPI)的筛选条件为代表中度相互作用可能性的阈值(结合分值>0.4)。然后将STRING数据库构建出的蛋白质相互作用网络结果导入信息分析学软件Cytoscape(3.4.0版本)中进行可视化分析,并根据蛋白之间的连接度找出Hub基因或其编码的处在关键节点的蛋白质,筛选条件为:degree≥6。最后运用Cytoscape中的iRegulon插件进行预测DEGs中Hub基因的转录因子,并分析这些转录因子所调节的Hub基因;筛选条件为:标准化富集分数(normalized enrichment score,NES)>3[12]。
1.5 统计学分析GSE41038原始数据运用GEO2R中自带统计学方法Benjamini&Hochberg(false discovery rate,FDR)处理,用SAM法筛选差异基因。GO分析通过DAVID数据库自带统计学工具,使用弗朗尼校正法(Bonferroni)、本杰明假阳性率(Benjamini false discovery rate)和靴带法(Bootstrap)等进行检验。KEGG分析通过DAVID数据库自带统计学工具进行Fisher精确概率检验及基因集富集分析(gene set enrichment analysis)。Cytoscape中的iRegulon模块运用自带的位置权重矩阵(position weight matrix,PWM)和Motif2TF处理。
2 结果 2.1 DEGs的筛选运用GEO2R功能筛选AS有关的DEGs。我们得到190个差异基因,其中上调的基因75个,下调的基因115个。上下调DEGs前10位结果见图 1。
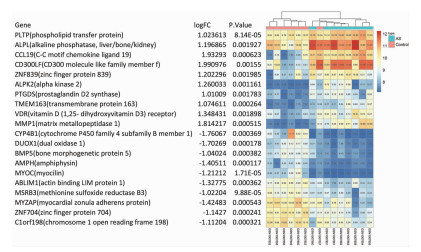
|
| 每个方格中的数字代表基因在样本中的表达量[取log2(表达量)后的结果],红色代表高表达,蓝色代表低表达 图 1 GSE41038样本集中的DEGs |
2.2 DEGs的GO富集分析
GO富集分析显示:在生物学过程层面DEGs主要涉及炎症反应、白细胞迁移和对缺氧的反应等23个功能簇(图 2A)。在细胞组成层面DEGs主要涉及胞外区、细胞外间隙和细胞外基质等6个功能簇(图 2B)。在分子功能层面DEGs主要涉及细胞外基质结构成分、肝素结合和血红素结合等7个功能簇(图 2C)。部分GO功能富集分析结果见图 2。
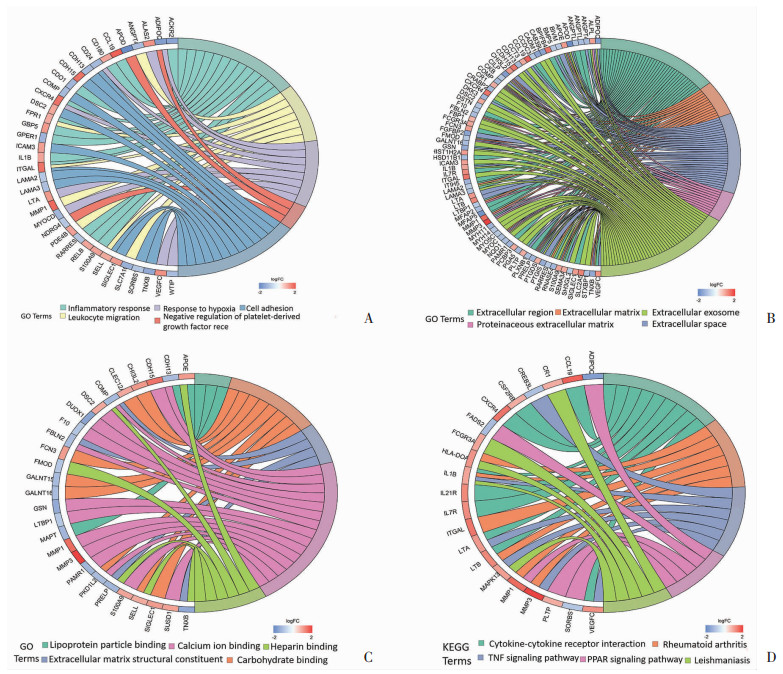
|
| A:生物学过程的前5个功能簇;B:细胞组成的前5个功能簇;C:分子功能的前5个功能簇;D:KEGG信号通路的前5个信号通路 图 2 DEGs的GO富集分析和KEGG通路分析 |
2.3 KEGG通路分析
KEGG通路分析显示DGEs主要参与类风湿性关节炎、细胞因子-细胞因子受体相互作用和PPRA信号通路等10条信号通路。其中类风湿性关节炎相关的通路最为显著富集。KEGG通路分析结果见图 2D。DEGs在风湿性关节炎相关的通路的富集情况见图 3A。
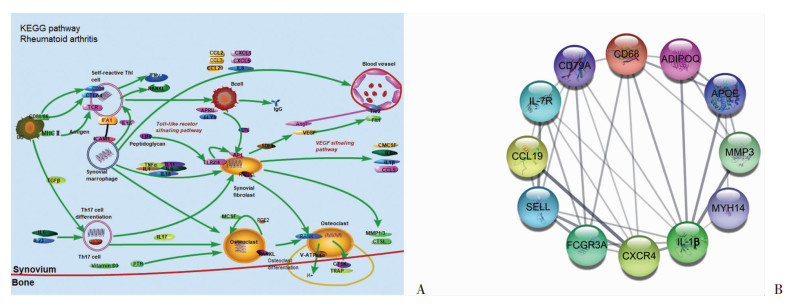
|
| A:通路中包含强直性脊柱炎中差异表达基因有MHCⅡ、IFA1、ICAM1、IL-1、LTB、IL-1β和MMP1/3;B:点代表蛋白,线代表相互作用,线条越粗表示相互作用越强 图 3 风湿性关节炎相关通路(A)和12个Hub基因编码蛋白之间的相互作用网络关系(B) |
2.4 编码蛋白相互作用网络构建
STRING数据库构建的PPI网络包括185个节点蛋白的158条关系。根据Degree筛选出表达上调的CXCR4、SELL、CD79A、MMP3、CD68、CCL19、IL-7R、IL-1β、MYH14、APOE、FCGR3A和表达下调的ADIPOQ等12个强直性脊柱炎基因编码的蛋白。其中,IL-1β和CXCR4是该PPIN中蛋白存在相互作用量最多的2个蛋白,删除IL-1β和CXCR4使该蛋白相互作用网络结构涣散,蛋白之间相互作用关系明显减少,因而IL-1β和CXCR4为PPIN的核心节点,提示IL-1β和CXCR4对AS的发病、诊断或者预后有重要的作用。蛋白相互作用网络见图 3B。
2.5 IL-1β和CXCR4两个基因功能及其相关疾病谱分析运用生物技术信息基因云(GCBI, http://www.gcbi.com.cn)在线数据库的基因雷达对IL-1β和CXCR4两个基因功能进行分析。结果显示在已发表的有关IL-1β和CXCR4的文献中,IL-1β和CXCR4主要与感染、炎症和肿瘤相关,提示IL-1β和CXCR4参与AS的病理生理并起重要作用。基因雷达调控网络显示IL-1β与BRN1、ZXDB及ZXDA等53个蛋白相互作用;CXCR4与AP2ALPHA、AR及ZNF333等70个蛋白相互作用。见图 4。
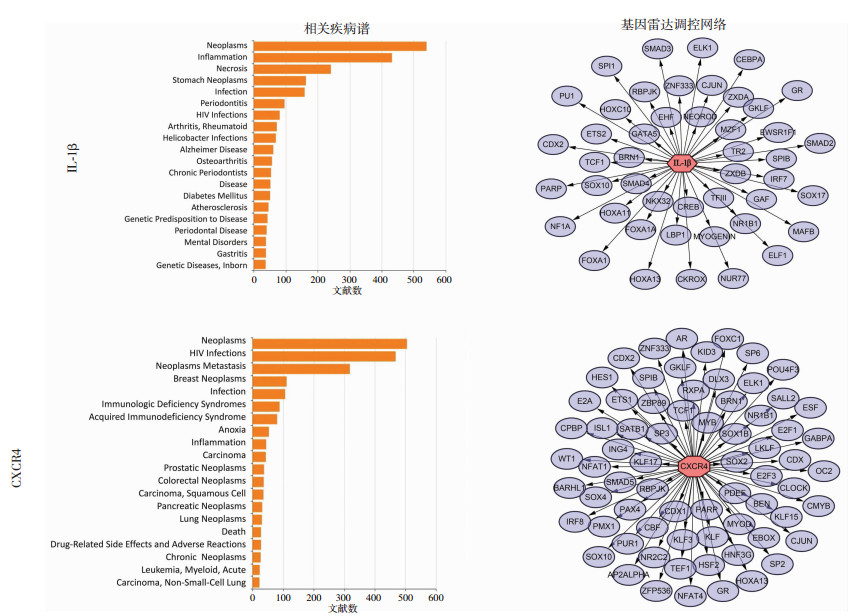
|
| 图 4 IL-1β、CXCR4相关疾病谱及其基因雷达调控网络 |
2.6 12个Hub基因相关的转录因子分析
运用生物信息分析学软件Cytoscape(3.4.0版本)中的iRegulon插件预测DEGs中Hub基因的转录因子,并分析这些转录因子所调节的DEGs[12]。发现与CXCR4、SELL、CD79A、MMP3、CD68、ADIPOQ、CCL19、IL-7R、IL-1β、MYH14、APOE和FCGR3A等相关的转录因子有BCL3、POLR2A、FOXJ1、GFI1、NR1H2、FOXN4、KLF16、AR、FOS、SRF、IRF4、TGIF1、GATA3、RCOR1、JUND、PBX3、NFATC1、ATF3、IRF8、SOX10、SIN3A、EGR1、ETS1、EVX2、CFL2、MTA3、IRF1、MYLK、MYF5、BCL6、POU2F2、GLI3、BCLAF1、PRKAA2、STAT5A、TCF12、ATF2、ARID3A、E2F1、PML和BRF2等41个。部分结果见表 1。部分Hub基因相关转录因子的靶基因作用关系见图 5。
| TF | NES | 靶基因数目 | 转录调控元件数目 |
| BCL3 | 6.102 | 7 | 5 |
| POLR2A | 5.978 | 4 | 6 |
| FOXJ1 | 5.899 | 8 | 10 |
| GFI1 | 5.807 | 4 | 3 |
| NR1H2 | 5.786 | 5 | 3 |
| FOXN4 | 5.679 | 2 | 5 |
| KLF16 | 5.515 | 5 | 7 |
| AR | 5.464 | 8 | 15 |
| FOS | 5.382 | 2 | 22 |
| SRF | 5.357 | 5 | 4 |
| IRF4 | 5.254 | 8 | 7 |
| TGIF1 | 5.249 | 7 | 4 |
| GATA3 | 5.035 | 6 | 3 |
| RCOR1 | 4.902 | 3 | 1 |
| JUND | 4.894 | 5 | 4 |
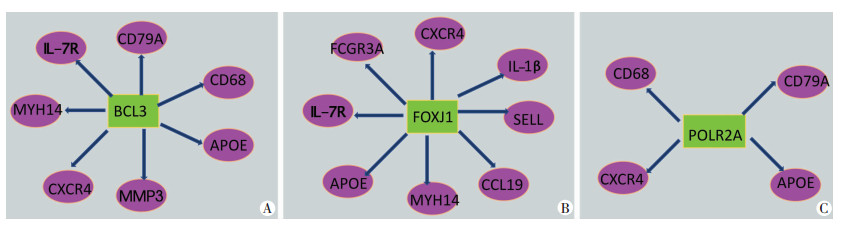
|
| A:作用于BCL3的转录因子相互作用网络;B:作用于FOXJ1的转录因子相互作用网络;C:作用于POLR2A的转录因子相互作用网络;绿色方格代表差异表达基因,紫色椭圆代表作用于靶基因的TFs 图 5 Hub基因相关转录因子的靶基因作用关系 |
3 讨论
强直性脊柱炎是一种常见的炎性风湿性疾病,表现为特征性的炎性背痛,并常常导致患者的机体结构和功能出现障碍以及生活质量下降和精神障碍[13-14]。年龄、性别、遗传、环境、感染、内分泌和自身免疫系统等在AS的发生、发展中发挥着重要作用。AS发病机制涉及众多基因,以及其转录因子和基因参与的相关信号通路。通过挖掘AS相关的基因表达变化及其背后的分子机制,可以为AS的诊断提供新的依据和找出新的药物治疗靶点。为深入了解强直性脊柱炎发生、发展的分子机制,应筛选可作为强直性脊柱炎诊断的关键基因和治疗药物靶点。经典的AS检验方法多为血清学检测,本研究利用生物信息学分析方法对GEO数据库下载强直性脊柱炎与对照组的膝关节滑膜组织的生物芯片数据进行差异基因分析,对差异基因进行功能聚类分析、功能富集分析和相互作用网络分析。通过对强直性脊柱炎与对照的膝关节滑膜组织的比较,我们得到190个差异基因,其中上调的基因75个,下调的基因115个。通过GO分析,本研究发现AS患者的DEGs主要涉及炎症反应、白细胞迁移、胞外区、细胞外基质、脂蛋白颗粒结合和碳水化合物结合等功能簇;KEGG分析提示其在涉及风湿性关节炎和细胞因子-细胞因子受体相互作用等通路中富集,这些参与KEGG信号通路的分子可以为探究AS的发病分子机制提供理论基础。
运用STRING数据库进一步分析差异基因并筛选出CXCR4、SELL、CD79A、MMP3、CD68、ADIPOQ、CCL19、IL-7R、IL-1β、MYH14、APOE和FCGR3A等12个强直性脊柱炎相关的基因编码的蛋白。对上述12个基因进行文献挖掘,发现CXCR4参与炎症[15],在属于免疫性疾病的干眼病中CXCR4表达增加,参与干眼病的发生、发展[16]。在系统性硬化病中SELL的mRNA表达增高[17]。CD79A属于与MHC Ⅱ类相关联的跨膜蛋白,B细胞中的CD79A/B与MHC Ⅱ类的关联有助于早期的抗原与特异性IgM应答[18]。MMP3在类风湿性关节炎中高表达,并可以作为治疗靶点[19];在活动期AS患者棘上韧带血管中MMP3高表达,并有可能参与韧带退变骨化的病理过程[20];治疗类风湿性关节炎时,依那西普可能通过抑制CD68和MMP3的表达来起作用[21]。ADIPOQ的水平是系统性红斑狼疮(SLE)蛋白尿相关的有用标志物,与蛋白尿水平呈正相关[22]。CCL19可以作为炎症调节的治疗靶点[23];已有文献证明CCL19和CCL21在许多疾病中介导细胞迁移和血管生成,两种趋化因子CCL19和CCL21在血清中表现出显著相关的表达,表明对AS发病机制有协同作用,可能作为AS患者韧带骨化的启动子[24]。IL-7R+ B细胞在关节炎中具有促炎性作用,通过抑制IL-7R信号传导可以抑制交感神经释放去甲肾上腺素[25]。IL-1β为常见的炎症因子,在本研究中为显著差异表达上调的基因,但有研究表明IL-1β与AS存在矛盾的相关性[26]。在骨关节炎中,药物Gypenoside(GP)剂量依赖性地抑制人骨关节炎软骨细胞中IL-1β诱导的NO和PGE2的产生,同时抑制受IL-1β正调节的MMP3和MMP13的表达以及NF-κB的活化[27]。FCGR3A在欧洲人中与风湿性关节炎的发病具有相关性[28]。
运用生物信息分析学软件Cytoscape中的iRegulon插件预测DEGs中Hub基因的转录因子,并分析这些转录因子所调节的DEGs,发现与CXCR4、SELL、CD79A、MMP3、CD68、ADIPOQ、CCL19、IL-7R、IL-1β、MYH14、APOE和FCGR3A等相关的转录因子有BCL3、POLR2A、FOXJ1、GFI1和NR1H2等41个;其中有部分TF在类风湿性关节炎中的作用已得到实验证实。比如本研究中Hub基因相关的转录因子ETS1,REDLICH等[29]以及GOLDRING等[30]的研究均表明在免疫系统过度激活的类风湿关节炎的滑膜细胞中通过炎症因子IL-1β、IL-1以及TNF-α等促进EST1的表达。SHAN等[31]研究表明ETS1与中国汉族人群AS的发病呈显著正相关。GATA3作为Th2细胞特异性转录因子,其mRNA表达在AS患者中明显升高[32],BAI等[33]的研究表明通过PCR技术检测发现GATA3的量明显高于健康对照组。因此EST1和GATA3可能参与AS的发生、发展并发挥重要作用。最近的研究表明IRF8在系统性红斑狼疮中高表达,并作为参与系统性红斑狼疮发生的miRNA-451a的作用靶点[34]。在自身免疫性疾病桥本氏甲状腺炎的患者中,IRF8的rs17445836是与桥本氏甲状腺炎发展相关的可能的遗传变体,IRF8的rs17445836与桥本氏甲状腺炎的甲状腺抗体的产生有关[35],因此,我们猜测IRF8对AS的发生发展起相应的调控作用。
综上所述,本研究提示新发现的Hub基因及其相关的转录因子可以为AS的诊治提供新的研究方向和理论依据。同时,基于本实验研究方法可操作性以及生物信息学技术的科学性,可以为基础医务工作者提供科研思路和研究方法。
| [1] | FELDTKELLER E, KHAN M A, VAN DER HEIJDE D, et al. Age at disease onset and diagnosis delay in HLA-B27 negative vs. positive patients with ankylosing spondylitis[J]. Rheumatol Int, 2003, 23(2): 61–66. DOI:10.1007/s00296-002-0237-4 |
| [2] | STONE M, WARREN R W, BRUCKEL J, et al. Juvenile-onset ankylosing spondylitis is associated with worse functional outcomes than adult-onset ankylosing spondylitis[J]. Arthritis Rheum, 2005, 53(3): 445–451. DOI:10.1002/art.21174 |
| [3] | DEAN L E, JONES G T, MACDONALD A G, et al. Global prevalence of ankylosing spondylitis[J]. Rheumatology(Oxford), 2014, 53(4): 650–657. DOI:10.1093/rheumatology/ket387 |
| [4] | BAKLAND G, NOSSENT H C, GRAN J T. Incidence and prevalence of ankylosing spondylitis in Northern Norway[J]. Arthritis Rheum, 2005, 53(6): 850–855. DOI:10.1002/art.21577 |
| [5] | KHAN M A. Epidemiology of HLA-B27 and arthritis[J]. Clin Rheumatol, 1996, 15(Suppl1): 10–12. DOI:10.1007/BF03342637 |
| [6] | RADNER H, RAMIRO S, BUCHBINDER R, et al. Pain management for inflammatory arthritis (rheumatoid arthritis, psoriatic arthritis, ankylosing spondylitis and other spondyloarthritis) and gastrointestinal or liver comorbidity[J]. Cochrane Database Syst Rev, 2012(1): CD008951. DOI:10.1002/14651858.CD008951.pub2 |
| [7] | BREWERTON D A, HART F D, NICHOLLS A, et al. Ankylosing spondylitis and HL-A 27[J]. Lancet, 1973, 1(7809): 904–907. |
| [8] | ZOCHLING J, VAN DER HEIJDE D, BURGOS-VARGAS R, et al. ASAS/EULAR recommendations for the management of ankylosing spondylitis[J]. Ann Rheum Dis, 2006, 65: 442–452. DOI:10.1136/ard.2005.041137 |
| [9] | HEIBERG M S, NORDVAG B Y, MIKKELSEN K, et al. The comparative effectiveness of tumor necrosis factor-blocking agents in patients with rheumatoid arthritis and patients with ankylosing spondylitis: a six-month, longitudinal, observational, multicenter study[J]. Arthritis Rheum, 2005, 52(8): 2506–2512. DOI:10.1002/art.21209 |
| [10] | PARK W, YOO D H, MIRANDA P, et al. Efficacy and safety of switching from reference infliximab to CT-P13 compared with maintenance of CT-P13 in ankylosing spondylitis: 102-week data from the PLANETAS extension study[J]. Ann Rheum Dis, 2017, 76(2): 346–354. DOI:10.1136/annrheumdis-2015-208783 |
| [11] | FAZEKAS D, KOLTAI M, TVREI D, et al. SignaLink 2-a signaling pathway resource with multi-layered regulatory networks[J]. BMC SystBiol, 2013, 7: 7. DOI:10.1186/1752-0509-7-7 |
| [12] | JANKY R, VERFAILLIE A, IMRICHOVÁ H, et al. iRegulon: from a gene list to a gene regulatory network using large motif and track collections[J]. PLoS Comput Biol, 2014, 10(7): e1003731. DOI:10.1371/journal.pcbi.1003731 |
| [13] | BAYSAL Ö, DURMUŞ B, ERSOY Y, et al. Relationship between psychological status and disease activity and quality of life in ankylosing spondylitis[J]. Rheumatol Int, 2011, 31(6): 795–800. DOI:10.1007/s00296-010-1381-x |
| [14] | BROPHY S, CALIN A. Definition of disease flare in ankylosing spondylitis: the patients'perspective[J]. J Rheumatol, 2002, 29(5): 954–958. |
| [15] | DE FILIPPO K, RANKIN S M. CXCR4, the master regulator of neutrophil trafficking in homeostasis and disease[J]. Eur J Clin Invest, 2018: e12949. DOI:10.1111/eci.12949 |
| [16] | NICOLLE P, LIANG H, REBOUSSIN E, et al. Proinflammatory markers, chemokines, and enkephalin in patients suffering from dry eye disease[J]. Int J Mol Sci, 2018, 19(4): 1221. DOI:10.3390/ijms19041221 |
| [17] | DASHTI N, MAHMOUDI M, GHARIBDOOST F, et al. Evaluation of ITGB2 (CD18) and SELL (CD62L) genes expression and methylation of ITGB2 promoter region in patients with systemic sclerosis[J]. Rheumatol Int, 2018, 38(3): 489–498. DOI:10.1007/s00296-017-3915-y |
| [18] | KATIKANENI D S, JIN L. B cell MHC class Ⅱ signaling: A story of life and death[J]. Human Immunol, 2018.[Epub ahead of print]. DOI: 10.1016/j.humimm.2018.04.013. |
| [19] | SIEBUHR A S, KJELGAARD-PETERSEN C F, SUN S, et al. Suppression of active, but not total MMP-3, is associated with treatment response in a phase Ⅲ clinical study of rheumatoid arthritis[J]. Clin Exp Rheumatol, 2018, 36(1): 94–101. |
| [20] |
许红飞, 初同伟, 张超, 等. MMP-3在强直性脊柱炎棘上韧带退变中的作用[J].
第三军医大学学报, 2012, 34(19): 1957–1960.
XU H F, CHU T W, ZHANG C, et al. Role of matrix metalloproteinase 3 in supraspinal ligament degeneration due to ankylosing spondylitis[J]. J Third Mil Med Univ, 2012, 34(19): 1957–1960. |
| [21] | SUZUKI Y, INOUE K, CHIBA J, et al. Histological analysis of synovium by treatment of etanercept for rheumatoid arthritis[J]. Int J Rheumatic Dis, 2009, 12(1): 7–13. DOI:10.1111/j.1756-185X.2009.01372.x |
| [22] | DIAZ-RIZO V, BONILLA-LARA D, GONZALEZ-LOPEZ L, et al. Serum levels of adiponectin and leptin as biomarkers of proteinuria in lupus nephritis[J]. PLoS ONE, 2017, 12(9): e0184056. DOI:10.1371/journal.pone.0184056 |
| [23] | AKHAVANPOOR M, GLEISSNER C A, GORBATSCH S, et al. CCL19 and CCL21 modulate the inflammatory milieu in atherosclerotic lesions[J]. Drug Des Devel Ther, 2014, 8: 2359–2371. DOI:10.2147/DDDT.S72394 |
| [24] | QIN Y, HE L D, SHENG Z J, et al. Increased CCL19 and CCL21 levels promote fibroblast ossification in ankylosing spondylitis hip ligament tissue[J]. BMC Musculoskelet Disord, 2014, 15: 316. DOI:10.1186/1471-2474-15-316 |
| [25] | PONGRATZ G, ANTHOFER J M, MELZER M, et al. IL-7 receptor α expressing B cells act proinflammatory in collagen-induced arthritis and are inhibited by sympathetic neurotransmitters[J]. Ann Rheum Dis, 2014, 73(1): 306–312. DOI:10.1136/annrheumdis-2012-202944 |
| [26] | MONNET D, KADI A, IZAC B, et al. Association between the IL-1 family gene cluster and spondyloarthritis[J]. Ann Rheum Dis, 2012, 71(6): 885–890. DOI:10.1136/annrheumdis-2011-200439 |
| [27] | WAN Z H, ZHAO Q. Gypenoside inhibits interleukin-1β-induced inflammatory response in human osteoarthritis chondrocytes[J]. J Biochem Mol Toxicol, 2017, 31(9): e21926. DOI:10.1002/jbt.21926 |
| [28] | LEE Y H, BAE S C, SONG G G. FCGR2A, FCGR3A, FCGR3B polymorphisms and susceptibility to rheumatoid arthritis: a meta-analysis[J]. Clin Exp Rheumatol, 2015, 33(5): 647–654. |
| [29] | REDLICH K, KIENER H P, SCHETT G, et al. Overexpression of transcription factor Ets-1 in rheumatoid arthritis synovial membrane:regulation of expression and activation by interleukin-1 and tumor necrosis factor alpha[J]. Arthritis Rheum, 2001, 44(2): 266–274. DOI:10.1002/1529-0131(200102)44:2<266::AID-ANR43>3.0.CO;2-G |
| [30] | GOLDRING M B, OTERO M, TSUCHIMOCHI K, et al. Defining the roles of inflammatory and anabolic cytokines in cartilage metabolism[J]. Ann Rheum Dis, 2008, 67(Suppl 3): 75–82. DOI:10.1136/ard.2008.098764 |
| [31] | SHAN S, DANG J, LI J, et al. ETS1 variants confer susceptibility to ankylosing spondylitis in Han Chinese[J]. Arthritis Res Ther, 2014, 16(2): R87. DOI:10.1186/ar4530 |
| [32] |
高静.强直性脊柱炎患者外周血Th1/Th2相关转录因子和细胞因子表达水平的研究[D].合肥: 安徽医科大学, 2012.
GAO J. The study on the levels of Th1/Th2 associated transcription factors and some cytokines in patients with Ankylosing spondylitis[D]. Hefei: Anhui Med Univ, 2012. |
| [33] | BAI L, LIU Y, HOU S, et al. Association of T-Bet, GATA-3, RORC, and FOXP3 copy number variations with acute anterior uveitis with or without ankylosingspondylitis in Chinese Han[J]. Invest Ophthalmol Vis Sci, 2016, 57(4): 1847–1852. DOI:10.1167/iovs.15-17960 |
| [34] | CHENG J, WU R, LONG L, et al. miRNA-451a targets IFN regulatory factor 8 for the progression of systemic lupus erythematosus[J]. Inflammation, 2017, 40(2): 676–687. DOI:10.1007/s10753-017-0514-8 |
| [35] | LIN J D, WANG Y H, LIU C H, et al. Association of IRF8 gene polymorphisms with autoimmune thyroid disease[J]. Eur J Clin Invest, 2015, 45(7): 711–719. DOI:10.1111/eci.12463 |



