2. 400038 重庆,陆军军医大学(第三军医大学)第三附属医院(野战外科研究所)癌症中心;
3. 102218 北京,北京清华长庚医院肝胆胰中心
2. Cancer Center, Institute of Surgery Research, Third Affiliated Hospital, Army Medical University (Third Military Medical University), Chongqing, 400042;
3. Center of Hepatobiliary Pancreatic Disease, Beijing Tsinghua Changgung Hospital, Beijing, 102218, China
YAN Jun, E-mail: yanj843@medmail.com.cn
肝细胞癌(hepatocellular carcinoma,HCC,简称肝癌)是目前世界上发病率第5、致死率第3的恶性肿瘤[1-2]。我国新发和死亡病例占全世界50%以上,是我国致死率第2高的癌症。肝细胞癌具有高侵袭性和高复发性等特征,是制约其疗效的“阿喀琉斯之踵”,但影响其预后的关键机制仍未阐明[2]。我国90%以上的肝癌病例合并乙型肝炎病毒(hepatitis B virus,HBV)感染。因此,进一步深入研究HBV引发肝癌的发病机制,可为肝细胞癌的防治提供新思路。
长链非编码RNA(long non-coding RNA, lncRNA)是长度大于200个核苷酸的非编码RNA[3-4]。研究表明lncRNA在表观遗传调控、基因表达剂量补偿效应、细胞周期和细胞分化调控等众多生物学事件中发挥着重要作用[5-6]。近期研究报道了一些在肝细胞癌发生过程中具有关键作用的lncRNA[7-11],但是lncRNA在HBV相关肝细胞癌中的作用研究才刚刚开始。有研究发现lncRNA-DANCR在HBV相关肝细胞癌中具有重要作用[12]。我们前期系统分析了HBV相关肝细胞癌进展过程中的lncRNA谱,发现了包括LINC01018、EHHADH-AS1和F11-AS1在内的多个关键lncRNA。与这些lncRNA具有共表达关系的多个编码基因与肝细胞癌患者的生存曲线具有极显著相关性[13]。同时通过生物信息学方法构建了内源性竞争性RNA网络(competitive endogenous RNA,ceRNA),发现上述lncRNA也参与多个ceRNA网络的调控[14]。这些研究提示它们可能在HBV相关的肝细胞癌发生、发展中具有关键作用,但是其具体的作用机制尚不明确。
本研究通过分析基因表达数据库GSE54238[12]中HBV感染导致肝硬化、早期肝癌和晚期肝癌的数据,深入研究EHHADH-AS1在HBV相关肝细胞癌发展过程中的表达改变情况、与其共表达的基因参与的信号通路;在此基础上,通过基因集富集分析法(gene set enrichment analysis, GSEA)分析HBV相关肝细胞癌组织中EHHADH-AS1高表达与低表达组中富集的KEGG信号通路,寻找EHHADH-AS1参与HBV相关肝细胞癌发展的可能机制;最后通过实验进一步验证EHHADH-AS1在HBV相关肝细胞癌发生、发展中的作用。
1 材料与方法 1.1 主要试剂胰蛋白酶、胎牛血清和高糖DMEM培养基购自美国HyClone公司;引物和siRNA由美国Invitrogen公司合成;TRIzol试剂购自德国Roche Molecular Biochemicals公司;逆转试剂盒和2× SyberGreen mixture试剂购自德国DBI公司;MTT试剂购自美国Sigma公司;流式细胞仪检测所需的试剂盒购于美国BD Bioscience公司。
1.2 EHHADH-AS1在HBV相关肝细胞癌组织的表达水平分析通过美国国立生物信息中心(National Center for Biotechnology Information,NCBI)的基因表达数据库(GEO),获取GSE54238[12]包含10个健康人、13个早期肝细胞癌和13个晚期肝细胞癌患者的肝组织芯片数据,通过R语言Limma包分析EHHADH-AS1在健康人和早期肝癌、晚期肝癌患者组织中的差异表达情况。
1.3 与EHHADH-AS共表达的编码基因的共表达网络的构建及信号通路富集分析通过R语言cor命令计算EHHADH-AS1与所有mRNA的皮尔森相关系数(Pearson correlation coefficient,PCC),选取相关系数的绝对值(|correlation|,|cor|) ≥0.7,且P<0.05的mRNA,分析与EHHADH-AS1具有共表达关系的mRNA,并构建共表达网络[15]。
针对与EHHADH-AS1具有共表达关系的所有编码基因,使用DAVID 6.8(https://david.ncifcrf.gov/)进行GO富集分析[16]和KEGG富集分析[17]。以P<0.05为差异具有统计学意义。
1.4 EHHADH-AS1以及与EHHADH-AS共表达的编码基因的转录因子的调控分析通过R语言TFBSTools包[18]采用JASPAR[19]中转录因子的结合信息,对EHHADH-AS1共表达基因中细胞周期(cell cycle)信号通路相关差异基因(上调倍数>3倍)的转录起始位置上游2 000以及下游500做转录因子结合预测。继而将可结合细胞周期相关差异基因启动区域的转录因子与EHHADH-AS1做转录因子结合预测分析,选取hit score>0.8作为最低阈值。
1.5 肝癌组织中EHHADH-AS1高表达和低表达组样本的GSEA分析根据EHHADH-AS1表达的丰度情况,将肝癌早期和晚期组织样本分为EHHADH-AS1高表达组和EHHADH-AS1低表达组,并进一步通过GSEA软件(http://software.broadinstitute.org/gsea/index.jsp)对这两个组进行KEGG基因富集分析。采用Kolmogorov-Smirnov检验判断具有统计学意义的KEGG信号通路。
1.6 细胞培养HepG2和Huh7细胞均来源于美国ATCC,使用含10%胎牛血清的高糖DMEM培养液,并置于37 ℃、5%CO2培养箱中培养,细胞生长至90%融合状态后传代,取处于对数期生长且状况良好的细胞以3×105/mL接种于6孔板或96孔板进行后续实验。
1.7 EHHADH-AS1 siRNA根据lncRNA的目的序列设计了2对siRNA序列,将siRNA的潜在目的序列在NCBI进行BLAST比对,确保与其他基因没有同源性。从中筛选抑制效率较高的siRNA序列,用于后续实验研究。本研究涉及的敲低效率最高的siRNA及引物序列如下,EHHADH-AS1 siRNA:正义链,5′-GGAUUUGGAGAGUUGAGAA-TT-3′;反义链,5′-UUCUCAACUCUCCAAAUCCTT-3′。阴性对照(NC)siRNA:正义链,5′-UUCUCCGAACGUGUCACGUTT-3′;反义链,5′-ACGUGACACGUUCGGAGAATT-3′。
各细胞株铺板后待其长至30%~50%融合度后,参照Lipofectamine试剂盒进行siRNA转染操作[20]。转染6 h后,细胞换为完全培养液继续培养24 h后进行后续相关检测。
1.8 定量PCR检测EHHADH-AS1和相关细胞周期基因的表达细胞经过处理后经TRIzol试剂提取RNA。使用德国DBI公司Bestar qPCR RT Kit逆转试剂盒并参照说明书将RNA逆转为cDNA。参照德国DBI公司2×SyberGreen mixture试剂盒(DBI Bioscience)的方法对获取的cDNA进行实时荧光定量PCR反应,检测EHHADH-AS1、相关细胞周期基因和内参基因β-actin的表达。引物序列参照PrimerBank(https://pga.mgh.harvard.edu/primerbank/)的序列合成(表 1)。
| 基因 | 引物序列 | 产物长度 |
| EHHADH-AS1 | 正义链:5′-CCCCAAGGAGATACAGAAGTGT-3′ 反义链:5′-CAGCTGGACACTCATCCCTG-3′ |
178 bp |
| CHAF1A | 正义链:5′-TCCCATCTCGCTGAAGAGGA-3′ 反义链:5′-CATACGTCACCCCTGCTCTC-3′ |
295 bp |
| STMN1 | 正义链:5′-GCCGTCGAGGAGACAATAGG-3′ 反义链:5′-TTCAGACAATGGGGAACCCG-3′ |
94 bp |
| PSRC1 | 正义链:5′-CCGGAGTAATGATAGGAAGGGG-3′ 反义链:5′-GCTTTTGGATGCAGGGAACAG-3′ |
115 bp |
| PTTG1 | 正义链:5′-CTCGGACTGCTAACTGGACC-3′ 反义链:5′-AAACAGCGGAACAGTCACGG-3′ |
154 bp |
| AURKA | 正义链:5′-GTCAAGTCCCCTGTCGGTTC-3′ 反义链:5′-GGTCCATGATGCCTCTAGCTG-3′ |
145 bp |
1.9 细胞活力实验
HepG2和Huh7细胞正常培养后铺于24孔板,过夜待细胞长至30%左右时转染si-EHHADH-AS1 6 h后换液;换液后继续培养16 h后消化细胞,并铺板至96孔板,24 h后进行MTT检测。
1.10 细胞增殖实验HepG2和Huh7细胞正常培养后铺于24孔板,过夜待细胞长至30%左右时转染EHHADH-AS1 siRNA 6 h后换液。流式细胞仪检测前4 h加入BrdU,并在转染后24 h后收集细胞。参考美国BD Pharmingen公司APC Brdu Flow Kit(货号557892)说明书进行BrdU流式染色。染色完成后用BD Canto流式细胞仪进行细胞增殖和细胞周期分析。
1.11 统计学分析采用SPSS 16.0统计软件。细胞实验均重复3次。两组间比较根据数据类型使用Student’s t检验。P<0.05为具有统计学意义。
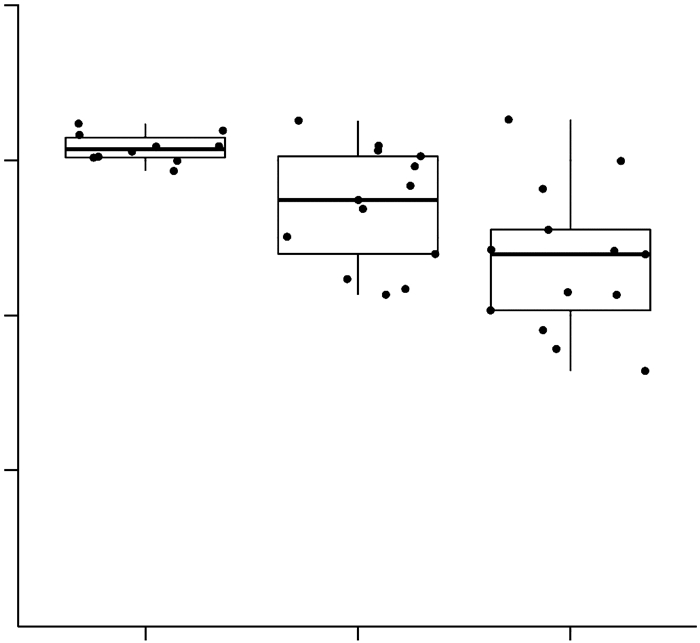
2 结果 2.1 EHHADH-AS1在健康人和早期肝细胞癌、晚期肝细胞癌患者组织中的表达对EHHADH-AS1在健康人、早期肝癌(eHCC)和晚期肝癌(aHCC)患者组织中的表达进行比较,发现EHHADH-AS1在肝癌组织中表达显著降低,并且随着肝癌进程加深而逐渐下调(图 1)。
|
| 两组间差异采用Student’s t检验,a:P<0.01,与健康人比较 图 1 EHHADH-AS1在健康人和早期肝癌(eHCC)、晚期肝癌(aHCC)患者组织中的表达 |
2.2 EHHADH-AS1与编码基因(mRNA)的共表达网络构建及信号通路富集分析
为了探究EHHADH-AS1在肝癌进程中的作用,构建EHHADH-AS1与mRNA的共表达网络。结果发现共有343个编码基因与EHHADH-AS1具有共表达关系(图 2)。

|
| 图形代表基因的种类,其中圆形代表lncRNA,方形代表mRNA;颜色深浅表示基因在肝癌组织中相比于正常组织的表达改变情况,其中绿色表示表达下调,红色表示表达上调;连线表示lncRNA和mRNA共表达情况,虚线表示lncRNA和mRNA负相关,实线表示lncRNA和mRNA正相关 图 2 EHHADH-AS1与mRNA的共表达网络 |
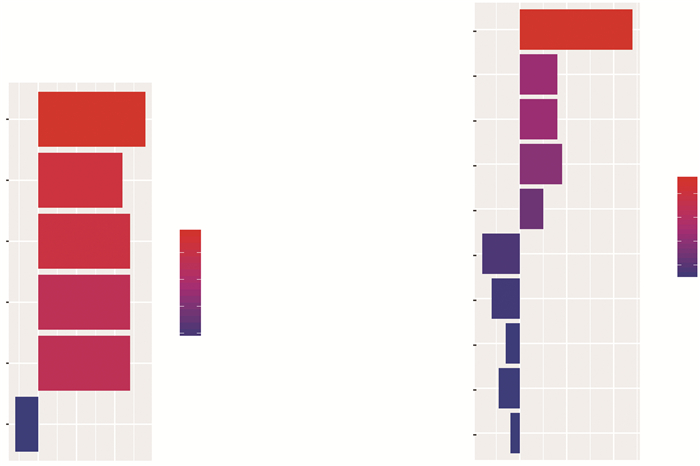
对与EHHADH-AS1具有共表达关系的编码基因进行KEGG分析,发现与EHHADH-AS1正相关的基因主要富集在药物代谢(drug metabolism)、脂肪酸代谢(fatty acid metabolism)、视黄醇代谢(retinol metabolism)、补体和血凝级联(complement and coagulation cascades)、氧化物酶体增殖物激活受体信号通路(PPAR signaling pathway)等KEGG信号通路上,提示与EHHADH-AS1正相关的基因主要参与细胞能量代谢;而与EHHADH-AS1负相关的编码基因主要富集在碱基切除修复(base excision repair)KEGG信号通路上(图 3A)。
|
| 纵坐标表示富集的信号通路,横坐标表示对应信号通路富集的基因数量;颜色表示-log10 (P值);正相关的信号通路其横坐标为正数,而负相关的信号通路其横坐标为负数 图 3 与EHHADH-AS1共表达基因的KEGG(A)和GO(B)信号通路富集分析 |
对与EHHADH-AS1具有共表达关系的编码基因(mRNA)进行GO富集分析,发现与EHHADH-AS1正相关的基因富集在氧化还原(oxidation reduction)、羧酸分解过程(carboxylic acid catabolic process)、有机酸分解过程(organic acid catabolic process)、脂肪酸代谢过程(fatty acid metabolic process)和氧结合(oxygen binding)等生物学过程上,提示与EHHADH-AS1正相关的基因主要参与细胞代谢和ATP生成(图 3B)。而与EHHADH-AS1负相关的编码基因主要富集在细胞周期(cell cycle)、细胞周期过程(cell cycle process)、微管细胞骨架组织(microtubule cytoskeleton organization)、有丝分裂(mitotic cell cycle)、纺锤结构(spindle organization)等生物学过程上(图 3B)。这些编码基因在肝细胞癌组织中是高表达的,且和细胞增殖分裂有关,提示EHHADH-AS1可能在调控肝细胞癌细胞增殖和细胞周期中具有抑制作用。
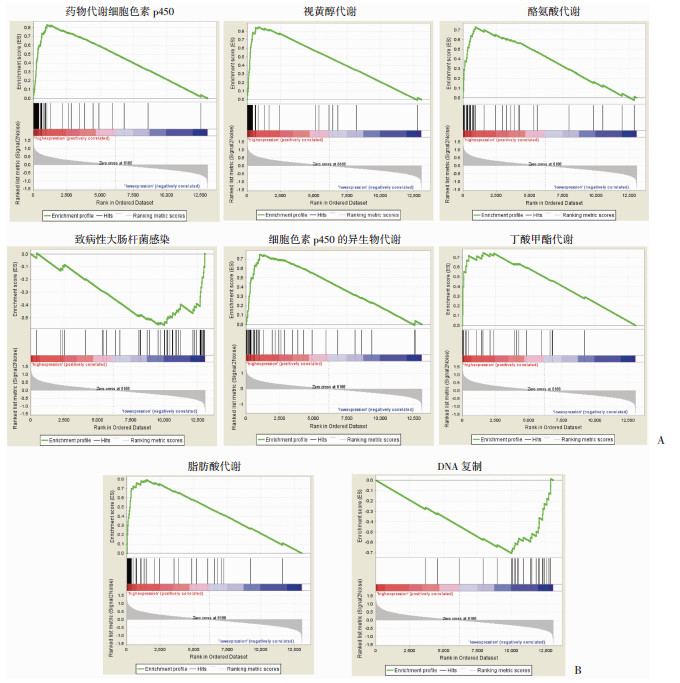
2.3 肝细胞癌组织中EHHADH-AS1高表达与低表达组的GSEA KEGG信号通路富集分析根据肝癌患者的组织样本中EHHADH-AS1的表达水平,将肝癌组织样本分为EHHADH-AS1高表达组和低表达组,并进行GSEA KEGG比较分析。结果发现药物代谢细胞色素P450(drug metabolism cytochrome P450)、酪氨酸代谢(tyrosine metabolism)、视黄醇代谢、脂肪酸代谢、细胞色素P450外源物代谢(metabolism of xenobiotics by cytochrome p450)、PPAR等信号通路在EHHADH-AS1高表达组中显著上调;而大肠杆菌感染(Escherichia coli infection)和DNA复制(DNA replication信号通路)在EHHADH-AS1低表达组中上调(图 4)。这与上述EHHADH-AS1共表达基因的信号通路富集分析的发现基本一致,进一步提示EHHADH-AS1的高表达与药物代谢和细胞代谢有关,而EHHADH-AS1低表达与DNA复制改变、细胞周期、细胞增殖等细胞事件关系密切。
|
| A:EHHADH-AS1高表达组中上调的KEGG通路(显示前6位最显著的KEGG信号通路);B:EHHADH-AS1低表达组中上调的KEGG通路(仅富集2个信号通路) Kolmogorov-Smirnov检验,P<0.05 图 4 肝细胞癌组织中EHHADH-AS1高、低表达组的GSEA分析 |
2.4 EHHADH-AS1 siRNA对HepG2细胞活力、细胞增殖和细胞周期的影响
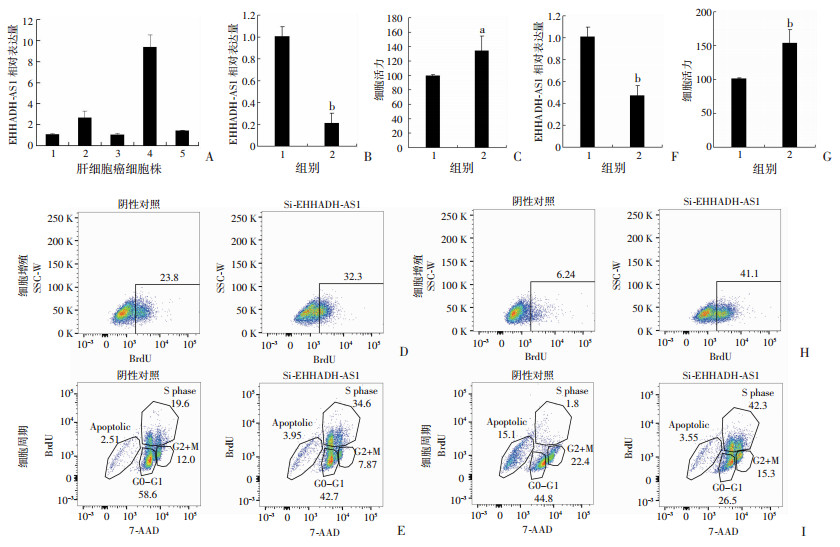
为进一步证实EHHADH-AS1在肝细胞癌发生、发展中的作用,首先分析了不同肝细胞癌细胞株中EHHADH-AS1的表达。结果发现HepG2细胞中EHHADH-AS1表达相对较低,而Huh7细胞EHHADH-AS1表达相对最高(图 5A)。分别选择HepG2和Huh7细胞进行siRNA转染,敲低EHHADH-AS1在HepG2和Huh7细胞中的表达。结果发现HepG2细胞经过EHHADH-AS1 siRNA转染后,EHHADH-AS1的表达均显著降低(P<0.01,图 5B),与本研究者前期文献报道的结果一致[14]。MTT实验结果表明HepG2经过EHHADH-AS1 siRNA转染24 h后,其细胞活力也均显著增加(图 5C)。BrdU染色实验表明HepG2细胞在转染EHHADH-AS1 siRNA 24 h后,BrdU阳性细胞的比例明显增加(图 5D),且S期的细胞比例也显著增加(图 5E)。在Huh7细胞中敲低EHHADH-AS1表达也有类似的发现(图 5F~I),这表明EHHADH-AS1可显著抑制肝癌细胞的增殖能力。综合上述结果,可见EHHADH-AS1表达相对较高的Huh7细胞经EHHADH-AS1 siRNA转染后其细胞活力、细胞增殖和细胞周期改变的倍率相对更大。
|
| A:不同肝细胞癌细胞株EHHADH-AS1的表达1:HepG2;2:SM-7721;3:PLC/PRF/5;4:Huh7;5:Hep3B;B:荧光定量PCR检测HepG2细胞EHHADH-AS1 siRNA转染24 h后EHHADH-AS1的表达;C:MTT检测HepG2细胞EHHADH-AS1 siRNA转染24 h后的细胞活力;D、E:流式细胞仪检测HepG2细胞增殖(BrdU+细胞比例)、细胞周期;F:荧光定量PCR检测Huh7细胞EHHADH-AS1 siRNA转染24 h后EHHADH-AS1的表达;G:MTT检测Huh7细胞EHHADH-AS1 siRNA转染24 h后的细胞活力;H、I:流式细胞仪检测Huh7细胞增殖(BrdU+细胞比例)、细胞周期1:阴性对照;2:Si-EHHADH-AS1,HepG2或Huh7细胞转染EHHADH-AS1 siRNA序列;Student’s t检验,a:P<0.05,b:P<0.01,与阴性对照比较 图 5 EHHADH-AS1 siRNA转染后对HepG2和Huh7细胞EHHADH-AS1表达、细胞活力、细胞增殖及细胞周期的影响 |
2.5 EHHADH-AS1 siRNA对HepG2细胞周期相关基因的影响
有研究表明lncRNA可与某些转录因子结合,从而影响该转录因子对其调控靶基因的结合,从而抑制靶基因的转录;如lncRNA HOXA11-AS通过与转录因子WDR5、EZH2结合从而抑制EZH2对P21蛋白的转录[21]。鉴于EHHAD-AS1对细胞周期的抑制作用,分析细胞周期信号通路中与EHHAD-AS1具有共表达关系、且相对正常组织改变倍数在3倍以上的基因,共发现5个基因,分别是CHAF1A、STMN1、PSRC1、PTTG1、AURKA。转录因子结合靶基因的预测分析发现有E2F1、E2F3、E2F4、SMAD3、TP53等5个转录因子既可结合至上述5个基因,又可以与EHHAD-AS1结合(图 6A)。鉴于此,本研究进一步通过定量PCR验证敲低EHHAD-AS1表达后是否影响上述细胞周期相关基因的表达。结果发现在HepG2细胞里转染EHHAD-AS1 siRNA后,细胞周期相关基因CHAF1A、STMN1和PTTG1的mRNA水平进一步升高(图 6B)。
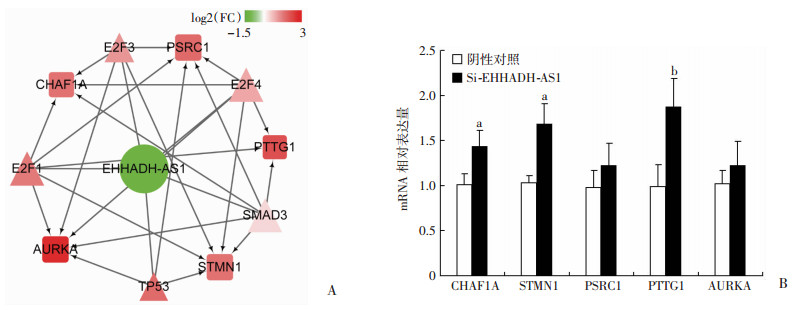
|
| A:采用TFBSTools包预测可同时与细胞周期相关基因启动子结合和与EHHADH-AS1结合的转录因子;B:荧光定量PCR检测HepG2细胞EHHADH-AS1 siRNA转染24 h后CHAF1A、STMN1、PSRC1、PTTG1、AURKA mRNA的表达两组间差异采用Student’s t检验,a: P<0.05,b:P<0.01,与阴性对照比较 图 6 EHHADH-AS1 siRNA转染后对HepG2细胞细胞周期相关基因表达的影响 |
3 讨论
HBV在肝细胞的癌变、侵袭及转移中具有重要的促进作用,而HBV相关肝细胞癌在我国具有较高的发病比例[22]。近期研究发现lncRNA在HBV相关肝细胞癌中也具有一定的作用,如lncRNA DANCR通过促进癌细胞的干细胞特性在增强肝细胞癌的恶性程度中发挥着重要作用[12]。我们前期通过深入挖掘公开发表的HBV相关肝细胞癌的数据,阐明了HBV相关肝细胞癌发生、发展过程中的lncRNA谱系改变特征,并发现了数个与HBV相关肝细胞癌患者生存预后密切相关的关键lncRNA[13]。在此基础上,我们还构建了内源性竞争性RNA(competing endogenous RNA,ceRNA)网络,初步探讨了这些lncRNA参与调控HBV相关肝细胞癌发生、发展的可能机制[14]。
本研究进一步研究了lncRNA EHHADH-AS1在HBV相关肝细胞癌发生、发展的可能作用和机制。首先EHHADH-AS1在HBV相关肝细胞癌肿瘤组织中的表达相较于正常组织明显降低,且在晚期肝细胞癌中更甚。而后续的实验研究证实:通过siRNA方法敲低肝癌细胞株HepG2和Huh7细胞中EHHADH-AS1的表达后,HepG2和Huh7细胞的细胞活性和增殖能力明显增加。这进一步说明EHHADH-AS1是一个具有抑制肝细胞癌细胞活性和增殖作用的分子。
为进一步探讨EHHADH-AS1参与调控HBV相关肝细胞癌发生、发展的可能作用机制,本研究进行了一系列的富集分析。通过分析与EHHADH-AS1有共表达关系的编码基因富集的信号通路发现:与EHHADH-AS1正相关的编码基因主要参与药物代谢、脂肪酸代谢、视黄醇代谢、补体和血凝级联、氧化物酶体增殖物激活受体等KEGG信号通路,同时GO生物学过程分析也发现了类似的信号通路。而与EHHADH-AS1负相关的编码基因主要富集在碱基切除修复、细胞周期、细胞周期过程、微管细胞骨架组织、有丝分裂、纺锤结构等信号通路上。这些证据强烈提示EHHADH-AS1可能正向调节肝细胞代谢和ATP生成,参与一些药物的代谢和外排,而负向调控肝癌细胞的癌变和增殖,抑制肝癌细胞的细胞周期进展。
进一步通过GSEA分析发现在HBV相关肝细胞癌组织中EHHADH-AS1表达相对较高的样本其药物代谢细胞色素P450、酪氨酸代谢、视黄醇代谢、脂肪酸代谢、细胞色素P450外源物代谢、PPAR等信号通路的基因表达也是上调的。而HBV相关肝细胞癌组织中EHHADH-AS1表达相对较低的样本中其DNA复制等信号通路的基因表达明显增加。这与上述分析的与EHHADH-AS1有共表达关系的编码基因富集的信号通路基本一致,这提示EHHADH-AS1表达对HBV相关肝细胞癌的发生、发展有着重要影响。后续实验利用siRNA方法敲低肝癌细胞株HepG2和Huh7细胞中EHHADH-AS1的表达后,其细胞活性和增殖均明显增加,且S期的细胞比例也显著增加。上述证据强烈表明EHHADH-AS1通过抑制肝癌细胞的DNA复制和细胞周期,从而实现抑制肿瘤细胞增殖和活力的作用。
本研究发现与EHHADH-AS1具有共表达关系的参与细胞周期调控的相关基因如CHAF1A、STMN1和PTTG1在HepG2细胞转染EHHADH-AS1 siRNA后显著升高。CHAF1A属于染色体组装复合体(CHAF1,Chromatin assembly factor Ⅰ)关键因子之一,是将组蛋白八聚体和正在复制的DNA组装的关键复合体。有研究表明CHAF1A通过参与调控细胞周期进而影响多种恶性肿瘤的发生、发展[23-24]。而在肝癌中发现CHAF1A与肿瘤的淋巴结转移、肿瘤的数量和分化程度呈正相关,且高表达CHAF1A的患者预后较差[25]。STMN1在肝癌组织中高表达,促进肝癌细胞的增殖和生长[26]。PTTG1在肝癌组织高水平表达,与肝细胞癌的临床分期呈正相关[27]。有研究发现HBV编码的蛋白HBX通过抑制PTTG1的泛素化而抑制PTTG1的降解[28]。这些证据强烈表明EHHADH-AS1通过调控多个细胞周期关键基因而实现其对肝癌细胞细胞周期的抑制作用。
近期有研究表明在肝癌中STMN1的表达是受E2F1的正向转录调控的[29],并且也有研究表明在垂体瘤中E2F1过表达可以促进PTTG1的表达[30],这与我们预测的结果一致。E2F1是E2F转录因子家族的重要成员,参与调控多种通路,包括细胞凋亡和增殖。E2F1的过度表达会加快细胞由G1期向S期过渡,导致细胞过度增殖,促进肿瘤形成[31],在肝细胞癌的发生、发展中具有关键作用[32]。结合本研究实验证据,提示EHHADH-AS1可能通过与E2F1结合,而影响E2F1对细胞周期调控的关键基因如STMN1、PTTG1的启动子区域的结合,进而抑制这些基因的表达,从而实现EHHADH-AS1对细胞周期的抑制作用。但是这一猜想仍需进一步的实验证实。
综上所述,本研究深入探讨了EHHADH-AS1在HBV相关性肝细胞癌的发生、发展中的可能作用和分子机制,并通过实验研究进一步证实EHHADH-AS1通过抑制肝癌细胞的细胞周期,从而达到抑制肿瘤细胞增殖和活力的作用。因此,进一步深入研究EHHADH-AS1在HBV相关肝细胞癌的发生、发展中的作用机制,有望为HBV相关肝细胞癌的诊断、治疗和预后提供新的分子和潜在的新靶标。
| [1] | TORRE L A, SIEGEL R L, WARD E M, et al. Global cancer incidence and mortality rates and trends: an update[J]. Cancer Epidemiol Biomarkers Prev, 2016, 25(1): 16–27. DOI:10.1158/1055-9965.EPI-15-0578 |
| [2] | PARKIN D M. The global health burden of infection-associated cancers in the year 2002[J]. Int J Cancer, 2006, 118(12): 3030–3044. DOI:10.1002/ijc.21731 |
| [3] | OKAZAKI Y, FURUNO M, KASUKAWA T, et al. Analysis of the mouse transcriptome based on functional annotation of 60, 770 full-length cDNAs[J]. Nature, 2002, 420(6915): 563–573. DOI:10.1038/nature01266 |
| [4] | GUTTMAN M, RINN J L. Modular regulatory principles of large non-coding RNAs[J]. Nature, 2012, 482(7385): 339–346. DOI:10.1038/nature10887 |
| [5] | TRIPATHI V, SHEN Z, CHAKRABORTY A, et al. Long noncoding RNA MALAT1 controls cell cycle progression by regulating the expression of oncogenic transcription factor B-MYB[J]. PLoS Genet, 2013, 9(3): e1003368. DOI:10.1371/journal.pgen.1003368 |
| [6] | BATISTA P J, CHANG H Y. Long noncoding RNAs: cellular address codes in development and disease[J]. Cell, 2013, 152(6): 1298–1307. DOI:10.1016/j.cell.2013.02.012 |
| [7] | CHAUHAN R, LAHIRI N. Tissue- and serum-associated biomarkers of hepatocellular carcinoma[J]. Biomark Cancer, 2016, 8(Suppl 1): 37–55. DOI:10.4137/BIC.S34413 |
| [8] | PARASRAMKA M A, MAJI S, MATSUDA A, et al. Long non-coding RNAs as novel targets for therapy in hepatocellular carcinoma[J]. Pharmacol Ther, 2016, 161: 67–78. DOI:10.1016/j.pharmthera.2016.03.004 |
| [9] | HUANG J L, REN T Y, CAO S W, et al. HBx-related long non-coding RNA DBH-AS1 promotes cell proliferation and survival by activating MAPK signaling in hepatocellular carcinoma[J]. Oncotarget, 2015, 6(32): 33791–33804. DOI:10.18632/oncotarget.5667 |
| [10] | DU Y, KONG G, YOU X, et al. Elevation of highly up-regulated in liver cancer (HULC) by hepatitis B virus X protein promotes hepatoma cell proliferation via down-regulating p18[J]. J Biol Chem, 2012, 287(31): 26302–26311. DOI:10.1074/jbc.M112.342113 |
| [11] | HUANG J F, GUO Y J, ZHAO C X, et al. Hepatitis B virus X protein (HBx)-related long noncoding RNA (lncRNA) down-regulated expression by HBx (Dreh) inhibits hepatocellular carcinoma metastasis by targeting the intermediate filament protein vimentin[J]. Hepatology, 2013, 57(5): 1882–1892. DOI:10.1002/hep.26195 |
| [12] | YUAN S X, WANG J, YANG F, et al. Long noncoding RNA DANCR increases stemness features of hepatocellular carcinoma by derepression of CTNNB1[J]. Hepatology, 2016, 63(2): 499–511. DOI:10.1002/hep.27893 |
| [13] |
涂晓丽, 文荃, 闫军. HBV+肝细胞癌患者lncRNA-mRNA共表达网络分析及作用[J].
第三军医大学学报, 2018, 40(1): 37–44.
TU X L, WEN Q, YAN J. Profiles and functions of lncRNA-mRNA co-expression network in HBV+ hepatocellular carcinoma patients[J]. J Third Med Univ, 2018, 40(1): 37–44. DOI:10.16016/j.1000-5404.201708015 |
| [14] | WEN Q, YANG Y, CHEN X, et al. Competing endogenous RNA screening based on lncRNA-mRNA co-expression profile in HBV-associated hepatocarcinogenesis[J]. J Tradit Chin Med, 2017, 37(4): 510–521. DOI:10.1016/s0254-6272(17)60108-0 |
| [15] | LIU N, LIU X, ZHOU N, et al. Gene expression profiling and bioinformatics analysis of gastric carcinoma[J]. Exp Mol Pathol, 2014, 96(3): 361–366. DOI:10.1016/j.yexmp.2014.02.007 |
| [16] | HUANG D A W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44–57. DOI:10.1038/nprot.2008.211 |
| [17] | YU G, WANG L G, HAN Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284–287. DOI:10.1089/omi.2011.0118 |
| [18] | TAN G, LENHARD B. TFBSTools: an R/bioconductor package for transcription factor binding site analysis[J]. Bioinformatics, 2016, 32(10): 1555–1556. DOI:10.1093/bioinformatics/btw024 |
| [19] | KHAN A, FORNES O, STIGLIANI A, et al. JASPAR 2018: update of the open-access database of transcription factor binding profiles and its web framework[J]. Nucleic Acids Res, 2018, 46(D1): D1284. DOI:10.1093/nar/gkx1188 |
| [20] | SUN X, HAN Q, LUO H, et al. Profiling analysis of long non-coding RNAs in early postnatal mouse hearts[J]. Sci Rep, 2017, 7: 43485. DOI:10.1038/srep43485 |
| [21] | LIU Z, CHEN Z, FAN R, et al. Over-expressed long noncoding RNA HOXA11-AS promotes cell cycle progression and metastasis in gastric cancer[J]. Mol Cancer, 2017, 16(1): 82. DOI:10.1186/s12943-017-0651-6 |
| [22] | LUPBERGER J, HILDT E. Hepatitis B virus-induced oncogenesis[J]. World J Gastroenterol, 2007, 13(1): 74–81. DOI:10.3748/wjg.v13.i1.74 |
| [23] | LIU T, WEI J, JIANG C, et al. CHAF1A, the largest subunit of the chromatin assembly factor 1 complex, regulates the growth of H1299 human non-small cell lung cancer cells by inducing G0/G1 cell cycle arrest[J]. Exp Ther Med, 2017, 14(5): 4681–4686. DOI:10.3892/etm.2017.5201 |
| [24] | MJELLE R, HEGRE S A, AAS P A, et al. Cell cycle regulation of human DNA repair and chromatin remodeling genes[J]. DNA Repair (Amst), 2015, 30: 53–67. DOI:10.1016/j.dnarep.2015.03.007 |
| [25] | XU M, JIA Y, LIU Z, et al. Chromatin assembly factor 1, subunit A (P150) facilitates cell proliferation in human hepatocellular carcinoma[J]. Onco Targets Ther, 2016, 9: 4023–4035. DOI:10.2147/OTT.S107050 |
| [26] | TSENG Y H, HUANG Y H, LIN T K, et al. Thyroid hormone suppresses expression of stathmin and associated tumor growth in hepatocellular carcinoma[J]. Sci Rep, 2016, 6: 38756. DOI:10.1038/srep38756 |
| [27] | SU M C, HSU H C, LIU Y J, et al. Overexpression of pituitary tumor-transforming gene-1 in hepatocellular carcinoma[J]. Hepatogastroenterology, 2006, 53(68): 262–265. |
| [28] | MOLINA-JIMENEZ F, BENEDICTO I, MURATA M, et al. Expression of pituitary tumor-transforming gene 1 (PTTG1)/ securin in hepatitis B virus (HBV)-associated liver diseases: evidence for an HBV X protein-mediated inhibition of PTTG1 ubiquitination and degradation[J]. Hepatology, 2010, 51(3): 777–787. DOI:10.1002/hep.23468 |
| [29] | CHEN Y L, UEN Y H, LI C F, et al. The E2F transcription factor 1 transactives stathmin 1 in hepatocellular carcinoma[J]. Ann Surg Oncol, 2013, 20(12): 4041–4054. DOI:10.1245/s10434-012-2519-8 |
| [30] | ZHOU C, WAWROWSKY K, BANNYKH S, et al. E2F1 induces pituitary tumor transforming gene (PTTG1) expression in human pituitary tumors[J]. Mol Endocrinol, 2009, 23(12): 2000–2012. DOI:10.1210/me.2009-0161 |
| [31] | STANELLE J, STIEWE T, THESELING C C, et al. Gene expression changes in response to E2F1 activation[J]. Nucleic Acids Res, 2002, 30(8): 1859–1867. DOI:10.1093/nar/30.8.1859 |
| [32] | FARRA R, GRASSI G, TONON F, et al. The role of the transcription factor E2F1 in hepatocellular carcinoma[J]. Curr Drug Deliv, 2017, 14(2): 272–281. DOI:10.2174/1567201813666160527141742 |



