2. 400016 重庆,重庆医科大学:分子医学与肿瘤研究中心
2. Research Center for Molecule Medicine and Tumor, Chongqing Medical University, Chongqing, 400016, China
囊型包虫病(cystic echinococcosis,CE)又称为棘球蚴病,由细粒棘球绦虫的幼虫棘球蚴(hydatid)寄生于人体所致。有关棘球蚴病及棘球蚴的生物学特性研究已有不少,但棘球蚴生长过程中棘球蚴液的来源及生成机制一直是个谜。水通道蛋白(aquaporins,AQPs)是广泛存在于原核和真核生物细胞膜上专门运输水的特异蛋白孔道,可选择性介导水和溶质分子的跨膜转运。基因组学研究表明,细粒棘球绦虫也存在编码AQPs的基因[1]。本课题组认为棘球蚴液的形成或积累可能与细粒棘球绦虫水通道蛋白(aquaporins of Echinococcus granulosus,EgAQPs)有关,研究棘球蚴液形成或积累过程中囊壁生发层细胞EgAQPs基因的转录表达水平,可为探讨EgAQPs基因转录表达水平变化与棘球蚴液形成或积累的关系提供依据。
实时荧光定量PCR(real-time quantitative PCR,RT-qPCR)是基因表达研究最常用的方法,具有准确度高、灵敏度好、高通量、只需要少量的RNA等优点[2]。利用RT-qPCR进行基因表达研究时,常需要使用内参基因对结果进行校正和标准化,以消除RNA起始量、反转录效率、扩增效率等因素的差异对基因表达水平的影响,从而真实反映基因表达情况。
内参基因是指在各种细胞和组织中相对表达恒定的一类基因。过去普遍认为,维持细胞正常生命代谢所必需的管家基因在不同组织及不同实验条件下都具有稳定的表达模式,是理想的内参基因。但越来越多的研究表明,在一种实验条件下表达稳定的内参基因不一定适用于另一种实验条件[3-7],研究者必须结合具体的实验条件和样品类型来选择合适的内参基因。
细粒棘球绦虫基因组的成功测序促进了目的基因表达模式的研究,然而对细粒棘球绦虫内参基因稳定性评价的相关研究尚少见报道。本研究选择棘球绦虫研究中较常用的ND1[8-9]、ATP6[8]、elp[10-12]、actin[13-14]和ActⅡ[14-15]5种内参基因,通过geNorm软件筛选出原头蚴成囊过程中稳定表达的内参基因,并在此基础上检测EgAQPs基因在原头蚴发育为棘球蚴过程中的转录表达水平,以探讨棘球蚴囊壁生发层细胞EgAQPs基因表达水平变化与棘球蚴液形成或积累的关系。
1 材料与方法 1.1 寄生虫标本和实验动物感染细粒棘球蚴的羊肝采集自新疆某屠宰场,通过冷链运至本实验室。75只健康雌性BALB/c小鼠,6~8周龄、体质量18~22 g,由重庆医科大学实验动物中心提供。动物的饲养及实验相关程序均遵循实验动物管理和保护的相关规定。
1.2 原头蚴的收集分别收集囊液中的原头蚴和贴于内囊壁上的原头蚴,用含有1%青-链霉素的无菌PBS反复清洗至能清晰观察到原头蚴自然下沉为止。清洗后的原头蚴悬液经0.4%台盼蓝染色后显微镜下观察其活力,活力>85%时可用于体外培养及棘球蚴小鼠模型的建立。
1.3 原头蚴的体外培养及收集向每个无菌细胞培养瓶中移入20 000个原头蚴并加入适量含10%胎牛血清的RPMI1640培养基,于37 ℃,5%CO2培养箱中培养。收集培养第3、15、25、35天的原头蚴,取0.5 mL培养物经0.4%台盼蓝染色,光学显微镜下拍照,倒置显微镜下测量囊大小(囊大小= 100 μL悬液中所有囊直径的平均值,计数3张玻片取平均值)并计算成囊率(成囊率=100 μL悬液中囊的数量/100 μL悬液中原头蚴与囊的数量之和×100%,计数3张玻片取平均值),剩下的培养物用适量无菌PBS清洗后装入无酶冻存管中,于液氮中保存备用。
1.4 模型小鼠体内的继发棘球蚴收集每只小鼠腹腔注射原头蚴悬液0.2 mL(约含原头蚴2 000个),于感染后第3、4、5、6、7个月各取15只小鼠进行解剖,收集继发棘球蚴包囊并测量大小,用适量无菌PBS清洗后装入无酶冻存管,在液氮中速冻后转入-80 ℃超低温冰箱保存备用。
1.5 总RNA的提取及cDNA第一链的合成用TRIzol(TaKaRa)提取原头蚴和继发棘球蚴囊壁组织总RNA,经1%琼脂糖凝胶电泳检测总RNA的完整性,并用Nano Drop ND-2000分光光度计测定浓度及D(260)/D(280)值。以各样本500 ng总RNA为模板,利用PrimeScriptTM RT reagent kit with gDNA Eraser(TaKaRa)反转录试剂盒合成cDNA第一链,-80 ℃超低温冰箱保存备用。
1.6 内参基因及EgAQPs基因的RT-qPCR引物合成根据细粒棘球绦虫基因组序列信息,采用Primer 3.0软件分别设计5个候选内参基因以及7个EgAQPs基因的RT-qPCR引物,引物序列及相关信息见表 1。引物均由上海生工生物工程有限公司合成。
| 基因名称 | 登录号 | 引物序列(5'→3') | 预期大小/bp |
| ActⅡ | AF003749 | 上游:ATGGTTGGTATGGGACGG 下游:TTCGTCACAATACCGTGCTC |
118 |
| ND1 | AF408688 | 上游:GCCGCCAGAACATCTAGGTA 下游:AGCACATCGAACCGACCTTA |
87 |
| elp | AB159143 | 上游:AAAGCTGAAGGAGGTGCGTA 下游:GTGCCGAATTAGCTTGGTGT |
124 |
| ATP6 | JF690977 | 上游:GTGTGCTTGGCCGAGTCTAT 下游:CACAAATTACCCAATGCCACT |
113 |
| actin | L07775 | 上游:AATTGGGACGACATGGAGAA 下游:GCGATTGGCTTTAGGATTGA |
117 |
| EgAQP4 | EgrG_000 125550 | 上游:CGATATCCGGAGGCAGTATG 下游:CAAGTCGTCAGCTGCATCCG |
196 |
| EgAQP9 | EgrG_001 190800 | 上游:CAGCTATTAATCGGGCTCCA 下游:CGGTCAAATCTCCTTTCAGG |
118 |
| EgAQP | EgrG_000 125500 | 上游:GGGTGGGGGGCTCTACGTT 下游:CTCATTGCTTCCAGGCGGT |
199 |
| EgAQP1 | EgrG_000 125400 | 上游:CTGTCTGTTAAGTCCGCC 下游:GCTTCTGCGTTGCTCACT |
162 |
| EgAQP3 | EGR_03137 | 上游:TGAAATCAGCCTCGGTTGGG 下游:CTGACTTCTTGACGAGACGA |
171 |
| EgAQP FA-CHIP | Egr_04161 | 上游:CCCCTGCTCTCTCTTGACCAC 下游:TCCGAACGACAACAACCCTCT |
130 |
| EgAQP AnG | Egr_04162 | 上游:TCTGGCTCTTAGTACGGATTTTTG 下游:TGATATGCGCTCCACTGGTG |
177 |
1.7 候选内参基因引物的特异性评价
以体外培养第3天原头蚴的cDNA为模板,通过RT-PCR扩增5个候选内参基因。将5个候选内参基因的PCR产物连接到pMD-18-T载体上,送至宝生物工程(大连)有限公司测序。
1.8 候选内参基因扩增效率评价以体外培养第3天原头蚴的cDNA为模板,用SYBR Premix Ex TaqTM Ⅱ试剂盒(TaKaRa)对各候选内参基因的引物进行RT-qPCR,每个样本重复3次。RT-qPCR在CFX-96 Real Time PCR仪(Bio-Rad)上进行,并绘制熔解曲线。将cDNA模板按1 :3梯度稀释成8个浓度梯度进行标准曲线分析,每个样本重复3次,对照组用RNA free water代替模板。
1.9 内参基因稳定性评价geNorm软件[16]通过计算各候选内参基因转录表达稳定性的平均值M,根据M值对各候选内参基因转录表达稳定性进行排序,M值越大,稳定性越差,M值越小,稳定性越好。geNorm程序默认的M阈值为1.5。此外,geNorm程序还可以通过候选内参基因标准化因子的配对差异分析(Vn/n+1)来判断所需内参基因的最适数目,geNorm程序默认的Vn/n+1阈值为0.15。
1.10 原头蚴成囊过程中EgAQPs基因的转录表达水平以geNorm程序推荐基因作为内参,通过RT-qPCR方法检测原头蚴成囊过程中EgAQPs基因的转录表达水平,并采用2-△Ct法计算原头蚴成囊过程中EgAQPs基因的相对表达量。
1.11 统计学分析数据以x±s表示。采用SPSS 18.0统计软件进行单因素方差分析,并通过SNK-q检验进行两两比较,检验水准:α=0.05。
2 结果 2.1 体外培养的原头蚴成囊效果原头蚴体外培养至第3天可见大多数原头蚴顶突外翻、大小均匀、头节呈梨形,未看到成囊现象;培养至第15天可见大部分原头蚴顶突和吸盘翻出,原头蚴成囊率为20.10%,囊大小为0.03 mm;培养至第25天可见原头蚴体积明显增大,外形呈透明的气球状,原头蚴成囊率为41.13%,囊大小为0.17 mm;培养至第35天时,原头蚴成囊率为100%,囊大小为0.17 mm。培养不同时间原头蚴成囊状态见图 1。
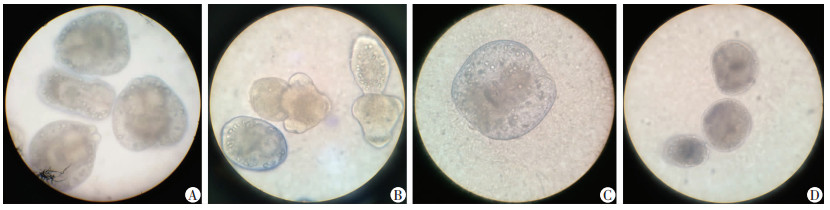
|
| A:3 d;B:15 d;C:25 d;D:35 d 图 1 体外培养不同时间原头蚴成囊状态显微镜观察(×40) |
2.2 模型小鼠体内原头蚴囊化为棘球蚴的效果
小鼠腹腔内可见数量不等、游离或黏附在肝、脾等器官表面的白色棘球蚴包囊,有些棘球蚴包囊里充满透亮的棘球蚴液(图 2)。感染后第3个月小鼠感染率为86.67%(13/15),感染后第4、5、6、7个月小鼠感染率均为100%,平均感染率为97.33%(73/75)。随着感染时间的延长,小鼠腹部增大明显,棘球蚴包囊直径3个月时(3.82±2.17)mm,4个月(7.08±3.73)mm,5个月(10.48±5.34)mm,6个月(14.19±7.42)mm,7个月(15.64±9.36)mm。本研究成功建立棘球蚴小鼠模型,可用于后续实验。
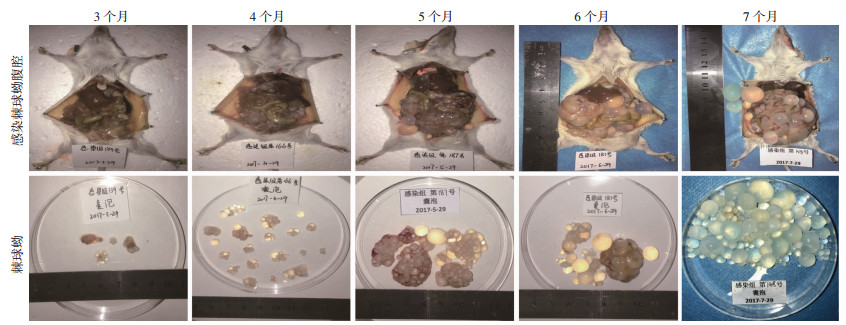
|
| 图 2 不同发育时间小鼠腹腔内棘球蚴大小 |
2.3 总RNA的完整性及纯度
在进行RT-qPCR时,高完整性和高纯度的RNA是实验成功的关键,体外培养的原头蚴总RNA及继发棘球蚴囊壁组织总RNA经1%琼脂糖凝胶电泳检测,均可以看到28S和18S两个条带且条带明亮,说明RNA完整。经浓度测定,各样本RNA的D(260)/D(280)值在1.8~2.1之间,表明RNA纯度较高,可用于后续的RT-qPCR实验。
2.4 候选内参基因引物的特异性、扩增效率评价5个候选内参基因的PCR产物经2%琼脂糖凝胶电泳检测,结果显示,5个内参基因的扩增产物均呈单一条带(图 3),产物的理论大小与电泳结果一致,说明引物特异性较好。将5个内参基因的PCR产物连接到pMD-18-T载体上进行测序,结果表明各候选内参基因的序列与GenBank中已有的序列一致,符合进一步实验的要求。
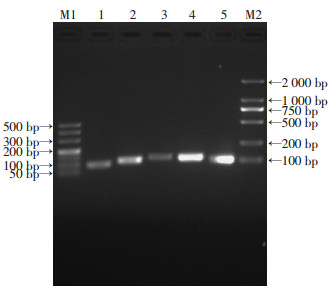
|
| M1:DNA Ladder 500;1~5:分别为ND1、ATP6、elp、actin、ActⅡ基因的PCR产物;M2:DNA Ladder 2000 图 3 候选内参基因PCR扩增产物琼脂糖凝胶电泳结果 |
绘制5个候选内参基因的熔解曲线,结果表明,5个候选内参基因的熔解曲线均呈单一峰型,且解链温度均高于80 ℃,说明引物特异性好。绘制5个内参基因的标准曲线,结果表明,5个候选内参基因的扩增效率(E)介于96.1%~104.2%之间,线性相关系数(r2)变化范围为0.989~0.997,符合RT-qPCR的要求。另外,阴性对照无扩增信号,说明实验体系无污染,实验样品可用于后续研究。
2.5 内参基因的稳定性分析5个候选内参基因在原头蚴成囊样本中的M值均小于geNorm软件所推荐的阈值1.5,各候选内参基因基本符合要求,但在规定的范围内数值变化较大,说明5个内参基因在原头蚴成囊样本中表达不一致。
以体外培养原头蚴为样本,通过geNorm软件分析内参基因elp、ATP6、ND1、actin、ActⅡ的M值分别为1.452、1.313、1.203、0.064、0.064。5个候选内参基因转录表达稳定性由高到低依次为actin=ActⅡ>ND1>ATP6>elp。对体外培养原头蚴样本中内参基因标准化因子的配对差异分析,结果表明Vn/n+1均 < 0.15,可选择2个内参基因的组合,即actin+ActⅡ。
以继发棘球蚴囊壁组织为样本,通过geNorm软件分析内参基因elp、ActⅡ、actin、ATP6、ND1的M值分别为1.366、0.925、0.599、0.449、0.449。5个候选内参基因转录表达稳定性由高到低依次为ATP6=ND1>actin>ActⅡ>elp。对继发棘球蚴囊壁组织中内参基因标准化因子的配对差异分析,结果表明Vn/n+1均 < 0.15,可选择2个内参基因的组合,即ATP6+ND1。
2.6 EgAQPs基因的转录表达水平根据geNorm程序的评价结果,以actin和ActⅡ作为内参基因,采用RT-qPCR检测体外培养的原头蚴中EgAQPs基因的转录表达水平。结果表明,EgAQP3和EgAQP9在体外培养第3天的原头蚴中转录表达量最高,随着培养时间的增加,转录表达量呈持续下降的趋势,且不同培养时间转录表达量的差异有统计学意义(P < 0.05);EgAQP4在体外培养第25天的原头蚴中转录表达量最高(P < 0.05),在体外培养第3、15、35天的原头蚴中转录表达量较低,差异无统计学意义(P > 0.05)。EgAQP、EgAQP1、EgAQPFA-CHIP和EgAQPAnG在体外培养不同时间的原头蚴中的转录表达水平很低,基本上检测不到(表 2)。
| 基因 | 3 d | 15 d | 25 d | 35 d |
| EgAQP4 | 52.168±8.388 | 82.807±5.134 | 310.352±41.183b | 65.620±9.300 |
| EgAQP9 | 4 461.928±380.842a | 2 248.720±403.970a | 1 433.409±152.659a | 598.793±96.980a |
| EgAQP | 0.450±0.084 | 2.081±0.256 | 0.839±0.110 | 0.849±0.105 |
| EgAQPAnG | 0.658±0.042 | 2.860±0.333 | 0.156±0.008 | 0.111±0.005 |
| EgAQPFA-CHIP | 5.963±0.819 | 2.957±0.286 | 56.998±7.948 | 4.225±0.333 |
| EgAQP1 | 5.939±0.710 | 3.212±0.481 | 3.396±0.314 | 0.495±0.079 |
| EgAQP3 | 4 326.867±314.876a | 2 431.923±130.834a | 1 245.960±21.482a | 668.556±88.057a |
| a:P < 0.05,同基因组内各时间点两两比较;b:P < 0.05,与同基因组内其他时间点比较 | ||||
根据geNorm程序的评价结果,以ATP6和ND1作为内参基因,采用RT-qPCR检测继发棘球蚴囊壁组织中EgAQPs基因的转录表达水平。结果表明,EgAQP3、EgAQP4和EgAQP9在原头蚴中转录表达量最高(P < 0.05),随着感染时间的延长,EgAQP3、EgAQP4和EgAQP9的转录表达水平在接种原头蚴感染小鼠后第3~7个月的继发棘球蚴囊壁组织中表达水平很低,差异无统计学意义(P > 0.05);EgAQP、EgAQP1、EgAQPFA-CHIP和EgAQPAnG在继发棘球蚴囊壁组织中的转录表达水平均很低,基本上检测不到(表 3)。
| 基因 | 原头蚴 | 3个月囊 | 4个月囊 | 5个月囊 | 6个月囊 | 7个月囊 |
| EgAQP4 | 65.222±1.617a | 0.997±0.092 | 0.518±0.027 | 0.367±0.023 | 0.112±0.004 | 0.372±0.004 |
| EgAQP9 | 392.863±16.821a | 1.808±0.135 | 3.060±0.251 | 0.854±0.042 | 6.778±0.938 | 1.827±0.154 |
| EgAQP | 0.070±0.015 | 0.257±0.023 | 0.108±0.005 | 0.017±0.001 | 0.027±0.001 | 0.098±0.008 |
| EgAQPAnG | 0.214±0.008 | 0.255±0.024 | 0.176±0.019 | 0.064±0.004 | 0.038±0.002 | 0.111±0.004 |
| EgAQPFA-CHIP | 0.970±0.134 | 2.014±0.127 | 1.171±0.112 | 0.173±0.005 | 0.223±0.011 | 0.479±0.032 |
| EgAQP1 | 1.010±0.096 | 1.315±0.112 | 1.164±0.119 | 0.416±0.007 | 0.101±0.003 | 0.059±0.006 |
| EgAQP3 | 334.830±8.300a | 1.899±0.053 | 3.070±0.154 | 0.917±0.035 | 5.515±0.570 | 1.957±0.220 |
| a:P < 0.05,与同基因组内感染后各时间点比较 | ||||||
3 讨论
在以往的研究中,研究者们多选择常用的管家基因对RT-qPCR结果进行标准化,但近年来,经典的管家基因的稳定性受到了质疑。越来越多的研究表明,在不同的实验对象以及不同的实验处理条件下,管家基因的表达量变异较大[17-18]。盲目的使用一种管家基因作为内参,一方面可能使基因表达的微小差异难以发现;另一方面可能导致错误甚至相反的结论[19]。因此,研究者应根据自己的实验对象及处理条件筛选稳定的内参,以保证实验结果的可靠性。
棘球蚴原头蚴体外培养模型具有生长周期短﹑生长条件可控制等优势,能够在离体的情况下观察虫体的发育过程,为研究虫体发育及生活各阶段的基因表达水平提供材料[20-21]。由于小鼠获取方便、易控,而且能够很好地反映人体相对应的病理生理特征,国内外学者建立棘球蚴感染动物模型时,多选择小鼠作为实验动物。本研究选择BALB/c小鼠作为实验动物,通过腹腔接种从羊肝分离的原头蚴的方法,建立棘球蚴小鼠模型,观察原头蚴在小鼠体内发育为棘球蚴的过程中EgAQPs基因转录表达水平的动态变化。由于棘球蚴在小鼠体内发育为不育囊[22],本研究所提取的继发棘球蚴囊壁组织总RNA实际上就是继发棘球蚴囊壁生发层细胞的总RNA。本研究从细粒棘球蚴原头蚴和继发棘球蚴囊壁生发层细胞的总RNA着手,筛选原头蚴成囊过程中稳定的内参基因,并通过RT-qPCR检测EgAQPs基因在原头蚴发育为棘球蚴过程中的转录水平相对表达量,为探讨棘球蚴囊壁生发层细胞EgAQPs基因表达水平变化与棘球蚴液形成或积累的关系提供依据。
研究者利用RT-qPCR进行棘球绦虫目的基因表达模式的研究时,往往根据约定俗成或者基于其他物种中的经验,多选择单一内参进行数据校正和标准化[12-13, 15]。研究表明,使用单一内参基因对RT-qPCR数据进行校正和标准化,往往会产生相对较大的误差[23],因此在RT-qPCR数据标准化过程中,往往需要使用2个或者2个以上的内参基因对目的基因进行校正和标准化[24-25],从而减少实验误差,得到更加准确的结果。因此,根据具体的实验条件,筛选稳定的内参,是保证棘球绦虫目的基因表达结果准确定量的前提。
目前,基于基因芯片技术筛选内参基因和采用基于统计学的软件筛选内参基因是内参基因筛选的两种主要方法。基于基因芯片技术筛选获得的内参基因多为新的内参基因,它们的表达稳定性尚不确定,必须通过RT-qPCR等方法进行评价,并与常用内参基因的表达稳定性进行比较,才能确定这些新的内参基因是否可用于RT-qPCR结果的校正和标准化[26-27]。因此,研究者们常采用基于统计学的软件(如geNorm软件、NormFinder软件、Bestkeeper软件)筛选内参基因[28-29]。NormFinder软件和Bestkeeper软件可以筛选出某一具体实验条件下,所有候选内参基因中的最佳内参基因,但不能确定最佳内参基因的数目。geNorm软件除了可以筛选出最稳定的内参基因,还可以确定所需内参基因的最适数目,是一款专门用于RT-qPCR方法筛选内参基因的软件。采用geNorm软件对内参基因进行评估时通常考虑2个主要参数,即稳定性的平均值M和配对差异值Vn/n+1。内参基因的筛选一般选取3~18个候选内参基因[30],本研究以体外培养的细粒棘球蚴原头蚴及棘球蚴小鼠模型中采集的继发棘球蚴囊壁组织为研究对象,选用文献报道较多的ND1、ATP6、elp、actin和ActⅡ共5个常用管家基因作为候选内参基因,利用geNorm软件评估这些内参基因的稳定性。本实验5个候选内参基因的M值均小于geNorm软件关于理想内参基因筛选的阈值1.5,说明均可用于原头蚴成囊的基因转录表达研究。国际惯例认为M值< 0.5的内参基因稳定性较好[20]。在体外培养的原头蚴中,actin和ActⅡ的M值均小于0.5,说明这两个内参基因更适合体外培养的原头蚴样品的校正;同理,在继发棘球蚴囊壁组织中,ATP6和ND1的M值均小于0.5,说明这两个内参基因更适合继发棘球蚴囊壁组织样品的校正。geNorm程序默认的Vn/n+1阈值为0.15,如果Vn/n+1 < 0.15,说明候选内参基因中有n个适合作为RT-qPCR的内参基因,不需要引入第n+1个内参基因;如果Vn/n+1>0.15,说明需要引入第n+1个内参基因进行校正。本研究中,Vn/n+1均小于0.15,根据经济适用的原则,可选择2个内参基因的组合,即actin+ActⅡ或ATP6+ND1。
本研究对原头蚴成囊过程内参基因转录表达的稳定性进行评价,支持了在不同实验条件下内参基因稳定性不同的观点。在本研究中,actin和ActⅡ为检测体外培养的原头蚴EgAQPs基因转录表达状态的理想内参基因,而ATP6和ND1为检测继发棘球蚴囊壁生发层细胞EgAQPs基因转录表达状态的理想内参基因。在以actin和ActⅡ为内参检测体外培养的原头蚴EgAQPs基因的转录表达水平和以ATP6和ND1为内参检测继发棘球蚴囊壁生发层细胞EgAQPs基因转录表达水平的实验中,结果均表明,EgAQP3、EgAQP4和EgAQP9在原头蚴中转录表达水平较高,在接种原头蚴感染小鼠后的第3~7个月的继发棘球蚴囊壁生发层细胞中转录表达水平很低,基本上检测不到;EgAQP、EgAQP1、EgAQPFA-CHIP和EgAQPAnG在原头蚴和继发棘球蚴囊壁生发层细胞中的转录表达水平均很低,基本上检测不到。随着感染时间的延长,小鼠腹腔内棘球蚴包囊大小呈持续增加的趋势,很好地反映了棘球蚴液由少变多的过程,结合EgAQPs基因在原头蚴和继发棘球蚴囊壁生发层细胞中的转录表达水平,因此推测原头蚴EgAQPs基因的转录表达水平显著高于继发棘球蚴囊壁生发层细胞可能正好反映了原头蚴内充满实质细胞,实质细胞的生命活动需要EgAQPs的功能;原头蚴在小鼠体内长成继发棘球蚴的过程中,原头蚴的部分实质细胞死亡后的裂解液可能是构成棘球蚴液的成分;继发棘球蚴囊壁生发层细胞EgAQPs基因低表达则有利于生发层细胞的存活,因为囊壁外的水不能通过这些AQPs进入生发层细胞而使之裂解,同时棘球蚴囊内的水也不能通过这些AQPs进入生发层细胞而使之裂解,或者通过生发层细胞膜上的AQPs转移出棘球蚴囊壁,这些因素则有利于棘球蚴液的积累,使得棘球蚴包囊越长越大,且可以在中间宿主的器官或组织中长期存活。
总之,本研究检测了原头蚴成囊过程中EgAQPs基因的转录表达水平。在以RT-qPCR方法检测体外培养的原头蚴EgAQPs基因转录表达状态的实验中actin和ActⅡ为理想的内参基因;在以RT-qPCR方法检测继发棘球蚴囊壁生发层细胞EgAQPs基因转录表达状态的实验中ATP6和ND1为理想的内参基因。由于候选内参基因数量有限,其他基因是否适合作为内参基因尚待在今后的研究中进一步探索。
| [1] | ZHENG H J, ZHANG W B, ZHANG L, et al. The genome of the hydatid tapeworm Echinococcus granulosus[J]. Nat Genet, 2013, 45(10): 1168–1175. DOI:10.1038/ng.2757 |
| [2] | WOLFINGER R D, BEEDANAGARI S, BIOTIER E, et al. Two approaches for estimating the lower limit of quantitation (LLOQ) of microRNA levels assayed as exploratory biomarkers by RT-qPCR[J]. BMC Biotechnol, 2018, 18: 6. DOI:10.1186/s12896-018-0415-4 |
| [3] | HUIS R, HAWKINS S, NEUTELINGS G. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum L)[J]. BMC Plant Biol, 2010, 10: 71. DOI:10.1186/1471-2229-10-71 |
| [4] | SUN H F, MENG Y P, CUI G M, et al. Selection of housekeeping genes for gene expression studies on the development of fruit bearing shoots in Chinese jujube (Ziziphus jujube Mill)[J]. Mol Biol Rep, 2009, 36(8): 2183–2190. DOI:10.1007/s11033-008-9433-y |
| [5] |
张然然, 刘华淼, 邢秀梅, 等. 鹿茸组织中内参基因的筛选和验证[J].
中国畜牧兽医, 2015, 42(4): 883–889.
ZHANG R R, LIU H M, XING X M, et al. Selection and validation of reference genes in velvet antlers tissues[J]. China Ani Husb Vet Med, 2015, 42(4): 883–889. DOI:10.16431/j.cnki.1671-7236.2015.04.017 |
| [6] |
肖翠, 严佳文, 龙桂友, 等. 柑橘内参基因的稳定性评价[J].
果树学报, 2012, 29(6): 978–984.
XIAO C, YAN J W, LONG G Y, et al. Stability evaluation of reference genes in citrus[J]. J Fruit Sci, 2012, 29(6): 978–984. DOI:10.13925/j.cnki.gsxb.2012.06.010 |
| [7] |
李君英, 张鹤, 韩永峰, 等. 组培条件下苹果实时定量PCR内参基因的筛选[J].
果树学报, 2016, 33(9): 1033–1042.
LI J Y, ZHANG H, HAN Y F, et al. Selection of reference genes for real-time quantitative PCR in apples (Malus domestica) in vitro[J]. J Fruit Sci, 2016, 33(9): 1033–1042. DOI:10.13925/j.cnki.gsxb.20160097 |
| [8] |
张颋, 贾利芳, 陈英, 等. 细粒棘球绦虫组织蛋白酶B的重组表达及生物信息学分析[J].
中国血吸虫病防治杂志, 2014, 26(6): 642–647.
ZHANG T, JIA L F, CHEN Y, et al. Cloning, expression and bioinformation analysis of cathepsin B of Echinococcus granulosus[J]. Chin J Schisto Control, 2014, 26(6): 642–647. DOI:10.3969/j.issn.1005-6661.2014.06.011 |
| [9] | ERYILDIZ C, SAKRU N. Molecular characterization of human and animal isolates of Echinococcus granulosus in the thrace region, Turkey[J]. Balkan Med J, 2012, 29(3): 261–267. DOI:10.5152/balkanmedj.2012.072 |
| [10] |
肖云峰, 刘辉, 吕国栋, 等. 细粒棘球绦虫核糖体蛋白S9基因克隆鉴定及其在不同发育阶段表达分析[J].
中国病原生物学杂志, 2014, 9(2): 150–154.
XIAO Y F, LIU H, LYU G D, et al. Cloning and analysis of the ribosomal protein S9 gene from Echinococcus granulosus and analysis of its expression in different stages of Echinococcus granulosus development[J]. J Pathog Biol, 2014, 9(2): 150–154. DOI:10.13350/j.cjpb.140214 |
| [11] |
吕国栋, 叶建蔚, 张传山, 等. 细粒棘球绦虫DNA聚合酶δ小亚基EgPolD2基因的克隆及序列分析[J].
中国病原生物学杂志, 2012, 7(7): 510–512.
LYU G D, YE J W, ZHANG C S, et al. Molecular cloning and sequence analysis of EgPolD2 from Echinococcus granulosus in Xinjiang[J]. J Pathog Biol, 2012, 7(7): 510–512. DOI:10.13350/j.cjpb.2012.07.019 |
| [12] |
刘辉, 肖云峰, 赵军, 等. 细粒棘球绦虫未知功能基因384p02i04的克隆及生物信息学分析[J].
新疆医科大学学报, 2016, 39(4): 426–429.
LIU H, XIAO Y F, ZHAO J, et al. Molecular cloning and bionformatics analysis of a novel gene 384p02i04 in Echinococcus granulosus[J]. J Xinjiang Med Univ, 2016, 39(4): 426–429. DOI:10.3969/j.issn.1009-5551.2016.04.010 |
| [13] |
苏梦, 郭小腊, 杨静, 等. 多房棘球绦虫apomucin基因qPCR引物的筛选及潜在应用[J].
中国寄生虫学与寄生虫病杂志, 2016, 34(5): 394–398.
SU M, GUO X L, YANG J, et al. Screen and applications of qPCR primers for apomucin gene of Echinococcus multilocularis[J]. Chin J Parasitol Parasit Dis, 2016, 34(5): 394–398. |
| [14] | ARIKOGLU H, ARSLAN A, HEPDOGRU M A, et al. Expression profile and polymorphisms of actin genes in protoscoleces of Echinococcus granulosus from sheep in central Turkey[J]. Vet Parasitol, 2009, 166(1/2): 80–85. DOI:10.1016/j.vetpar.2009.08.007 |
| [15] |
郝力力, 李必富. 应用荧光定量PCR对细粒棘球绦虫miRNAs在虫体不同发育阶段表达量的研究[J].
中国农学通报, 2012, 28(26): 63–66.
HAO L L, LI B F. Quantification analysis of miRNAs expression level in different developing stages of Echinococcus granulosus using real-time PCR[J]. Chin Agric Sci Bull, 2012, 28(26): 63–66. DOI:10.3969/j.issn.1000-6850.2012.26.015 |
| [16] | VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol, 2002, 3(7): RESEARCH0034. |
| [17] |
周兰, 张利义, 张彩霞, 等. 苹果实时荧光定量PCR分析中内参基因的筛选[J].
果树学报, 2012, 29(6): 965–970.
ZHOU L, ZHANG L Y, ZHANG C X, et al. Screening of genes for real-time fluorescence quantitative PCR in apple[J]. J Fruit Sci, 2012, 29(6): 965–970. DOI:10.13925/j.cnki.gsxb.2012.06.008 |
| [18] | CONGIU M, SLAVIN J L, DESMOND P V. Expression of common housekeeping genes is affected by disease in human hepatitis C virus-infected liver[J]. Liver Int, 2011, 31(3): 386–390. DOI:10.1111/j.1478-3231.2010.02374.x |
| [19] |
赵晶, 赵俊杰, 康丽丽, 等. 人不同孕期胎盘组织中实时荧光定量PCR内参基因的选择[J].
西安交通大学学报(医学版), 2014, 35(2): 218–221.
ZHAO J, ZHAO J J, KANG L L, et al. Selection of suitable reference genes for quantitative real-time PCR in human villi and placenta of different pregnancy[J]. J Xi'an Jiaotong Univ (Med Sci), 2014, 35(2): 218–221. DOI:10.7652/jdyxb201401 |
| [20] |
倪兴维, 贾万忠, 者永辉, 等. 棘球蚴体外培养技术的应用[J].
中国寄生虫学与寄生虫病杂志, 2011, 29(4): 314–318.
NI X W, JIA W Z, ZHE Y H, et al. Application of in vitro cultivation technique for metacestodes in study of Echinococcus spp.[J]. Chin J Parasitol Parasit Dis, 2011, 29(4): 314–318. |
| [21] |
袁丽英, 张壮志, 石保新, 等. 细粒棘球绦虫—原头蚴在两种细胞培养液中体外培养的初步观察[J].
畜牧兽医杂志, 2008, 27(5): 16–18.
YUAN L Y, ZHANG Z Z, SHI B X, et al. In vitro cultivation protoscoleces of the protoslices of Echinococcus granulosus in medium RPMI-1640 and MEM[J]. J Ani Sci Vet Med, 2008, 27(5): 16–18. |
| [22] |
李红卫, 袁芳, 李燕兵, 等. 昆明种小鼠棘球蚴病感染动物模型的建立[J].
宁夏医科大学学报, 2011, 33(5): 411–413, 424.
LI H W, YUAN F, LI Y B, et al. Establishment of infection models of hydatidosis using Kunming mice[J]. J Ningxia Med Univ, 2011, 33(5): 411–413, 424. DOI:10.3969/j.issn.1674-6309.2011.05.004 |
| [23] | DIE J V, ROMÁN B, NADAL S, et al. Evaluation of candidate reference genes for expression studies in Pisum sativum under different experimental conditions[J]. Planta, 2010, 232(1): 145–153. DOI:10.1007/s00425-010-1158-1 |
| [24] |
王彦杰, 董丽, 张超, 等. 牡丹实时定量PCR分析中内参基因的选择[J].
农业生物技术学报, 2012, 20(5): 521–528.
WANG Y J, DONG L, ZHANG C, et al. Reference gene selection for real-time quantitative PCR normalization in tree Peony (Paeonia suffruticosa Andr.)[J]. J Agric Biotechnol, 2012, 20(5): 521–528. DOI:10.3969/j.issn.1674-7968.2012.05.008 |
| [25] |
冯波, 郭前爽, 毛必鹏, 等. 松墨天牛化学感受组织荧光定量PCR内参基因的鉴定与筛选[J].
昆虫学报, 2016, 59(4): 427–437.
FENG B, GUO Q S, MAO B P, et al. Identification and selection of valid reference genes for assaying gene expression in the chemosensory tissues of Monochamus alternates (Coleoptera: Cerambycidae) by RT-qPCR[J]. Acta Entomol Sinica, 2016, 59(4): 427–437. DOI:10.16380/j.kexh.2016.04.008 |
| [26] | LONG X Y, WANG J R, OUELLET T, et al. Genome-wide identification and evaluation of novel internal control genes for Q-PCR based transcript normalization in wheat[J]. Plant Mol Biol, 2010, 74(3): 307–311. DOI:10.1007/s11103-010-9666-8 |
| [27] |
袁伟, 万红建, 杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J].
植物学报, 2012, 47(4): 427–436.
YUAN W, WAN H J, YANG Y J. Characterization and selection of reference genes for real-time quantitative RT-PCR of plants[J]. Chin Bull Bot, 2012, 47(4): 427–436. DOI:10.3724/SP.J.1259.2012.00427 |
| [28] |
刘颖, 王双耀, 高乔, 等. 栉孔扇贝内参基因稳定性研究[J].
生态毒理学报, 2013, 8(4): 616–622.
LIU Y, WANG S Y, GAO Q, et al. Study on the expression stability of reference genes in Chlamys farreri[J]. Asian J Ecotoxico, 2013, 8(4): 616–622. DOI:10.7524/AJE.1673-5897.20130304001 |
| [29] |
张翠彩, 逄波, 蒋秀高, 等. 霍乱弧菌基因表达分析中内参基因的选择[J].
中国人兽共患病学报, 2014, 30(5): 433–438.
ZHANG C C, PANG B, JIANG X G, et al. Selection of reference genes for gene expression analysis in Vibrio cholera[J]. Chin J Zoonos, 2014, 30(5): 433–438. DOI:10.3969/cjz.j.issn.1002-2694.2014.05.001 |
| [30] |
岳秀利, 高新菊, 王进军, 等. 二斑叶螨内参基因的筛选及解毒酶基因的表达水平[J].
中国农业科学, 2013, 46(21): 4542–4549.
YUE X L, GAO X J, WANG J J, et al. Selection of reference genes and study of the expression levels of detoxifying enzymes of Tetranychus urticae (Acari: Tetranychidae)[J]. Scien Agric Sinica, 2013, 46(21): 4542–4549. DOI:10.3864/j.issn.0578-1752.2013.21.017 |



