胰腺癌具有恶性程度高、早期诊断困难和预后差的特点[1-5]。有研究发现胰腺癌患者中约90%存在远处转移[6],转移主要位于肝脏、腹部和肺。预计到2030年,胰腺癌将位于恶性肿瘤病死率的第2位[7]。我国2015年胰腺癌发病率已上升至第9位,死亡率位居第6位[8]。胰腺癌的发病率和致死率接近[9]。胰腺癌患者生存时间短的主要原因是早期诊断率低,确诊时往往已是晚期,失去最佳的手术治疗机会[10]。为了改善胰腺癌患者的预后,寻找可靠的胰腺癌早期诊断标志物和治疗靶点是临床诊治的迫切需求。
环状RNA(circular RNA, circRNA)是一类不具有5′和3′末端的呈闭合环状结构的特殊内源性非编码RNA(no coding RNA,ncRNA)[11],已经成为RNA研究领域的新热点。circRNA广泛存在于多种真核生物中,且在同种生物的不同组织中的表达具有明显的组织特异性[12-19]。2013年JECK等[11]研究发现某些circRNA的表达量远高于其线性转录本的表达量,且由于它不具有线性的5′和3′末端,不易被RNA酶降解,因此比线性更稳定[20]。circRNA主要定位在细胞质中,且大多数具有高度保守的序列[11, 20-21]。近年来针对circRNA的研究显示,其在生物的生长发育过程中的基因表达方面具有重要的调控作用。circRNA主要参与转录和转录后水平的调控[20, 22]。有研究发现circRNA与多种疾病的发生和发展密切相关,特别是在肿瘤的发生和发展中发挥了重要的生物学功能[23-24]。circRNA具有miRNA应答元件(microRNA response element,MRE),可以通过MRE与miRNA结合,发挥“miRNA海绵(miRNA sponge)”功能,竞争性结合并抑制miRNA的活性,调控miRNA的下游靶基因的表达,完成在转录后水平上对肿瘤靶基因表达的调控[25-26]。有研究发现吸附miRNA-7的环状RNA(circular RNA sponge for miR-7,ciRS-7)调控miR-7的下游靶基因表达水平增加[27];cir-SRY作为“miR-138海绵”抑制miR-138的活性[26],影响肿瘤的侵袭和转移[28];cir-ITCH结合miR-7、miR-17和miR-214,发挥抗肿瘤功能[29, 30]; hsa_circRNA_001569抑制miR-145活性,促进结肠癌的增殖和侵袭[31]。QU等[32]检测了胰腺导管细胞癌和正常胰腺组织内circRNA的表达差异,发现ciRS-7的表达水平与患者肿瘤直径、分期及临床预后负相关,推测其可能成为胰腺癌新的治疗靶点,同时发现ci-sirt7对其亲本基因有顺式调控作用。
本研究旨在通过基因芯片筛选出在高侵袭性胰腺癌细胞Hs 766T-L2中表达上调的circRNA-IARS,并用qRT-PCR技术检测胰腺癌患者癌组织及癌旁组织中circRNA-IARS和linar-IARS的表达水平,分析其表达与临床病理特征和预后的关系,探讨circRNA-IARS作为胰腺癌早期诊断和预后预测因素及成为治疗靶点的价值和意义。
1 材料与方法 1.1 材料 1.1.1 临床标本本研究经西南医院伦理委员会批准(2012年)。共选取2012年1月至2017年1月期间陆军军医大学第一附属医院肝胆外科手术治疗的115例胰腺癌患者的癌组织,均经手术病理证实(所有病例术前未行放疗、化疗或免疫治疗);其中男性91例,女性24例;年龄>60岁52例,≤60岁63例;胰头部肿瘤90例,胰体尾部肿瘤25例;直径>2的81例,≤2的34例;有神经损害43例,有十二指肠侵袭16例,淋巴结侵袭41例,有血管侵袭29例,有肝转移15例。根据国际抗癌联盟(UICC,union for international cancer control)的临床分期标准进行肿瘤TNM分期,Ⅰ、Ⅱa期65例,Ⅱb、Ⅲ、Ⅳ期50例。临床病历资料均来自患者的住院病历,由肝胆外科研究所临床随访中心在患者手术出院后每3个月电话随访1次,直至失访或患者死亡,其中6例无随访资料。
1.1.2 细胞与试剂人胰腺癌细胞株Hs 766T购于中国医学科学院基础医学研究所细胞中心,Hs 766T-L2来自第2代Hs 766T裸鼠胰腺癌肝转移原代细胞[33]。总RNA提取试剂TRIzol购于美国Thermo公司,反转录试剂盒、SYBR Green试剂盒购于日本TaKaRa公司,内参照和特异性引物由中国生工生物工程(上海)公司合成,胎牛血清购于日本ZETA LIFE公司,青链霉素溶液、RPMI1640培养基购于美国Gibco公司。
1.2 实验方法 1.2.1 细胞培养Hs 766T、Hs 766T-L2细胞培养于含10%胎牛血清和1%青链霉素溶液的RPMI1640培养基,置于37 ℃、5%CO2的培养箱。
1.2.2 circRNA芯片采用中国康成生物公司circRNA芯片(version 11.0.1.1)比较胰腺癌细胞Hs 766T和Hs 766T-L2中circRNA表达差异。circRNA芯片结果结合circBase/miRBase/TargetScan Human7.1/ microRNA.org数据库生物信息学分析,筛选出circRNA-IARS进一步研究。
1.2.3 qRT-PCR检测TRIzol提取组织总RNA,用RNase焦磷酸二乙酯水溶解,然后用Thermo NanoDrop3000分光光度计测定总RNA的浓度和纯度。筛选胰腺癌标本的标准:①确保RNA纯度,RNA的光密度值标准为:1.8<D(260)/D(280)<2.0;②RNA琼脂糖凝胶电泳分析,显示有清晰的3个条带,且28S的条带不弱于18S的条带。按照反转录试剂盒说明书反转录生成cDNA,再以cDNA为模板按照SYBRⅡ试剂盒说明书进行PCR检测。GAPDH作为内参对照。circRNA-IARS上游引物:5′-CCAACATTACAGACCGGTGG-3′;下游引物:5′-CTCGAAGTTGGAA-AGTGGAGTG-3′。linar-IARS上游引物:GGTCAGTGGCGGAACTTGAAGAAC;下游引物:CAGCGTGAAGGAATGGTCAGGTG。GAPDH上游引物:5′-ATTCCATGGCACCGTCAAGGCTGA-3′;下游引物:5′-TTCTCCATGGTGGTGAAGACGCCA-3′。反应条件:95 ℃预变性30 s,进入下列循环:95 ℃ 5 s,60 ℃ 30 s,检测,再重复39个循环,共计40个循环,然后95 ℃ 10 s,65 ℃ 5s,95 ℃ 5 s,0.5 ℃ 5 s,检测。以2-△△Ct方法计数circRNA-IARS的相对表达量。
1.3 统计学分析数据录入Excel表格,以RNA的表达量中位数为分界点,将circRNA-IARS和linar-IARS的表达水平分为高表达组和低表达组。采用SPSS 19.0统计软件分析数据,每组实验至少重复3次。所有数据先行正态分布拟合检验,符合正态分布的定量资料组间比较采用t检验,非正态分布资料行非参数检验;正态分布资料用x±s表示,非正态分布资料用中位数(25%,75%)表示;胰腺癌和癌旁组织间表达差异用独立样本Mann-Whitney U检验;相关性用χ2检验,不满足χ2检验条件的用Fisher精确检验(Monte Carlo法);生存分析用Kaplan-Meier检验,并绘制生存曲线,影响预后的多因素分析用COX回归。
2 结果 2.1 circRNA在胰腺癌细胞Hs 766T和Hs 766T-L2间的表达差异利用circRNA芯片比较胰腺癌高侵袭性Hs 766T-L2细胞与低侵袭性Hs 766T细胞之间circRNA的表达差异,发现49条circRNA表达有差异,其中28条circRNA在Hs 766T-L2细胞内高表达,21条低表达(表 1)。通过circBase/miRBase/TargetScan Human7.1 /microRNA.org等数据库生物信息学分析,发现circRNA-IARS可能通过Rho信号通路调控肿瘤的侵袭转移。进一步进行3次独立重复的qRT-PCR检测,验证芯片结果,结果显示circRNA-IARS在高侵袭性Hs 766T-L2细胞中表达量为(4.11±0.65),明显高于低侵袭性Hs 766T细胞[(1.03±0.25),P<0.01],与基因芯片检测结果一致。
| 环状RNA名称 | 变化倍数 |
| hsa_circ_0000228 | 1.36 |
| hsa_circ_0004370 | 1.33 |
| hsa_circ_0069748 | 1.36 |
| hsa_circ_0003868 | 1.32 |
| hsa_circ_0075917 | 1.43 |
| hsa_circ_0076780 | 1.39 |
| hsa_circ_0078346 | 1.35 |
| hsa_circ_0080121 | 1.40 |
| hsa_circ_0082271 | 1.36 |
| hsa_circ_0004351 | 1.31 |
| hsa_circ_0008016 | 1.80 |
| hsa_circ_0084151 | 2.28 |
| hsa_circ_0005962 | 1.33 |
| hsa_circ_0086694 | 1.35 |
| hsa_circ_0088045 | 1.53 |
| hsa_circ_0088046 | 1.35 |
| hsa_circ_0092275 | 1.30 |
| hsa_circ_0092363 | 1.37 |
| hsa_circ_0092330 | 1.58 |
| hsa_circ_0092283 | 1.38 |
| hsa_circ_0092290 | 1.33 |
| hsa_circ_0086740 | -1.70 |
| hsa_circ_0005939 | -1.67 |
| hsa_circ_0083126 | -1.54 |
| hsa_circ_0086645 | -1.50 |
| hsa_circ_0078738 | -1.47 |
| hsa_circ_0034762 | -1.47 |
| hsa_circ_0016404 | -1.46 |
| hsa_circ_0076413 | -1.42 |
| hsa_circ_0009021 | -1.40 |
| hsa_circ_0001626 | -1.40 |
| hsa_circ_0001529 | -1.39 |
| hsa_circ_0007409 | -1.39 |
| hsa_circ_0076058 | -1.39 |
| hsa_circ_0088227 | -1.38 |
| hsa_circ_0007018 | -1.37 |
| hsa_circ_0000981 | -1.33 |
| hsa_circ_0008419 | -1.33 |
| hsa_circ_0083766 | -1.33 |
| hsa_circ_0008628 | -1.33 |
| hsa_circ_0073858 | -1.33 |
| hsa_circ_0083964 | -1.33 |
| hsa_circ_0036627 | -1.33 |
| hsa_circ_0087421 | -1.32 |
| hsa_circ_0052760 | -1.32 |
| hsa_circ_0043893 | -1.32 |
| hsa_circ_0087493 | -1.32 |
| hsa_circ_0008729 | -1.32 |
| hsa_circ_0087305 | -1.31 |
2.2 筛选circRNA-IARS在胰腺癌患者癌组织中高表达
对25例胰腺癌组织及配对癌旁组织circRNA-IARS的表达量数据进行正态分布拟合检验,发现数据属于非正态分布资料,故表达量用中位数(25%,75%)表示,且采用独立样本Mann-Whitney U检验分析。结果显示患者癌组织中circRNA-IARS的相对表达量为6.664(2.158,12.098),而癌旁组织中相对表达量为1.248(0.340,3.745),显著低于癌组织(P<0.01)。linar-IARS在癌组织和癌旁组织表达水平的差异无统计学意义(P>0.05),提示胰腺癌与circRNA-IARS高表达有关,而与linar-IARS表达水平无关。
2.3 癌组织circRNA-IARS的表达与胰腺癌临床病理特征的关系分别以胰腺癌组织中circRNA-IARS和linar-IARS的中位表达量为分界点,将其分为高表达组和低表达组,并分析circRNA-IARS和linar-IARS表达水平与临床病理特征之间的关系。结果显示,circRNA-IARS的高表达与肿瘤的淋巴结侵袭、血管侵袭、肝转移及TNM分期有关(P<0.05),与患者的年龄、性别、肿瘤位置、肿瘤直径、神经侵害、十二指肠侵袭、组织分化无关(P>0.05);而linar-IARS的表达水平与胰腺癌的临床病理特征均无关(表 2)。
| 临床病理特征 | 环状RNA-IARS | P值 | 线性IARS | P值 | ||
| 高表达 (n=57) |
低表达 (n=58) |
高表达 (n=57) |
低表达 (n=58) |
|||
| 性别 | 0.612 | 0.384 | ||||
| 男 | 44 | 47 | 47 | 44 | ||
| 女 | 13 | 11 | 10 | 14 | ||
| 年龄/岁 | 0.506 | 0.932 | ||||
| ≤60 | 33 | 30 | 31 | 32 | ||
| >60 | 24 | 28 | 26 | 26 | ||
| 肿瘤位置 | 0.783 | 0.860 | ||||
| 胰头 | 44 | 46 | 45 | 45 | ||
| 胰体/尾 | 13 | 12 | 12 | 13 | ||
| 肿瘤直径/cm | 0.639 | 0.380 | ||||
| ≤2 | 18 | 16 | 19 | 15 | ||
| >2 | 39 | 42 | 38 | 43 | ||
| 神经侵害 | 0.516 | 0.202 | ||||
| 是 | 23 | 20 | 18 | 25 | ||
| 否 | 34 | 38 | 39 | 33 | ||
| 十二指肠侵袭 | 0.616 | 0.616 | ||||
| 是 | 7 | 9 | 7 | 9 | ||
| 否 | 50 | 49 | 50 | 49 | ||
| 组织分化 | 0.068 | 0.861 | ||||
| 低分化 | 13 | 9 | 11 | 13 | ||
| 中分化 | 42 | 38 | 41 | 39 | ||
| 高分化 | 2 | 9 | 5 | 6 | ||
| 淋巴结侵袭 | 0.027 | 0.607 | ||||
| 是 | 26 | 15 | 19 | 22 | ||
| 否 | 31 | 43 | 38 | 36 | ||
| 血管侵袭 | 0.016 | 0.308 | ||||
| 是 | 20 | 9 | 12 | 17 | ||
| 否 | 37 | 49 | 45 | 41 | ||
| 肝转移 | 0.011 | 0.155 | ||||
| 是 | 12 | 3 | 10 | 5 | ||
| 否 | 45 | 55 | 47 | 53 | ||
| 肿瘤TNM分期 | 0.007 | 0.647 | ||||
| Ⅰ、Ⅱa | 25 | 40 | 31 | 34 | ||
| Ⅱb、Ⅲ、Ⅳ | 32 | 18 | 26 | 24 | ||
| 在对组织分化进行检验时,有至少1格理论频数<5,故采用Fisher精确概率检验(Monte Carlo法) | ||||||
2.4 circRNA-IARS高表达与患者预后的关系
以circRNA-IARS在胰腺癌组织中的中位表达量为分界点,将胰腺癌患者分为高表达组(n=57)和低表达组(n=58),并对两组患者随访4年。以Kaplan-Meier法进行单因素生存分析,结果发现血管侵袭、肝转移、肿瘤TNM分期及circRNA-IARS表达水平是影响胰腺癌患者预后的独立危险因素(P<0.05),而年龄、肿瘤位置、肿瘤直径、神经侵害、十二指肠侵袭、组织分化、淋巴结侵袭及linar-IARS表达水平与胰腺癌的预后无关(P>0.05,表 3)。采用COX回归法对独立危险因素进行多因素分析,结果显示,circRNA-IARS表达水平和肿瘤TNM分期是影响患者预后的危险因素(P<0.05),而血管侵袭不是影响因素(P>0.05,表 4)。K-M法绘制circ-IARS生存曲线,结果发现circRNA-IARS高表达组患者的生存时间显著低于circRNA低表达组患者(P<0.05,图 1)。
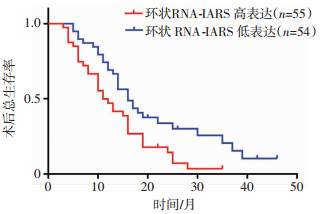
|
| 图 1 不同circ-IARS表达水平的病例生存分析 |
| 临床病理特征 | 例数 | 中位生存期/月 | P值 | χ2值 |
| 性别 | 0.253 | 1.308 | ||
| 男 | 86 | 16 | ||
| 女 | 23 | 14 | ||
| 年龄/岁 | 0.964 | 0.002 | ||
| ≤60 | 60 | 14 | ||
| >60 | 49 | 16 | ||
| 肿瘤位置 | 0.149 | 2.079 | ||
| 胰头 | 84 | 16 | ||
| 胰体、胰尾 | 25 | 12 | ||
| 肿瘤直径/cm | 0.073 | 3.207 | ||
| ≤2 | 32 | 18 | ||
| >2 | 77 | 14 | ||
| 神经侵害 | 0.062 | 3.490 | ||
| 是 | 40 | 14 | ||
| 否 | 69 | 16 | ||
| 十二指肠侵袭 | 0.204 | 1.613 | ||
| 是 | 16 | 10 | ||
| 否 | 93 | 16 | ||
| 组织分化 | 0.821 | 0.395 | ||
| 低分化 | 22 | 14 | ||
| 中分化 | 77 | 14 | ||
| 高分化 | 10 | 18 | ||
| 淋巴结侵袭 | 0.185 | 1.757 | ||
| 是 | 40 | 12 | ||
| 否 | 69 | 16 | ||
| 血管侵袭 | 0.024 | 5.127 | ||
| 是 | 27 | 14 | ||
| 否 | 82 | 16 | ||
| 肝转移 | <0.001 | 65.493 | ||
| 是 | 15 | 5 | ||
| 否 | 94 | 16 | ||
| 肿瘤TNM分期 | 0.010 | 6.626 | ||
| Ⅰ、Ⅱa | 60 | 16 | ||
| Ⅱb、Ⅲ、Ⅳ | 49 | 11 | ||
| 环状表达 | 0.013 | 6.233 | ||
| 高表达 | 55 | 13 | ||
| 低表达 | 54 | 16 | ||
| 线状表达 | 0.882 | 0.022 | ||
| 高表达 | 56 | 16 | ||
| 低表达 | 53 | 14 |
| 独立影响因素 | P值 | HR | 95%CI | |
| 单因素 | 多因素 | |||
| 血管侵袭 | 0.024 | 0.205 | ||
| 肿瘤TNM分期 | 0.010 | 0.033 | 1.598 | 1.040~2.456 |
| 环状RNA表达 | 0.013 | 0.039 | 1.577 | 1.023~2.432 |
3 讨论
circRNA被首次发现至今,已经有四十余年的历史[12]。在发现circRNA之后的近30年时间里,对其结构和功能的研究都比较少。基因测序技术的发展掀起了科研人员研究circRNA的热潮。已有研究显示,circRNA因其独特的闭合环状结构而呈现高度保守的特征和组织特异性,而且在多种生物体的各种体液中广泛存在[11-19]。随着针对circRNA研究的不断深入,特别是在《Nature》等杂志上发表的两篇关于circRNA生物学功能的研究[26, 34],进一步提高了研究人员对circRNA的重视程度。近年来,研究人员发现越来越多的疾病发生、发展与circRNA有关(如动脉粥样硬化[35]、病毒性肝炎[36]等),特别是许多肿瘤的发生、发展和侵袭、转移也与circRNA关系密切[27, 30, 37-39]。QU等[32]对比研究了circRNA在胰腺导管腺癌和正常胰腺组织中的表达差异, 发现环状RNA ciRS-7的表达水平与患者肿瘤直径、分期及临床预后呈负相关,推测其可能成为胰腺癌新的治疗靶点,同时发现ci-sirt7对其亲本基因有顺式调控作用。本研究利用circRNA基因芯片比较了胰腺癌高侵袭性Hs 766T-L2细胞与低侵袭性Hs 766T细胞之间的circRNA的表达差异,芯片结果显示有49条circRNA的表达呈现明显差异,其中28条circRNA在Hs 766T-L2细胞内高表达,21条低表达。通过生物信息学分析,我们发现circRNA-IARS可能通过Rho信号通路调控肿瘤转移微环境,促进肿瘤细胞的侵袭、转移。进一步经过3次独立重复的qRT-PCR检测发现,circRNA-IARS在Hs 766T-L2细胞中表达水平明显高于Hs 766T细胞。此结果与基因芯片检测数据是一致的,因此我们推测,circ-IARS的高表达可能与胰腺癌的侵袭能力有关。
为了进一步研究circ-IARS在胰腺癌中的表达情况,我们设计了circ-IARS和linear IARS的引物,并通过qRT-PCR技术检测其在胰腺癌组织和癌旁组织内的表达情况。结果显示,与正常胰腺相比,胰腺癌中circRNA-IARS的表达水平明显上调,而linar-IARS的表达水平没有明显差异,此结果提示胰腺癌的发生与circRNA-IARS的高表达有关,与linar-IARS表达水平无关。
已有研究发现,circRNA的表达与肿瘤的侵袭、转移能力有关。SAYDAM等[40]研究指出高表达的ciRs-7通过结合miR-7,激活PI3K/AKT通路,进而推进细胞周期,增强肝癌细胞的侵袭能力。本研究表明,circ-IARS高表达与胰腺癌的血管侵袭和肝转移有关,其是胰腺癌发生的促癌基因。首先,我们检测了115例胰腺癌组织中circ-IARS的表达量,通过统计学分析后发现,circRNA-IARS的高表达与胰腺癌的淋巴结侵袭、血管侵袭、肝转移及TNM分期呈明显正相关,与年龄、性别、肿瘤位置、肿瘤直径、神经侵害、十二指肠侵袭和组织分化无关。这表明circRNA-IARS的高表达与胰腺癌的侵袭转移有关。我们利用Kaplan-Meier法行单因素生存分析发现,血管侵袭、肝转移、肿瘤TNM分期及circRNA-IARS高表达水平是影响胰腺癌患者预后的独立危险因素,而性别、年龄、肿瘤位置、肿瘤直径、神经侵害、十二指肠侵袭、组织分化、淋巴结侵袭及linar-IARS表达水平与胰腺癌的预后无关。通过COX回归多因素分析发现,circRNA-IARS高表达、肝转移和肿瘤TNM分期是影响患者预后的危险因素。K-M法绘制circ-IARS表达水平相关的生存曲线,结果发现,circ-IARS高表达组术后生存时间明显比低表达组短,即胰腺癌组织中circRNA-IARS的表达水平与患者的术后生存时间及预后呈负相关。至于患者生存期缩短是由肿瘤生物学特性还是术后化疗造成的,需要更进一步的研究。
综上所述,本研究采用特异性方法检测115例胰腺癌组织中circ-IARS的表达,并进一步通过临床生存分析评估其在胰腺癌转移和预后中的作用。我们发现,circRNA-IARS在胰腺癌患者癌组织中表达水平明显升高,并且与胰腺癌的发生、转移及预后有关。由此可推测,circRNA-IARS可作为评判胰腺癌治疗效果和预测预后的临床检测标志物。虽然对于circRNA-IARS在促进胰腺癌的侵袭转移过程中的具体调控机制并不十分清楚,我们在以后的研究中,将着眼于circRNA的特征如发挥“miRNA海绵”作用抑制miRNA的表达、对胰腺癌进展过程中的相关指标的影响及与促肿瘤侵袭转移因素的关系进行深入探索。我们也相信随着科学研究的不断深入,研究人员能够发现越来越多的与胰腺癌相关的circRNA,并研究它们在胰腺癌的预防、早期诊断及靶向治疗方面的作用和意义。
| [1] | DUAN Y F, LI D F, LIU Y H, et al. Decreased expression of DAB2IP in pancreatic cancer with wild-type KRAS[J]. Hepatobiliary Pancreat Dis Int, 2013, 12(2): 204–209. DOI:10.1016/s1499-3872(13)60032-6 |
| [2] | DAMASKOS C, GARMPIS N, KARATZAS T, et al. Histone Deacetylase (HDAC) Inhibitors: Current Evidence for Therapeutic Activities in Pancreatic Cancer[J]. Anticancer Res, 2015, 35(6): 3129–3135. |
| [3] | NIPP R D, RYAN D P. Predicting a response to FOLFIRINOX in pancreatic cancer[J]. J Natl Cancer Inst, 2015, 107(8): djv175–djv175. DOI:10.1093/jnci/djv175 |
| [4] | HOFMANN B T, SCHLVTER L, LANGE P, et al. COSMC knockdown mediated aberrant O-glycosylation promotes oncogenic properties in pancreatic cancer[J]. Mol Cancer, 2015, 14(1). DOI:10.1186/s12943-015-0386-1 |
| [5] | VENNIN C, PAJIC M, TIMPSON P. Imaging fibrosis in pancreatic cancer using second harmonic generation[J]. Pancreatology, 2015, 15(2): 200–201. DOI:10.1016/j.pan.2015.02.004 |
| [6] | FRAMPAS E, DAVID A, REGENET N, et al. Pancreatic carcinoma: Key-points from diagnosis to treatment[J]. Diagn Interv Imaging, 2016, 97(12): 1207–1223. DOI:10.1016/j.diii.2016.07.008 |
| [7] | RAHIB L, SMITH B D, AIZENBERG R, et al. Projecting cancer incidence and deaths to 2030: the unexpected burden of thyroid, liver, and pancreas cancers in the United States[J]. Cancer Res, 2014, 74(11): 2913–2921. DOI:10.1158/0008-5472.CAN-14-0155 |
| [8] | CHEN W, ZHENG R, BAADE P D, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115–132. DOI:10.3322/caac.21338 |
| [9] | SIEGEL R, NAISHADHAM D, JEMAL A. Cancer statistics, 2012[J]. CA Cancer J Clin, 2012, 62(1): 10–29. DOI:10.3322/caac.20138 |
| [10] | REAL F X. A "catastrophic hypothesis" for pancreas cancer progression[J]. Gastroenterology, 2003, 124(7): 1958–1964. DOI:10.1016/S0016-5085(03)00389-5 |
| [11] | JECK W R, SORRENTINO J A, WANG K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141–157. DOI:10.1261/rna.035667.112 |
| [12] | SANGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci U S A, 1976, 73(11): 3852–3856. DOI:10.1073/pnas.73.11.3852 |
| [13] | MATSUMOTO Y, FISHEL R, WICKNER R B. Circular single-stranded RNA replicon in Saccharomyces cerevisiae[J]. Proc Natl Acad Sci U S A, 1990, 87(19): 7628–7632. DOI:10.1073/pnas.87.19.7628 |
| [14] | NIGRO J M, CHO K R, FEARON E R, et al. Scrambled exons[J]. Cell, 1991, 64(3): 607–613. DOI:10.1016/0092-8674(91)90244-s |
| [15] | COCQUERELLE C, DAUBERSIES P, MAJÉRUS M A, et al. Splicing with inverted order of exons occurs proximal to large introns[J]. EMBO J, 1992, 11(3): 1095–1098. |
| [16] | CAPEL B, SWAIN A, NICOLIS S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019–1030. DOI:10.1016/0092-8674(93)90279-Y |
| [17] | COCQUERELLE C, MASCREZ B, HǪTUIN D, et al. Mis-splicing yields circular RNA molecules[J]. FASEB J, 1993, 7(1): 155–160. DOI:10.1096/fasebj.7.1.7678559 |
| [18] | DANAN M, SCHWARTZ S, EDELHEIT S, et al. Transcriptome-wide discovery of circular RNAs in Archaea[J]. Nucleic Acids Res, 2012, 40(7): 3131–3142. DOI:10.1093/nar/gkr1009 |
| [19] | WANG P L, BAO Y, YEE M C, et al. Circular RNA is expressed across the eukaryotic tree of life[J]. PLoS ONE, 2014, 9(6): e90859. DOI:10.1371/journal.pone.0090859 |
| [20] | ZHANG Y, ZHANG X O, CHEN T, et al. Circular intronic long noncoding RNAs[J]. Mol Cell, 2013, 51(6): 792–806. DOI:10.1016/j.molcel.2013.08.017 |
| [21] | SALZMAN J, GAWAD C, WANG P L, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS ONE, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733 |
| [22] | QU S, YANG X, LI X, et al. Circular RNA: A new star of noncoding RNAs[J]. Cancer Lett, 2015, 365(2): 141–148. DOI:10.1016/j.canlet.2015.06.003 |
| [23] | WANG F, NAZARALI A J, JI S. Circular RNAs as potential biomarkers for cancer diagnosis and therapy[J]. Am J Cancer Res, 2016, 6(6): 1167–1176. |
| [24] | DIMAURO S, SCHON E A. Mitochondrial DNA mutations in human disease[J]. Am J Med Genet, 2001, 106(1): 18–26. DOI:10.1002/ajmg.1392 |
| [25] | JECK W R, SHARPLESS N E. Detecting and characterizing circular RNAs[J]. Nat Biotechnol, 2014, 32(5): 453–461. DOI:10.1038/nbt.2890 |
| [26] | HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384–388. DOI:10.1038/nature11993 |
| [27] | HANSEN T B, KJEMS J, DAMGAARD C K. Circular RNA and miR-7 in cancer[J]. Cancer Res, 2013, 73(18): 5609–5612. DOI:10.1158/0008-5472.CAN-13-1568 |
| [28] | ZHAO Z J, SHEN J. Circular RNA participates in the carcinogenesis and the malignant behavior of cancer[J]. RNA Biol, 2017, 14(5): 514–521. DOI:10.1080/15476286.2015.1122162 |
| [29] | LI F, ZHANG L, LI W, et al. Circular RNA ITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway[J]. Oncotarget, 2015, 6(8): 6001–6013. DOI:10.18632/oncotarget.3469 |
| [30] | HUANG G, ZHU H, SHI Y, et al. cir-ITCH plays an inhibitory role in colorectal cancer by regulating the Wnt/β-catenin pathway[J]. PLoS ONE, 2015, 10(6): e0131225. DOI:10.1371/journal.pone.0131225 |
| [31] | XIE H, REN X, XIN S, et al. Emerging roles of circRNA_001569 targeting miR-145 in the proliferation and invasion of colorectal cancer[J]. Oncotarget, 2016, 7(18). DOI:10.18632/oncotarget.8589 |
| [32] | QU S, SONG W, YANG X, et al. Microarray expression profile of circular RNAs in human pancreatic ductal adenocarcinoma[J]. Genom Data, 2015, 5: 385–387. DOI:10.1016/j.gdata.2015.07.017 |
| [33] | ZOU Y, LI J, CHEN Z, et al. miR-29c suppresses pancreatic cancer liver metastasis in an orthotopic implantation model in nude mice and affects survival in pancreatic cancer patients[J]. Carcinogenesis, 2015, 36(6): 676–684. DOI:10.1093/carcin/bgv027 |
| [34] | WERFEL S, NOTHJUNGE S, SCHWARZMAYR T, et al. Characterization of circular RNAs in human, mouse and rat hearts[J]. J Mol Cell Cardiol, 2016, 98: 103–107. DOI:10.1016/j.yjmcc.2016.07.007 |
| [35] | WANG K, LONG B, LIU F, et al. A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223[J]. Eur Heart J, 2016, 37(33): 2602–2611. DOI:10.1093/eurheartj/ehv713 |
| [36] | BURD C E, JECK W R, LIU Y, et al. Expression of linear and novel circular forms of an INK4/ARF-associated non-coding RNA correlates with atherosclerosis risk[J]. PLoS Genet, 2010, 6(12): e1001233. DOI:10.1371/journal.pgen.1001233 |
| [37] | KONG X, LI G, YUAN Y, et al. MicroRNA-7 inhibits epithelial-to-mesenchymal transition and metastasis of breast cancer cells via targeting FAK expression[J]. PLoS ONE, 2012, 7(8): e41523. DOI:10.1371/journal.pone.0041523 |
| [38] | QIN M, LIU G, HUO X, et al. Hsa_circ_0001649: A circular RNA and potential novel biomarker for hepatocellular carcinoma[J]. Cancer Biomark, 2016, 16(1): 161–169. DOI:10.3233/CBM-150552 |
| [39] | SAYDAM O, SENOL O, WVRDINGER T, et al. miRNA-7 attenuation in Schwannoma tumors stimulates growth by upregulating three oncogenic signaling pathways[J]. Cancer Res, 2011, 71(3): 852–861. DOI:10.1158/0008-5472.CAN-10-1219 |



