2. 330031 南昌, 南昌大学医学院玛丽女王学院中英2014级6班
2. Class 6, Joint Program Grade 2014, Nanchang University Queen Mary School, Nanchang, Jiangxi Province, 330031, China
抗菌药物的滥用导致临床病原菌对抗菌药物的耐药性不断增强。近年来, “多耐药”或“泛耐药”鲍曼不动杆菌所致的临床感染病例快速增加, 成为临床抗感染治疗的棘手问题[1]。在“后抗生素时代”, 人们一方面加强抗菌药物的使用管理, 延长抗生素的使用寿命; 另一方面更加关注新型抗菌药物、制剂的研发, 寻找防控细菌感染的新策略。噬菌体作为一类能感染、裂解细菌等微生物的病毒, 其在细菌感染防控中的作用又重新被人们所重视。
根据噬菌体对宿主菌作用方式的不同, 可分为裂解性噬菌体和溶源性噬菌体。裂解性噬菌体感染细菌后, 可利用宿主菌胞内的物质和代谢途径进行自我高效复制, 迅速增殖形成数以百计的子代噬菌体, 并通过裂解酶(lysin/endolysin)破坏宿主菌的细胞壁, 最终导致宿主菌的裂解死亡。目前, 裂解酶作为一种潜在的抗菌制剂, 与抗生素相比, 具有使用剂量小, 特异性强等优点[2-3]。然而, 由于大部分噬菌体的裂解酶蛋白结构缺少信号肽, 自身不能够穿透宿主菌的细胞膜, 被限制在细胞质中。因此, 噬菌体裂解宿主菌不仅需要裂解酶, 还需要另一种重要蛋白质, 即穿孔素(Holin)的辅助作用。穿孔素通过在细胞膜上形成跨膜蛋白通道, 使裂解酶能够穿过细胞膜到达细胞壁肽聚糖层而发挥裂解功能[4]。现有研究表明, 双链DNA噬菌体普遍采用这种“穿孔素-裂解酶”二元裂解系统, 发挥裂解宿主菌的作用[5-6]。
目前, 关于鲍曼不动杆菌噬菌体的研究逐渐增多, 但对其裂解酶基因及其编码蛋白的报道较少, 且穿孔素基因的研究尚未见报道[7]。本研究以临床分离的鲍曼不动杆菌为宿主菌, 从医院污水中分离鉴定其裂解性噬菌体, 并对其进行全基因组测序。同时, 利用生物信息学相关软件和方法对其裂菌相关重要功能基因及其编码蛋白的结构以及生物学功能等进行分析, 预测鲍曼不动杆菌噬菌体的穿孔素及裂解酶基因的生物特性, 为噬菌体裂解酶的基因工程制备提供理论基础和参考。
1 材料与方法 1.1 主要试剂和仪器琼脂粉、酵母提取物、蛋白胨购自英国OXOID公司, 氯化钠购自重庆川东化工集团公司, 噬菌体基因组DNA提取试剂盒(Code No.AB114)购自北京艾比根生物技术有限公司。ThermoFisher Micro 21R离心机为赛默飞世尔科技(中国)有限公司产品, JEM-1400Plus透射电镜为日本JEOL公司产品, HiSeq2000高通量测序仪为美国Illumina公司产品。
1.2 菌株和培养基鲍曼不动杆菌临床分离菌株为陆军军医大学附属西南医院临床检验中心保存, 以其为宿主菌从医院污水中分离获得裂解性噬菌体。自配LB液体培养基(g/L):氯化钠10.0 g, 蛋白胨10.0 g, 酵母提取物5.0 g; 固体培养基:在LB培养基中加入琼脂粉15.0 g, 加水至1 L, pH=7.4, 高压灭菌后分装备用。
1.3 噬菌体的分离与鉴定 1.3.1 噬菌体富集与分离在本院污水处理站, 取消毒前污水100 mL, 用纱布粗滤, 去除明显杂质, 然后4 ℃ 10 000 r/min离心10 min除去沉淀, 上清液再经0.22 μmol/L滤膜过滤除菌; 污水滤液与50 mL LB培养液等体积混合后, 加入2 mL对数生长期的鲍曼不动杆菌宿主菌液[D(600)=0.6], 37 ℃、160 r/min振荡过夜培养; 次日4 ℃ 10 000 r/min离心10 min收集上清, 经滤膜过滤除菌后的滤液即为富集的鲍曼不动杆菌噬菌体原液。噬菌体的分离采用双层琼脂平板法, 以LB固体培养基做平板下层, 随后将噬菌体原液梯度稀释, 分别取10 μL稀释液与200 μL宿主菌液混合, 室温放置15 min, 加入45 ℃、0.5%的半固体LB培养基3 mL(水浴保温), 轻微涡旋混匀后, 倾倒在制备好的LB固体平板上制成双层平板; 待上层培养基凝固后, 倒置, 37 ℃培养箱中过夜培养后, 观察噬菌斑生长情况。若出现明显噬菌斑, 说明分离到了以鲍曼不动杆菌为宿主菌的噬菌体, 命名为噬菌体SWH-Ab-1。
1.3.2 噬菌体的纯化挑取1个边缘光滑、透明的较大噬菌斑, 接种到2 mL对数生长期的宿主菌液中, 37 ℃振荡培养6 h, 4 ℃ 10 000 r/min离心, 上清液用0.22 μmol/L滤膜过滤除菌后, 用双层琼脂平板法重复培养5次, 即可得到纯化的噬菌体。
1.3.3 噬菌体的电镜观察采用磷钨酸负染法, 方法如下:取20 μL纯化的噬菌体溶液, 滴在石蜡片上, 将铜网浸置于噬菌体液滴内, 15 min后将铜网自液滴中取出, 用滤纸吸去多余液体, 在铜网上加1滴2%磷钨酸染色10 min, 用滤纸吸去染液, 干燥后用透射电镜观察。
1.4 噬菌体SWH-Ab-1全基因组测序与分析 1.4.1 噬菌体核酸提取和酶切图谱将20 μL分离获得的噬菌体SWH-Ab-1(106 PFU/mL)接种至10 mL宿主菌液中(108 CFU/mL), 37 ℃振荡过夜培养至菌液澄清, 扩增噬菌体用于提取基因组DNA。混合培养物10 000×g 4 ℃离心10 min去除细胞碎片和残渣, 收集上清液再用0.22 μmol/L的微孔滤膜抽滤, 去除残留的宿主菌。然后采用噬菌体基因组DNA提取试剂盒提取噬菌体基因组DNA, 操作步骤按试剂盒说明书进行。用限制性核酸内切酶SpeⅠ、NdeⅠ和HindⅢ分别消化噬菌体SWH-Ab-1的基因组DNA, 并用0.8%的琼脂糖凝胶进行电泳分析。
1.4.2 噬菌体基因组DNA的纯度鉴定针对噬菌体SWH-Ab-1的宿主菌鲍曼不动杆菌的16S rDNA设计qPCR引物(8F-AGAGTTTGATCCTGGCTCAG, 338R-TGCTGCCTCCCGTAGGAGT), 建立qPCR方法, 反应体系:SYBR Premix Ex TaqTM Ⅱ(2×)终浓度1×, 用量12.5 μL; 8F(10 μmol/L)终浓度0.4 μmol/L, 用量1 μL; 338R(10 μmol/L)终浓度0.4 μmol/L, 用量1 μL; DNA模板2.0 μL; ddH2O 8.5 μL; 总体积25 μL。在95 ℃(2 min)→95 ℃(30 s)→54 ℃(30 s)的条件下扩增40个循环, 检测所提取的噬菌体DNA中宿主菌的16S rDNA的残留量, 并与同浓度的宿主菌基因组DNA的量比较, 确定提取的噬菌体DNA的纯度。
1.4.3 全基因组测序浓度和纯度达到测序要求的噬菌体基因组DNA由深圳华大基因科技有限公司进行基因组测序。构建插入片段约500 bp的PCR-free测序文库, 采用Hiseq2000测序仪进行测序。获得的原始测序数据, 经过预处理去除接头、引物及低质量数据后, 并通过优化参数Kmer值, 进行噬菌体基因组序列的Survey组装, 获得噬菌体SWH-Ab-1最好的DNA序列拼接结果。同时, 采用CGView(http://wishart.biology.ualberta.ca/cgview/)方法绘制出噬菌体的全基因组图谱。应用NCBI的核酸比对软件Nucleotide blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)和数据库对SWH-Ab-1与其他噬菌体的基因同源性进行分析。
1.4.4 编码基因预测及重要功能基因定位分析将噬菌体SWH-Ab-1的基因组DNA序列以FASTA格式输入Glimmer软件(https://www.ncbi.nlm.nih.gov/genomes/MICROBES/glimmer_3.cgi), Genetic code选项选择11(Bacteria), 对噬菌体SWH-Ab-1的功能编码基因进行预测。同时, 在NCBI网站应用BLAST程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)对预测所得的功能基因进行逐个检索分析, 以FASTA格式输入编码基因的氨基酸序列, 选择blastp(protein-protein BLAST)程序, 其他为默认参数。另外, 利用blastp程序, 对预测获得的穿孔素和裂解酶的氨基酸序列与数据库中的蛋白质进行同源比对分析。
1.5 噬菌体SWH-Ab-1穿孔素和裂解酶的蛋白结构预测分析 1.5.1 跨膜区域和信号肽的预测利用TMHMM Server v.2.0在线分析软件(http://www.cbs.dtu.dk/services/TMHMM/)对蛋白质的跨膜区域进行预测, 以FASTA格式输入功能基因编码蛋白的氨基酸序列, 输出形式选择Extensive, 图形输出(with graphics)。信号肽的预测采用SignalP 4.1服务器(http://www.cbs.dtu.dk/services/SignalP/), 以FASTA格式输入功能基因编码蛋白的氨基酸序列, 全部参数值均选择为默认选项。
1.5.2 蛋白质的二级结构预测将穿孔素和裂解酶基因编码蛋白的氨基酸序列输入在线分析软件ProtParam (http://web.expasy.org/protparam/), 点击Compute parameters, 分析SWH-Ab-1的穿孔素和裂解酶蛋白的理化性质; 同时使用SOPM在线分析软件(https://npsa prabi.ibcp.fr/cgi-bin/npsa_automat.pl)预测蛋白质的二级结构, 结构形式选项(Number of conformational states)选择4, 包括:螺旋(helix)、片状(sheet)、折叠(turn)和卷曲(coil), 其他选项均选择默认参数。
1.6 噬菌体SWH-Ab-1穿孔素和裂解酶的生物功能预测使用ProtFun 2.2 Server (http://www.cbs.dtu.dk/services/ProtFun/)预测蛋白质的生物功能, 以FASTA格式输入功能基因编码蛋白的氨基酸序列, 点击提交。
2 结果 2.1 鲍曼不动杆菌噬菌体的分离与纯化以鲍曼不动杆菌临床分离株为宿主菌, 采用双层琼脂平板培养法从医院污水中分离获得1株噬菌体, 命名为SWH-Ab-1。噬菌体SWH-Ab-1在双层琼脂平板上形成数十个直径为1~2 mm, 边缘光滑、圆形的噬菌斑, 如图 1所示。从图 1中可以看到, 双层琼脂平板上的噬菌斑清晰, 斑内无细菌生长, 表明分离获得的噬菌体SWH-Ab-1能裂解宿主菌, 提示该噬菌体为裂解性噬菌体。
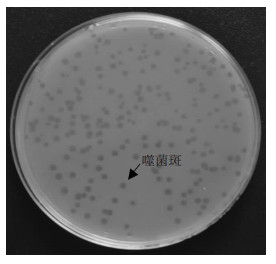
|
| 图 1 以鲍曼不动杆菌为宿主分离出的噬菌体SWH-Ab-1 |
挑取单个噬菌斑用双层琼脂平板进行纯化培养, 经3~5次重复纯化后, 平板上形成的噬菌斑大小较均一、边缘光滑、透明, 表明噬菌体已被纯化。同时测得噬菌体的活性滴度为8×109 PFU/mL。纯化后的噬菌体SWH-Ab-1保存在4 ℃冰箱备用, 并同时保种于中国典型培养物保藏中心(CCTCC No.M2014507)。
2.2 噬菌体SWH-Ab-1的形态特征纯化后的噬菌体SWH-Ab-1用透射电镜进行形态观察, 如图 2所示。电镜下可见噬菌体SWH-Ab-1有一个多面体立体对称的头部, 头部直径约为136 nm; 有一长尾, 尾长约270 nm。根据噬菌体分类方法, 鲍曼不动杆菌噬菌体SWH-Ab-1应属于有尾病毒目, 长尾噬菌体(Siphoviridae)科。
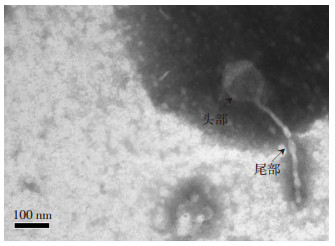
|
| 图 2 噬菌体SWH-Ab-1的电镜照片 |
2.3 噬菌体SWH-Ab-1的基因组DNA纯度及酶切分析
提取噬菌体SWH-Ab-1的基因组DNA, 经qPCR检测所提取的噬菌体DNA中宿主菌的16S rDNA的残留量, 并与同浓度的宿主菌基因组DNA的量比较, 结果显示基因组DNA纯度为96.62%, 满足后续DNA测序分析要求。噬菌体SWH-Ab-1的基因组DNA用限制性核酸内切酶SpeⅠ、NdeⅠ和HindⅢ分别消化, 电泳结果如图 3所示。从图 3中可以看出, 噬菌体SWH-Ab-1的基因组DNA可以被SpeⅠ、NdeⅠ和HindⅢ限制性内切酶切开, 因为所用限制性内切酶只能识别并切开双链DNA, 提示噬菌体SWH-Ab-1的DNA为双链DNA结构, 即dsDNA。
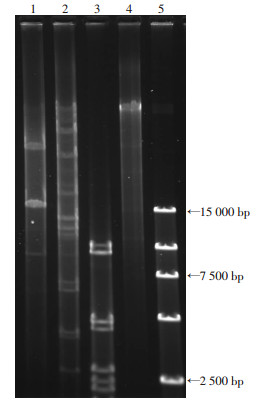
|
| 1:SpeⅠ酶切; 2:NdeⅠ酶切; 3:HindⅢ酶切; 4:噬菌体DNA; 5:DNA标准 图 3 鲍曼不动杆菌噬菌体SWH-Ab-1单酶切电泳结果 |
2.4 噬菌体SWH-Ab-1的基因组DNA序列
纯度合格的噬菌体SWH-Ab-1的基因组DNA, 采用Illumina Hiseq2000对其进行全基因组测序。测序结果经Kmer值参数优化, 拼接完成后, 将噬菌体SWH-Ab-1的基因组DNA序列提交到NCBI的GenBank数据库中, 获取到登录号:MG459218。噬菌体SWH-Ab-1的基因组序列采用CGView软件绘制出噬菌体的全基因组图谱(图 4)。结果显示, 噬菌体SWH-Ab-1的基因组序列大小为41 567 bp, G+C%含量为39.42%。应用BLASTN程序对噬菌体SWH-Ab-1基因组进行同源性分析, 共检索到17条同源序列, 其中与不动杆菌噬菌体vB_ApiP_P1序列间的覆盖率为85%, 同源性为94%。尽管两种不动杆菌噬菌体的基因同源性较高, 但通过噬菌体的形态学比较发现, 噬菌体vB_ApiP_P1为短尾噬菌体, 而噬菌体SWH-Ab-1为长尾噬菌体, 进一步对其基因序列进行分析, 表明两种噬菌体的编码基因所处位置也不相同, 提示噬菌体SWH-Ab-1可能为一种新发现的鲍曼不动杆菌裂解性噬菌体。
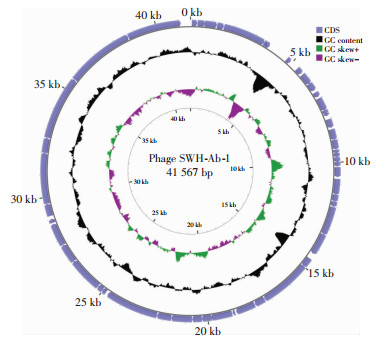
|
| 图 4 鲍曼不动杆菌噬菌体SWH-Ab-1全基因组图谱 |
2.5 噬菌体SWH-Ab-1编码基因预测及重要功能基因定位分析
利用Glimmer 3.0软件进行序列分析, 在噬菌体SWH-Ab-1基因组中共找到51个可能的编码基因, 均位于正链上。将获得的51个编码基因的氨基酸序列采用BLAST程序逐个进行检索分析, 发现ORF02和ORF03开放阅读框可能是噬菌体SWH-Ab-1穿孔素(Bacteriophage SWH-Ab-1 holin, HolSA1)和裂解酶(SWH-Ab-1 endolysin, LysSA1)的编码区。HolSA1基因的开放阅读框位于320~655 bp, LysSA1基因的开放阅读框位于642~1 199 bp, 穿孔素基因位于裂解酶基因的上游(图 5)。
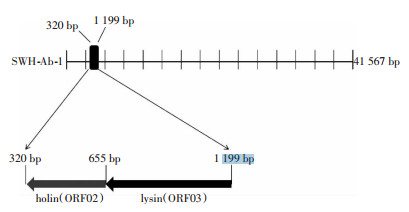
|
| 图 5 HolSA1(ORF02)和LysSA1(ORF03)功能基因位置 |
在NCBI数据库中对噬菌体SWH-Ab-1的功能基因编码蛋白序列进行同源性比对, 发现噬菌体SWH-Ab-1的ORF02与不动杆菌噬菌体vB_AbaP_PD-6A3、Fri1、vB_AbaP_AS11、vB_AbaM_IME200、SH-Ab 15519、vB_AbaP_B5的穿孔素的同源性分别为98%、93%、92%、92%、92%、92%。噬菌体SWH-Ab-1的ORF03与不动杆菌噬菌体vB_AbaP_B5、Fri1、vB_AbaP_CEB1、vB_ApiP_P2、SH-Ab 15519、vB_AbaP_AS11的裂解酶的同源性分别为97%、97%、97%、96%、97%、96%。结果提示, 不同鲍曼不动杆菌噬菌体的穿孔素和裂解酶的基因轻微可能不同, 但基因序列同源性较高。
2.6 HolSA1和LysSA1跨膜区域和信号肽预测使用TMHMM Server在线软件对噬菌体SWH-Ab-1的HolSA1和LysSA1氨基酸序列进行跨膜预测分析, 结果如图 6所示。图中outside、TM helix及inside分别表示膜外区域、跨膜螺旋区及膜内区域。从图 6A中可知, HolSA1蛋白的33~43、94~111位氨基酸肽链处于膜内, 1~9、67~70位氨基酸肽链处于膜外, 10~ 32、44~66、71~93位氨基酸肽链形成了3个典型的跨膜螺旋区。图 6B显示, LysSA1蛋白均处于膜外, 不存在跨膜结构域。结果提示噬菌体SWH-Ab-1的HolSA1蛋白属于Ⅰ型穿孔素蛋白。
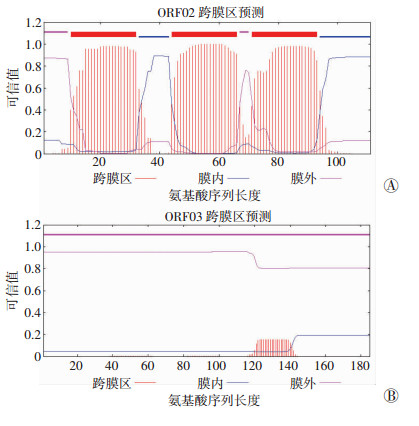
|
| A:HolSA1蛋白的跨膜结构; B:LysSA1蛋白的跨膜结构 图 6 鲍曼不动杆菌噬菌体SWH-Ab-1穿孔素和裂解酶蛋白跨膜区域预测 |
用SignalP 4.1 Server软件对HolSA1蛋白和LysSA1蛋白的信号肽结构进行预测分析, 预测结果如图 7所示。图中的C-score表示剪切位点分值, Y-score表示综合剪切点分值, S-score表示信号肽分值。信号肽剪切位点预测通过Y最大值来判断, 是否为分泌蛋白通过S平均值来判断。结果显示, HolSA1蛋白的S平均值为0.278(< 0.5), LysSA1蛋白的S平均值为0.106(< 0.45), 由此推断噬菌体SWH-Ab-1的穿孔素和裂解酶不存在信号肽, 不属于分泌蛋白。结果提示, 噬菌体SWH-Ab-1需要穿孔素蛋白在细胞膜上形成跨膜孔, 裂解酶才能穿过细胞膜从而发挥裂菌功能。
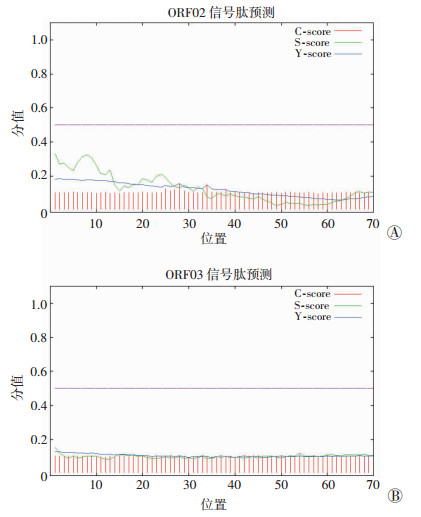
|
| A:HolSA1蛋白信号肽分析; B:LysSA1蛋白信号肽分析 图 7 鲍曼不动杆菌噬菌体SWH-Ab-1穿孔素和裂解酶蛋白信号肽预测 |
2.7 HolSA1和LysSA1蛋白的二级结构预测
使用ProtParam在线软件对HolSA1蛋白和LysSA1蛋白进行理化性质分析, 同时SOPMA预测蛋白的二级结构, 结果表明:HolSA1蛋白由111个氨基酸组成, 相对分子质量为11.957 96×103, 等电点为4.87, 属于稳定的疏水性蛋白, 其二级结构中, α-螺旋、β-折叠、β-转角和无规则卷曲分别占氨基酸总数的66.67%、9.01%、6.31%和18.02%;LysSA1蛋白由185个氨基酸组成, 相对分子质量为21.010 4×103, 等电点为9.56, 属于稳定的亲水性蛋白, 其二级结构中, α-螺旋、β-折叠、β-转角和无规则卷曲分别占氨基酸总数的48.11%、12.43%、13.51%和25.95%。从预测结果可知, 噬菌体SWH-Ab-1穿孔素和裂解酶的二级结构组分中, 以α-螺旋和无规则卷曲为主。
2.8 HolSA1和LysSA1蛋白的功能预测利用蛋白结构功能域及功能分类的ProtFun分析软件对HolSA1和LysSA1蛋白的生物功能进行预测。结果显示, HolSA1可能具有脂肪酸代谢(概率为2.655)、转运载体(概率为2.296)、运输和结合(概率为1.672)、信号转导(概率为1.314)、翻译(概率为1.158)等生物学功能; LysSA1可能具有氨基酸生物合成(概率为10.810)、翻译(概率为5.992)、脂肪酸代谢(概率为4.426)、辅因子生物合成(概率为3.637)、生长因子(概率为2.416)等生物学功能。
3 讨论鲍曼不动杆菌作为一种可长时间存在于诊室、医疗仪器、床单位的物表等医院环境中的条件致病菌, 已成为了医院获得性感染的重要致病菌之一。目前, 碳青霉烯类抗生素是临床治疗鲍曼不动杆菌严重感染的有效手段, 但随着该类抗生素在临床治疗中的大量和(或)不合理使用, 造成了鲍曼不动杆菌耐药菌株越来越多, 包括我国在内的许多国家或地区出现了多重耐药或“泛耐药”的鲍曼不动杆菌, 给临床治疗带来了严峻挑战[8]。因此, 如何防控鲍曼不动杆菌感染已经成为一个严峻和紧迫的临床现实问题。近年来, 裂解性噬菌体作为一类能感染裂解细菌的特殊病毒, 其在细菌感染防控方面的作用又重新进入了人们的研究视野。
本研究以鲍曼不动杆菌临床分离株为宿主菌, 从医院污水中成功分离筛选出1株鲍曼不动杆菌噬菌体, 命名为噬菌体SWH-Ab-1。SWH-Ab-1噬菌体与鲍曼不动杆菌宿主菌在双层琼脂平板上共培养后, 在平板上层形成大小均一、边缘光滑、透明的噬菌斑, 具有典型裂解性噬菌体的特性。噬菌体SWH-Ab-1的头部呈多面体立体对称, 头部直径约为136 nm, 尾长约270 nm。根据噬菌体分类方法, 如噬菌体尾长为头部直径的两倍, 则确定为长尾噬菌体。因此, 本研究分离的鲍曼不动杆菌噬菌体SWH-Ab-1应属于有尾病毒目, 长尾噬菌体(Siphoviridae)科。噬菌体SWH-Ab-1的基因组DNA可以被SpeⅠ、NdeⅠ和HindⅢ限制性内切酶切开, 因为所用限制性内切酶只能识别并切开双链DNA, 结果证实噬菌体SWH-Ab-1的DNA为双链DNA(dsDNA)结构。
噬菌体在抗感染中的应用可以追溯到20世纪70年代。大量实验研究发现, 噬菌体治疗细菌感染具有显著效果, 并且抗菌作用具有高度专一性, 避免了对机体微生态的影响。但噬菌体治疗仍面临诸如噬菌体的细菌抗性、抗菌谱窄、生物安全性、药代动力学等多种挑战。现有研究表明, 裂解酶是裂解性噬菌体感染宿主菌后, 在增殖晚期表达的一类肽聚糖水解酶。该酶通过水解细菌胞壁肽聚糖上糖与肽间的酰胺键或肽内氨基酸残基间的连接键而使细菌裂解。由于裂解酶缺少信号肽结构, 不能穿过细胞膜, 主要聚集在宿主菌的细胞质中, 其裂菌作用必须依靠holin蛋白(穿孔素)的辅助, 通过后者的寡聚化诱导细胞质膜的损伤, 形成跨膜通道, 穿过细胞膜而作用于细胞壁上的肽聚糖靶点。由此可见, 双链DNA噬菌体的裂解效应是holin蛋白和裂解酶协同作用的结果, holin蛋白和裂解酶是噬菌体发挥裂菌作用的重要功能性蛋白质, 成为噬菌体抗菌研究关注的关键基因[9]。
随着基因测序技术的发展, 使得噬菌体等微生物的全基因组测序变得简单易行。本研究采用二代测序技术对鲍曼不动杆菌噬菌体SWH-Ab-1的全基因组序列进行测定, SWH-Ab-1基因组为全长41 567 bp, 目前, 已有多种鲍曼不动杆菌噬菌体的全基因组序列被获得, 但每种噬菌体基因组的大小差异显著, 序列最小的噬菌体只有30 kb左右, 而最大的可长达170 kb, 表明噬菌体基因组具有多样性。通过对噬菌体SWH-Ab-1功能基因的定位分析, 发现ORF02(位于320~655 bp)和ORF03(位于642~1 199 bp)可能是噬菌体SWH-Ab-1穿孔素和裂解酶的编码基因, 分别编码111个氨基酸和185个氨基酸。穿孔素基因位于裂解酶基因的上游, 同时, 两个基因序列之间出现了多个碱基的重叠现象。噬菌体属于病毒类微生物, 结构简单, 基因组DNA序列较短, 重叠基因不仅使噬菌体能经济、高效地利用DNA遗传信息量, 节约碱基, 而且更便于对基因表达起调控作用。穿孔素蛋白之所以能够程序化地破坏宿主细胞膜结构, 精确地控制噬菌体的释放, 可能与这种基因重叠现象有关[10]。因此, 我们推测噬菌体SWH-Ab-1存在“穿孔素-裂解酶”二元裂解系统。此外, 噬菌体基因的同源性分析结果显示, 虽然鲍曼不动杆菌噬菌体SWH-Ab-1与不动杆菌噬菌体vB_ApiP_P1具有较高同源性, 但二者的编码基因所处位置和噬菌体形态不相同, 表明SWH-Ab-1可能是一株新的鲍曼不动杆菌噬菌体。
目前研究发现, 不同噬菌体来源的穿孔素蛋白所形成的跨膜区数量不同, 可分为3种类型[11]。Ⅰ型穿孔素蛋白至少含有90个氨基酸, 具有3个跨膜区, 蛋白的N-端伸向细胞周质, C-端位于细胞质内[12]; Ⅱ型穿孔素蛋白含有65~85个氨基酸, 形成2个跨膜区, N-端和C-端都位于细胞质, 绝大多数的穿孔素属于Ⅰ型和Ⅱ型; Ⅲ型穿孔素只有一个跨膜区, N-端位于细胞质, C-端伸向细胞周质, 只有T4噬菌体和与之有亲缘关系的T-even噬菌体的穿孔素属于Ⅲ型。噬菌体SWH-Ab-1的HolSA1具有3个典型的跨膜螺旋区, 属于Ⅰ型穿孔素蛋白, 是一种疏水性的跨膜蛋白。而LysSA1蛋白不存在跨膜结构域, 均处于膜外, 同时裂解酶缺乏信号肽序列, 不属于分泌蛋白。上述结果提示, 噬菌体SWH-Ab-1与大部分噬菌体的裂解酶一样, 不能够直接穿透宿主菌的细胞膜, 必须借助噬菌体编码的穿孔素蛋白的辅助作用, 才能够穿过宿主细胞膜, 进入周间质进而作用于细胞壁的肽聚糖层, 水解破坏细胞壁, 发挥其裂菌作用。
噬菌体裂解酶具有与细菌共同进化的选择优势和高效的裂解活性, 其作用不受细菌耐药性的影响, 不易诱导细菌产生新的抗性, 而且易于人工设计和基因工程改造。这些特点表明噬菌体裂解酶在临床耐药细菌防控与治疗中具有巨大潜力。近年来, 利用基因工程技术制备噬菌体的特异性裂解酶作为抗菌剂成为噬菌体研究领域的新热点[13-14]。然而, 到目前为止, 裂解酶研究多以革兰阳性细菌的噬菌体为主, 鲍曼不动杆菌(革兰阴性细菌)噬菌体裂解酶研究尚处于起步阶段[15-16]。其原因不仅在于鲍曼不动杆菌外膜的低通透性, 限制了裂解酶对其肽聚糖的降解, 另外一个原因是对鲍曼不动杆菌噬菌体裂解酶的基因组成、蛋白结构等特点还不十分清楚。本研究对噬菌体SWH-Ab-1的穿孔素和裂解酶等功能基因序列进行同源性比对, 发现噬菌体SWH-Ab-1穿孔素和裂解酶的编码基因与多株不动杆菌噬菌体具有高度同源性。结果提示, 噬菌体功能基因定位尽管不同, 但其编码穿孔素和裂解酶的基因序列同源性较高, 编码蛋白质的氨基酸序列、结构和生物功能相似, 为制备噬菌体穿孔素和裂解酶的基因工程制剂提供了可能。
本研究还对噬菌体SWH-Ab-1的裂菌功能基因编码蛋白的氨基酸序列、二级结构以及生物学功能等进行预测。结果发现, HolSA1蛋白是一种等电点为4.87、相对分子量为11.957 96×103的稳定性疏水性蛋白, 二级结构以α-螺旋和无规则卷曲为主, 可能具有脂肪酸代谢、转运载体、运输和结合等生物学功能; LysSA1蛋白相对分子质量为21.010 4×103, 等电点为9.56, 是一种稳定的亲水性蛋白, 二级结构以α-螺旋和无规则卷曲为主, 发挥生物作用的机制更复杂和多样, 可能具有氨基酸生物合成、脂肪酸代谢、辅因子生物合成等生物学功能。上述预测分析结果不仅证实噬菌体SWH-Ab-1是通过“穿孔素-裂解酶”二元裂解系统发挥裂菌作用, 而且为裂解酶的基因工程制备提供了有用的线索。但由于生物信息学是根据已知的噬菌体基因信息, 通过预测分析得到的结果, 可能存在较大的局限性和不确定性, 关于噬菌体SWH-Ab-1的穿孔素蛋白和裂解酶蛋白的生物学功能和性质还需要进一步验证。
| [1] | EVANS B A, HAMOUDA A, AMYES S G. The rise of carbapenem-resistant Acinetobacter baumannii[J]. Curr Pharm Des, 2013, 19(2): 223–238. DOI:10.2174/13816128130204 |
| [2] | FISCHETTI V A. Bacteriophage lytic enzymes:novel anti-infectives[J]. Trends Microbiol, 2005, 13(10): 491–496. DOI:10.1016/j.tim.2005.08.007 |
| [3] |
王铮, 沈文彬, 张浩天, 等. 噬菌体裂解酶作为抗菌药物的研究进展[J].
上海交通大学学报(医学版), 2013, 33(3): 368–373.
WANG Z, SHENG W B, ZHANG H T, et al. Prospects of bacteriophage endolysins as antibacterial agents[J]. J Shanghai Jiaotong Univ, 2013, 33(3): 368–373. DOI:10.3969/j.issn.1674-8115.2013.03.023 |
| [4] | FERNANDES S, SÃO-JOSÉ C. More than a hole:the holin lethal function may be required to fully sensitize bacteria to the lytic action of canonical endolysins[J]. Mol Microbiol, 2016, 102(1): 92–106. DOI:10.1111/mmi.13448 |
| [5] | WON G, HAJAM I A, Lee J H. Improved lysis efficiency and immunogenicity of Salmonella ghosts mediated by co-expression of λ phage holin-endolysin and X174 gene E[J]. Sci Rep, 2017, 7: 45139. DOI:10.1038/srep45139 |
| [6] | FARKASOVSKÁ J, GODÁNY A. The lysis system of the Streptomyces aureofaciens phage mu1/6[J]. Curr Microbiol, 2008, 57(6): 631–637. DOI:10.1007/s00284-008-9255-0 |
| [7] |
李新伊, 刘新. 鲍曼不动杆菌噬菌体的研究进展[J].
中国微生态学杂志, 2015, 27(3): 364–367.
LI X Y, LIU X. The progress in researches on Acinetobacter baumannii phage[J]. Chin J Microecol, 2015, 27(3): 364–367. DOI:10.13381/j.cnki.cjm.201503032 |
| [8] | KARAGEORGOPOULOS D E, FALAGAS M E. Current control and treatment of multidrug-resistant Acinetobacter baumannii infection[J]. Lancet Infect Dis, 2008, 8(12): 751–762. DOI:10.1016/S1473-3099(08)70279-2 |
| [9] | KRUPOVIC M, BAMFORD D H. Holin of bacteriophage lambda:structural insights into a membrane lesion[J]. Mol Microbiol, 2008, 69(4): 781–783. DOI:10.1111/j.1365-2958.2008.06335.x |
| [10] | BARENBOIM M, CHANG C Y, DIB HAJJ F, et al. Characterization of the dual start motif of a class Ⅱ holin gene[J]. Mol Microbiol, 1999, 32(4): 715–727. DOI:10.1046/j.1365-2958.1999.01385.x |
| [11] | PARK T, STRUCK D K, DEATON J F, et al. Topological dynamics of holins in programmed bacterial lysis[J]. Proc Natl Acad Sci USA, 2006, 103(52): 19713–19718. DOI:10.1073/pnas.0600943103 |
| [12] | GRVNDLING A, BLÄSI U, YOUNG R. Biochemical and genetic evidence for three transmembrane domains in the class I holin, lambda S[J]. J Biol Chem, 2000, 275(2): 769–776. DOI:10.1074/jbc.275.2.769 |
| [13] | FENTON M, ROSS P, MCAULIFFE O, et al. Recombinant bacteriophage lysins as antibacterials[J]. Bioeng Bugs, 2010, 1(1): 9–16. DOI:10.4161/bbug.1.1.9818 |
| [14] | SCHMELCHER M, DONOVAN D M, LOESSNER M J. Bacteriophage endolysins as novel antimicrobials[J]. Future Microbiol, 2012, 7(10): 1147–1171. DOI:10.2217/fmb.12.97 |
| [15] | HUANG G, SHEN X, GONG Y, et al. Antibacterial properties of Acinetobacter baumannii phage Abp1 endolysin (PlyAB1)[J]. BMC Infect Dis, 2014, 14: 681. DOI:10.1186/s12879-014-0681-2 |
| [16] | THUMMEEPAK R, KITTI T, KUNTHALERT D, et al. Enhanced antibacterial activity of acinetobacter baumannii bacteriophage ØABP-01 endolysin (LysABP-01) in combination with colistin[J]. Frontiers Microbiol, 2016, 7(681). DOI:10.3389/fmicb.2016.01402 |



