2.400016 重庆, 重庆医科大学药学实验教学中心;
3.400016 重庆,重庆医科大学基础医学院免疫学教研室
2. Laboratory of Immunology, Chongqing Medical University,Chongqing, 400016, China;
3. Center of Pharmacy Experimental Teaching, Chongqing Medical University, Chongqing, 400016, China
地塞米松磷酸钠是临床最常用的地塞米松类药物[1],其在临床应用过程中,药物残液可经各种途径污染环境。自然环境中甾体类激素较难降解,常规的污水处理方法尚不能有效地清除该污染物,国内外多篇论文相继报道了在工厂、医院废水、城市废水、饮用水中均存在不同程度地塞米松类药物污染[2-5]。已有实验证实地塞米松可引起胎兔发育迟缓和上腭腭裂[6],使大鼠骨骼产生骨质疏松[7],模拟环境污染量的地塞米松可引起小鼠精神行为的改变和发育障碍,提示环境地塞米松类药物污染对生物可导致不良影响甚至潜在危害[8]。
微生物降解法是根据微生物适应外环境的代谢多样性,利用自身的代谢过程来降解污染物,运行成本低、无二次污染。由于普通的水处理难以清除水中的甾体类激素,而用化学反应的方法,又可能生成其他的反应物质,有些生成物的毒性甚至比其原有的物质对环境的危害还要大,从而使利用微生物降解甾体类激素日益受到人们的重视[9]。针对环境激素对人类及其他生物的危害,相继从污水处理站的活性污泥、海水、医院废水中分离到降解各种激素的微生物[10],但与降解甾体类激素相关的功能基因及降解途径却仍在探索之中。
本文从医院废水中分离具有降解地塞米松作用的细菌,为探讨该菌对地塞米松的降解作用和降解途径,采用Illumina Hiseq4000结合第三代测序技术对该菌株的基因组测序,并对基因组完成图进行分析,为进一步研究细菌对甾体类激素的降解机制和开发具有应用价值的工程菌奠定基础。
1 材料和方法 1.1 材料分离地塞米松降解菌的废水取自重庆市某医院(29°33’N,106°28’E)净化处理前的污水处理池,废水采集后置-20 ℃冰箱保存。实验中所用培养基均为本实验室配制:① 缺碳培养液:1 000 mL培养液中含K2HPO4·7H2O 1.6 g,KH2PO4 0.4 g,(NH4)2SO4 0.5 g,MgSO4·7H2O 0.2 g,NaCl 0.1 g;② 蔗糖无机盐培养液:在1 000 mL缺碳培养液中加入蔗糖1.0 g;③ 地塞米松磷酸钠无机盐培养液:在缺碳培养液中加入地塞米松磷酸钠,使其终浓度为500 μg/mL,于使用前配制。LB固体培养基干粉为北京鼎国生物有限公司售品,按说明书配制。细菌DNA提取试剂盒(DNA preparation kit)、RT reagent Kit with gDNA Eraser、SYBR Premix Ex Taq Ⅱ购自日本TaKaRa公司;RNAprep Pure Bacteria Kit购自中国北京天根生化科技有限公司(TIANGEN)。
1.2 方法 1.2.1 菌株的富集、分离和驯化参照王毅等[11]的方法,先将医院废水用孔径为80 μm的滤纸片过滤以除掉其中的固体杂质,再取过滤后的废水30 mL,6 000 r/min离心10 min,弃上清液,保留沉淀。取1 mL沉淀加入30 mL地塞米松磷酸钠培养液中,置37 ℃,200 r/min震荡培养5 d,根据菌液是否混浊判断有无细菌生长。用接种环挑取变混浊的地塞米松磷酸钠培养液,分区划线法接种于LB琼脂平板,37 ℃孵箱中培养2 d,观察有无菌落生长。取生长的菌落同时分别接种于10 mL地塞米松磷酸钠培养液和10 mL缺碳培养液中,培养5 d后,取在地塞米松磷酸钠培养液中生长,在缺碳培养液中不能生长的菌落,反复转种于地塞米松磷酸钠培养液,37 ℃孵箱中培养5 d,连续10次进行驯化培养。将驯化培养后的菌液转种地塞米松磷酸钠培养液培养18 h,分装于2 mL EP管中,加甘油封存,保藏于-80 ℃。
1.2.2 分离菌的鉴定将驯化后的菌液培养18 h,分区划线接种于LB琼脂平板,37 ℃孵箱中培养24 h,观察菌落特征,涂片革兰染色观察细菌形态和染色性,扫描电镜观察细菌形态。提取细菌DNA,以此为模板,用27F/1492R通用引物进行16SrDNA PCR扩增,扩增产物送上海美吉公司测序,将所测序列上传至NCBI数据库中进行Blast比对。选取16株相似度最高的16SrDNA序列,用CLUSTAL W进行多序列比对,然后将比对结果用MEGA6.0采用NJ(neighbor-joining)法构建系统进化树,其中NJ树种设置为bootstrap=1 000。
1.2.3 分离菌对地塞米松的降解性能测定参照石中全[5]的方法,将分离并经驯化的菌株转种于地塞米松磷酸钠培养液中,37 ℃,180 r/min震荡培养10 d,在培养的不同时间点取菌液1 mL,6 000 r/min离心10 min,取上清液,以不接种细菌的地塞米松磷酸钠培养液为对照,用高效液相色谱法(HPLC)检测细菌在不同时间点对地塞米松磷酸钠和地塞米松的降解作用。
1.2.4 全基因组测序和分析将提取的DNA送上海美吉生物有限公司用Illumina Hiseq4000结合第三代测序技术对基因组进行测序,先分别构建PE文库(500 bp)和PacBio文库(8~10 kb),再对获得的数据质控后用生物信息学分析及DNAplotter绘制全基因组模式图。将所测的序列分别与Nr、genes、string和GO数据库进行blastp比对,获得COG (Clusters of Orthologous Groups of proteins)(http://www.ncbi.nlm.nih.gov/COG/).)、GO(Gene Ontology)(http://www.geneontology.org/.)、KEGG(Kyoto Encyclopedia of Genes and Genomes)(http://www.genome.jp/kegg/.)等相关功能注释信息。另外,选取4株具有高度同源性并具有特殊代谢能力的Burkholderia菌株,从NCBI数据库中下载其全基因组序列与分离菌比较,绘制系统进化树。
1.2.5 RT-qPCR验证为证实从Burkholderia sp.CQ001基因组中筛选出的可能与降解甾类化合物相关的部分关键基因是否与地塞米松的降解有关,将驯化菌分别接种于地塞米松磷酸钠无机盐培养液(诱导组)和蔗糖无机盐培养液(非诱导组),置37 ℃,200 r/min震荡培养24 h,用RNAprep Pure Bacteria Kit (TIANGEN)提取总RNA,RT reagent Kit with gDNA Eraser(TaKaRa)逆转录合成cDNA,SYBR Premix Ex Taq Ⅱ(TaKaRa)配合Applied Biosystems 7300 Real Time PCR System采用两步法对与降解地塞米松相关的基因进行RT-qPCR扩增。选择在不同宿主中表达稳定的16SrRNA为看家基因,引物序列(表 1)由上海生工设计并合成。运用2-ΔΔCt算法[12]进行相对定量,以比值的形式反映以地塞米松磷酸钠作为底物诱导相对于未用地塞米松磷酸钠诱导的表达差异。
| 基因 | 引物序列(5′→3′) |
| 16SrRNA | 正义GGAGGAAGGCGATAAGGTT |
| 反义ACATCCGACTTGACAGACC | |
| KshA | 正义ATGTCCGCACGCCCTTAC |
| 反义TCACTGCGGCTCGAACAC | |
| KshB1 | 正义ATGAGCGATTCGCGCTTC |
| 反义TCAGTCGGGAAACACCACG | |
| KshB2 | 正义GTGGACAGCCGGGTCAC |
| 反义CTACTTGTCGATCACGAGATCC | |
| ABC | 正义ATGGCAAATCACATGCGTTC |
| 反义TCACTGCCCGGCCGTCG |
2 结果 2.1 地塞米松降解菌的分离和鉴定
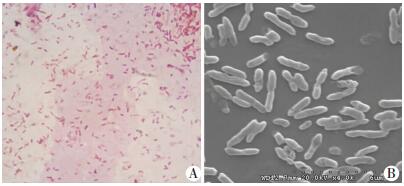
从医院废水中分离到一株能降解地塞米松磷酸钠和地塞米松的细菌,该菌在在LB琼脂平板上形成中等大小(d=1.5~2.0 mm)、圆形、表面光滑湿润、边缘整齐、易挑起的浅黄色不透明菌落。涂片革兰染色后油镜下观察为革兰阴性杆菌,菌体呈直的或略为弯曲的杆状,(6~15) μm×(4~15) μm大小(图 1)。经16s rDNA序列鉴定,初步确定该菌属于伯克霍尔德菌属(Burkholderia),命名为Burkholderia sp.CQ001。通过系统进化树构建发现,在物种进化水平上,该菌株的16SrRNA序列与Burkholderia contaminans J2956的同源性最高。
|
| A:革兰染色×1 000;B:扫描电镜×4 000 图 1 分离菌的菌落特征、形态和染色性 |
2.2 Burkholderia sp.CQ001对地塞米松的降解特性
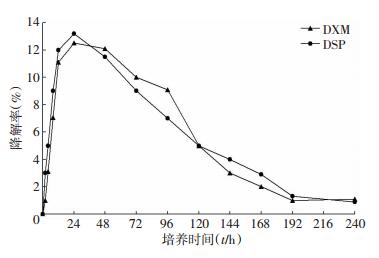
通过HPLC检测发现Burkholderia sp.CQ001能将地塞米松磷酸钠先降解为地塞米松后再将地塞米松降解为其他物质,在pH7.5的地塞米松磷酸钠培养液中,37 ℃震荡培养约24 h出现降解高峰,对地塞米松磷酸钠及地塞米松的总降解率分别为84.8%和77.11%(图 2)。
|
| 图 2 Burkholderia sp.CQ001对地塞米松磷酸钠和地塞米松的降解曲线 |
2.3 Burkholderia sp.CQ001全基因组测序结果
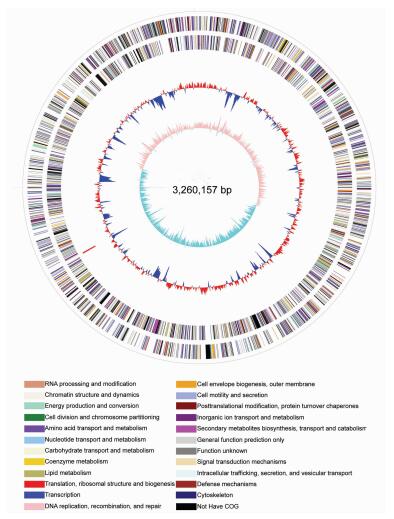
Burkholderia sp.CQ001全基因组测序结果已上传至GenBank,登录号为PRJNA329146。该菌株包含2条染色体和4个巨型质粒,1号染色体基因组全长3 735 298 bp,2号染色体基因组全长3 260 157 bp。4个巨型质粒的基因组长度分别为1 537 783 bp,385 838 bp,97 411 bp,2 873bp。基因组结果的信息统计见表 2所示。其全基因组序列与Burkholderia vietnamiensis G4的亲缘关系最为接近。与代谢相关基因大部分集中在2号染色体上。根据所测基因序列绘制2号染色体的基因组模式图(图 3),其中,第1圈和第2圈为正链、负链上的CDS,不同的颜色表示不同的COG功能分类;第3圈为rRNA和tRNA;第四圈为GC含量;最内1圈为GC skew值。
| 基因组信息 | 数据 |
| 基因数目 | 8 758 |
| 基因总长度 | 7 660 596 bp |
| 基因平均长度 | 874 bp |
| 基因密度 | 0.971基因/kb |
| 基因区域GC含量(%) | 66.9 |
| 基因/基因组(%) | 84.9 |
| 属间区域长度 | 1358764 bp |
| 属间区域GC含量(%) | 61.0 |
| 属间长度/基因组(%) | 15.1 |
|
| 图 3 Burkholderia sp.CQ001的2号染色体基因组模式图 |
2.4 Burkholderia sp.CQ001的基因功能注释和分类
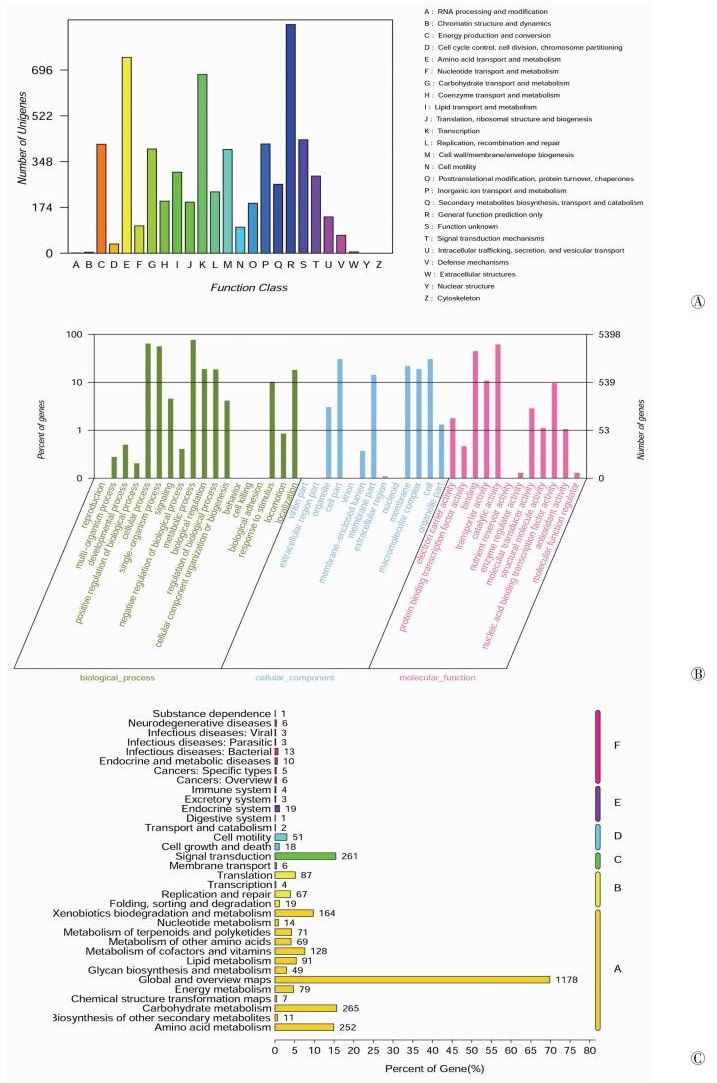
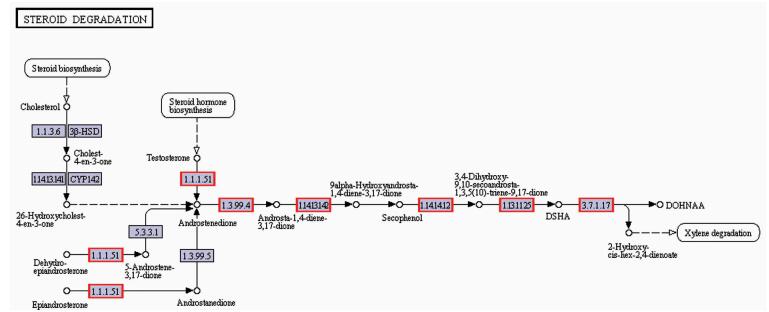
将Burkholderia sp.CQ001基因组中与代谢相关的8758个编码基因与数据库blaste比对后获得COG、GO、KEGG基因功能注释(图 4)。在与COG数据库比对后,有69.8%的编码基因得到功能分类(图 4A),其中,与能量产生和转换(C)相关的基因有408个,与碳水化合物转运和代谢(G)相关的基因有396个,与次生代谢产物合成、运输和分解(Q)相关的基因262个。与KEGG数据库比对分析表明(图 4C),在该菌可进行的117条代谢通路中,涉及一条甾体代谢通路(图 5),其中含有8个与编码甾类化合物代谢相关的摄取转运蛋白和5种降解关键酶的基因(表 3)。以上分析表明Burkholderia sp.CQ001是一株具有强大的代谢功能和代谢途径丰富的细菌,可为进一步研究细菌对甾类化合物的降解机制和开发具有应用价值的工程菌提供丰富的资源。
|
| A: COG功能分类统计图;B: GO功能分类统计图;C:KEGG代谢通路二级分类图 图 4 Burkholderia sp.CQ001基因组功能分类 |
|
| 图 5 Burkholderia sp.CQ001 KEGG甾类化合物代谢通路 |
| 功能 | 降解酶 | 定义 | ORF | 直系同源基因 |
| 摄取转运 | ABC transporter | phospholipid/cholesterol/gamma-HCH transport system substrate-binding protein | orf03369_1 | K02067 |
| 甾核开环 | 3a-HSD | 3(or 17) beta-hydroxysteroid dehydrogenase [EC:1.1.1.51] | orf05510_1 | K05296 |
| KstD | 3-oxosteroid 1-dehydrogenase [EC:1.3.99.4] | orf05595_1 | K05898 | |
| KshA | 3-ketosteroid9alpha-monooxygenase subunit A [EC:1.14.13.142] | orf05568_1 | K15982 | |
| 3-ketosteroid 9alpha-monooxygenase subunit B [EC:1.14.13.142] | orf05593_1 | K15983 | ||
| KshB | putative ferredoxin oxidoreductase protein | orf06355_1 | COG1018 | |
| 开环甾核分解代谢 | HsaA | 3-hydroxy-9, 10-secoandrosta-1, 3, 5(10)-triene-9, 17-dione monooxygenase [EC:1.14.14.12] | orf03896_1 | K16047 |
| HsaB | 3-hydroxy-9, 10-secoandrosta-1, 3, 5(10)-triene-9, 17-dione monooxygenase reductase component [EC:1.5.1.-] | orf05596_1 | K16048 | |
| HsaC | 3, 4-dihydroxy-9, 10-secoandrosta-1, 3, 5(10)-triene-9, 17-dione 4, 5-dioxygenase [EC:1.13.11.25] | orf05585_1 | K16049 | |
| HsaD | 4, 5:9, 10-diseco-3-hydroxy-5, 9, 17-trioxoandrosta-1(10), 2-diene-4-oate hydrolase [EC:3.7.1.17] | orf05553_1 | K16050 | |
| 整体调控 | kstR2 | TetR family regulatory protein | orf00285_1 | COG1309 |
2.5 地塞米松降解相关基因的功能验证
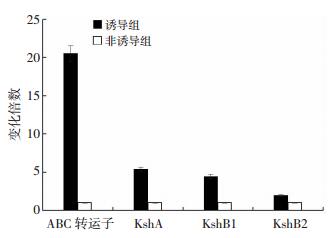
从Burkholderia sp.CQ001基因组中筛选出的可能与降解甾类化合物相关的部分关键基因,根据相关文献阐述的甾体多步降解反应的发生机制[10, 13-14],结合地塞米松的化学结构,推测该菌株在降解地塞米松的过程中,其甾核开环可能仅仅涉及KSH这一双组分酶的作用。用RT-qPCR检测可能涉及地塞米松降解相关基因的表达变化,结果显示,在以地塞米松磷酸钠为碳源的条件下培养24 h,ABC转运子,KshA,KshB表达量不同程度地高于以蔗糖为碳源的培养条件,其中,ABC转运子的增高最为显著,增加20.53倍(图 6)。表明这些基因的表达均参与了地塞米松的降解,推测KshA及KshB1应属于地塞米松强烈诱导型加氧酶及还原酶,而KshB2对地塞米松的刺激没有B1敏感,这可能与KstR2的调控机制有关[15]。
|
| 诱导组:地塞米松磷酸钠;非诱导组:蔗糖 图 6 RT-qPCR检测Burkholderia sp.CQ001相关基因在2种碳源下的相对表达量 |
3 讨论
伯克霍尔德菌(Burkholderia)是一种广泛存在于水、土壤、植物和人体的革兰氏阴性杆菌,该菌具有非常独特的代谢能力,能以200多种有机物为碳源生长繁殖[16],在农业生产和环境保护中受到越来越多研究者的青睐,被冠以“环保卫士”的称号。其中,B.vietnamiensis strain G4可以降解地表水及地下水中具有生物毒性和致癌性的三氯乙烯(TCE),B.xenovorans LB400对工业污水中的多氯联苯(PCBs)具有较强的降解能力,有的菌还能降解致癌物多环芳烃(PAHs),乙二胺四乙酸(EDTA)等[17-19],但尚未见该菌能够降解地塞米松类甾体激素的报道。
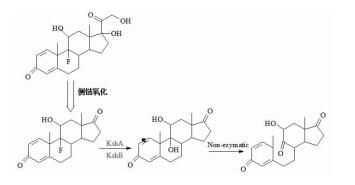
目前,关于甾类物质降解机制探讨,主要以红球菌属和分枝杆菌属为主[20-22]。Burkholderia sp.CQ001能够在以地塞米松磷酸钠为唯一碳源的环境中生长良好,证明该菌能够有效地利用地塞米松类甾体激素。结合相关文献报道,本研究对Burkholderia sp.CQ001的全基因组测序分析和对涉及地塞米松类甾体激素降解的部分关键基因的验证表明,该菌中Mce家族编码的ABC转运子极大可能的参与了地塞米松的主动摄取[23-24];从基因组中未筛选出在甾核开环反应过程中的胆固醇氧化酶(ChO),但筛选出3β-羟基甾体脱氢酶/异构酶(3β-HSD)[25],推测该菌降解甾醇的第一步关键酶为3β-HSD,但是地塞米松类激素属于以甾体为源头代谢后的衍生物,不需要3β-HSD及3-甾酮-Δ1-脱氢酶(KstD)的脱氢与异构;因此,地塞米松甾核开环最关键的一步是3-甾酮-9α-羟基化酶(KSH)作用于地塞米松B环使其羟基化,所生成的9α-羟基-1, 4-二烯甾体极不稳定,较高的分子势能自动引发B环C9(10) 自动裂解(如图 7所示)[26]。KSH由末端加氧酶(KshA)和铁硫蛋白还原酶(KshB)双组分构成,在Burkholderia sp.CQ001中发现KshB存在2个同工酶,即KshB1和KshB2。KshB1应属于地塞米松强烈诱导型还原酶,而KshB2对地塞米松的刺激却没有B1敏感。开环后的甾体由Hsa家族蛋白连续催化裂解可汇入微生物主能量代谢通路,最终分解为ATP与小分子有机盐及其辅酶[27]。KstR2是实现甾类底物条件诱导的关键调节因子,为一种转录抑制型调控蛋白[28]。由于目前关于开环甾核的分解代谢及支链分解代谢和转录调控机制非常复杂,将在后续其他功能基因的研究中深入探讨。
|
| 图 7 地塞米松甾核开环关键步骤及所涉及酶类 |
本研究首次揭示了Burkholderia sp.CQ001具有降解地塞米松类甾体激素的性能,根据全基因组测序及分析初步筛选了该菌降解甾体类化合物的相关基因,并对降解地塞米松类甾体激素的关键编码基因ABC转运子及KSH进行了表达验证,从基因水平证实了该菌具有降解地塞米松类甾体激素的能力,这不仅丰富了伯克霍尔德氏菌的遗传学信息,更为后续深入研究甾体激素的降解机制和降解相关酶提供了信息,为利用生物修复技术清除环境地塞米松类激素污染和利用微生物代谢生产甾类药物提供了理论基础。
| [1] | DONIA M, MANGANO K, QUATTROCCHI C, et al. Specific and Strain-Independent Effects of Dexamethasone in the Prevention and Treatment of Experimental Autoimmune Encephalomyelitis inRodents[J]. ScandinavianJournalofImmunology, 2010, 72(5): 396–407. DOI:10.1111/j.1365-3083.2010.02451.x |
| [2] | CHANG H, HU J, SHAO B. Occurrence of natural and synthetic glucocorticoids in sewage treatment plants and receiving river waters[J]. Environ Sci Technol, 2007, 41(10): 3462–8. DOI:10.1021/es062746o |
| [3] | SC V D L, HERINGA M B, MAN H Y, et al. Detection of multiple hormonal activities in wastewater effluents and surface water, using a panel of steroid receptor CALUX bioassays[J]. Environmental Science & Technology, 2008, 42(15): 5814–20. DOI:10.1021/es702897y |
| [4] | SCHRIKS M, VAN LEERDAM J A, VAN DER LINDEN S C, et al. High-resolution mass spectrometric identification and quantification of glucocorticoid compounds in various wastewaters in the Netherlands[J]. Environ Sci Technol, 2010, 44(12): 4766–4774. DOI:10.1021/es100013x |
| [5] |
石中全, 周裕珍, 杨致邦, 等. 废水中地塞米松污染的探讨[J].
中国医药指南, 2012, 10(9): 319–321.
SHI Z Q, ZHOU Y Z, YANG Z B, et al. Approach the Contamination of Dexamethasone in the Effluent Water[J]. Guide of China Medicine, 2012, 10(9): 319–321. DOI:10.3969/j.issn.1671-8194.2012.09.258 |
| [6] | TAKAHASHI T, NAKAMURA Y, TSUYA A, et al. Pharmacokinetics of aprepitant and dexamethasone after administration of chemotherapeutic agents and effects of plasma substance P concentration on chemotherapy-induced nausea and vomiting in Japanese cancer patients[J]. Cancer Chemotherapy and Pharmacology, 2011, 68(3): 653–659. DOI:10.1007/s00280-010-1519-2 |
| [7] |
卢其腾, 农小连, 吴家昌, 等. 去势联合地塞米松建立大鼠骨质疏松模型的研究[J].
中国骨质疏松杂志, 2015(1): 34–38.
LU Q T, LONG X L, WU J C, et al. Study of the establishment of rat osteoporosis model using ovariectomy combined with dexamethasone[J]. Chinese Journal of Osteoporosis, 2015(1): 34–38. DOI:10.3969/j.issn.1006-7108.2015.01.008 |
| [8] |
杨茜, 李晓愚, 斯丹, 等. 饮水中地塞米松污染对小鼠肠道菌群的影响[J].
南方医科大学学报, 2016(2): 238–243.
YANG Q, LI X Y, SI D, et al. Effect of dexamethasone contamination in drinking water on intestinal flora in mice[J]. Journal of Southern Medical University, 2016(2): 238–243. DOI:10.3969/j.issn.1673-4254.2016.02.16 |
| [9] |
黄卫, 宋海燕. 环境中类固醇雌激素的转化机制研究进展[J].
广东化工, 2009, 36(7): 71–73.
HUANG W, SONG H Y. Process of the Transform Mechanisms of Steroid Estrogens in Environments[J]. Guangdong Chemical Industry, 2009, 36(7): 71–73. DOI:10.3969/j.issn.1007-1865.2009.07.027 |
| [10] | GARCÍ A J L, UHÍA I, GALÁN B. Catabolism and biotechnological applications of cholesterol degrading bacteria[J]. Microbial Biotechnology, 2012, 5(6): 679–699. DOI:10.1111/j.1751-7915.2012.00331.x |
| [11] | WANG Y, YANG Z B, ZHU L L, et al. Isolation and identification of dexamethasone sodium phosphate degrading Pseudomonas alcaligenes[J]. Journal of Basic Microbiology, 2015, 55(2): 262–268. DOI:10.1002/jobm.201300912 |
| [12] | LIVAK K J, SCHMITTGEN T D. Analysis of Relative Gene Expression Data Using Real-Time Quantitative PCR and the 2 -ΔΔCT, Method[J]. Methods, 2001, 25(4): 402–408. DOI:10.1006/meth.2001.1262 |
| [13] | YAM K C, D'ANGELO I, KALSCHEUER R, et al. Studies of a Ring-Cleaving Dioxygenase Illuminate the Role of Cholesterol Metabolism in the Pathogenesis of Mycobacterium tuberculosis[J]. Plos Pathogens, 2009, 5(3): e1000344. DOI:10.1371/journal.ppat.1000344 |
| [14] | OUELLET H, JOHNSTON J B, DE MONTELLANO P R. Cholesterol catabolism as a therapeutic target in Mycobacterium tuberculosis[J]. Trends Microbiol, 2011, 19(11): 530–539. DOI:10.1016/j.tim.2011.07.009 |
| [15] | KENDALL S L, WITHERS M, SOFFAIR C N, et al. A highly conserved transcriptional repressor controls a large regulon involved in lipid degradation in Mycobacterium smegmatis, and Mycobacterium tuberculosis[J]. Molecular Microbiology, 2007, 65(3): 684–699. DOI:10.1111/j.1365-2958.2007.05827.x |
| [16] | LEMAIRE B, CHIMPHANGO S, STIRTON C, et al. Biogeographical patterns of legume-nodulating Burkholderia: from African Fynbos to continental scales[J]. Applied & Environmental Microbiology, 2016, 82(17): 5099–115. DOI:10.1128/AEM.00591-16 |
| [17] | HAMID S, BAE W, KIM S, et al. Enhancing co-metabolic degradation of trichloroethylene with toluene using Burkholderia vietnamiensis G4 encapsulated in polyethylene glycol polymer[J]. Environmental Technology, 2014, 35(12): 1470–1477. DOI:10.1080/09593330.2013.871045 |
| [18] | CHEN W T, SHEN S M, SHU C M. Application of ethylene diamine tetra acetic acid degrading bacterium Burkholderia cepacia on biotreatment process[J]. Bioresource Technology, 2015, 193: 357–362. DOI:10.1016/j.biortech.2015.06.099 |
| [19] | CHAIN P S G, DENEF V J, KONSTANTINIDIS K T, et al. Burkholderia xenovorans LB400 harbors a multi-replicon, 9.73-Mbp genome shaped for versatility[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(42): 15280–15287. DOI:10.1073/pnas.0606924103 |
| [20] | MALAVIYA A, GOMES J. Androstenedione production by biotransformation of phytosterols[J]. Bioresource Technology, 2008, 99(15): 6725–6737. DOI:10.1016/j.biortech.2008.01.039 |
| [21] | SRIPALAKIT P, WICHAI U, Saraphanchotiwitthaya A. Biotransformation of various natural sterols to androstenones by Mycobacterium, sp. and some steroid-converting microbial strains[J]. Journal of Molecular Catalysis B Enzymatic, 2006, 41(1-2): 49–54. DOI:10.1016/j.molcatb.2006.04.007 |
| [22] | VASILEVSKAYA A V, YANTSEVICH A V, SERGEEV G V, et al. Identification of Mycobacterium tuberculosis, enzyme involved in vitamin D and 7-dehydrocholesterol metabolism[J]. Journal of Steroid Biochemistry & Molecular Biology, 2016, S0960-0760(16): 30151-0. DOI:10.1016/j.jsbmb.2016.05.021 |
| [23] | GARCÍA J L, UHÍA I, GARCÍA E, et al. Bacterial degradation of cholesterol and other contaminant steroids[M]// Microbial Bioremediation of Nonmetals: Current Research. 2011:23-43. |
| [24] | MOHN W W, VAN DER GEIZE R, STEWART G R, et al. The Actinobacterial mce4 Locus Encodes a Steroid Transporter[J]. J Biol Chem, 2008, 283(51): 35368–35374. DOI:10.1074/jbc.M805496200 |
| [25] | UHÍA, GALÁN B, MORALES V, et al. Initial step in the catabolism of cholesterol by Mycobacterium smegmatis, mc2155[J]. Environmental Microbiology, 2011, 13(4): 943–959. DOI:10.1111/j.1462-2920.2010.02398.x |
| [26] | DONOVA M V, EGOROVA O V. Microbial steroid transformations: current state and prospects[J]. Applied Microbiology and Biotechnology, 2012, 94(6): 1423–1447. DOI:10.1007/s00253-012-4078-0 |
| [27] | DRESEN C, LIN L Y, D'ANGELO I, et al. A Flavin-dependent Monooxygenase from Mycobacterium tuberculosis Involved in Cholesterol Catabolism[J]. J Biol Chem, 2010, 285(29): 22264–22275. DOI:10.1074/jbc.M109.099028 |
| [28] | UHÍA I, GALÁN B, MEDRANO F J, et al. Characterization of the KstR-dependent promoter of the gene for the first step of the cholesterol degradative pathway in Mycobacterium smegmatis[J]. Microbiology, 2011, 157(9): 2670-80. DOI:10.1099/mic.0.049213-0 |



