2. 400016 重庆,重庆医科大学基础医学院:发育生物学研究室
2. Department of Developmental Biology, College of Basic Medical Sciences, Chongqing Medical University, Chongqing, 400016, China
FAS是肿瘤坏死因子(tumor necrosis factor,TNF)受体超家族成员, 与FAS配体结合介导多种组织中的细胞凋亡。FAS相关蛋白1(FAS-associated protein 1,FAF1) 是FAF1基因编码的一种蛋白质,作为FAS配体受体连接物共同调控多种器官系统的细胞凋亡[1-2]。FAF1编码的蛋白不仅广泛参与胃癌[2]、子宫颈癌[3]和淋巴癌[4]等肿瘤的发生、发展过程,也参与细胞卵裂[5]和神经嵴细胞迁移分化[6]等早期胚胎发育过程。临床研究发现,唇腭裂患者的外周血淋巴细胞中FAF1基因表达显著降低,且在人群7 597例的相关性研究中相对危险度高达1.47[6];动物实验结果显示,在斑马鱼中利用吗啡啉反义寡核苷酸(morpholino,MO)敲低faf1时其颅面软骨整体偏小、舌骨角角度明显增大[6]。体外实验报道,FAF1促进β-catenin泛素化降解抑制wnt/β-catenin信号通路,影响成骨细胞分化和骨形成[7-8]。但利用MO敲低基因的实验技术具有严重的脱靶效应,体外实验仍无法明确faf1突变导致唇腭裂的致病机制,因此faf1基因有待进一步研究。
CRISPR/Cas9是近年来最高效的基因组编辑技术,相对于利用MO敲低基因技术,其脱靶效应更低,可靠性更强[9-12]。FAF1基因在人类与斑马鱼之间具有高度保守性[13]。本研究采用CRISPR/Cas9技术在野生型斑马鱼中敲除faf1基因,对比faf1突变型斑马鱼与野生型斑马鱼的形态差异,研究faf1基因对斑马鱼生长发育的影响。
1 材料与方法 1.1 材料 1.1.1 实验动物野生型斑马鱼(AB品系)。胚胎饲养于holtfreter溶液中,28.5 ℃培养。
1.1.2 主要试剂质粒pXT7-Cas9、质粒pT7-gRNA(中国海洋大学海洋生物多样性与进化研究所赵呈天教授馈赠),XbaⅠ、EcoRⅤ(TaKaRa),2×Taq PCR MasterMix(北京强欣博瑞),Gel Extraction Kit(Lot00D2500010000J11N090,Omega),mMESSAGE mMACHINE® T7 Kit(AM1344,Ambion),MAXIscript T7 Kit(AM1312,Ambion),RNeasy® Mini Kit(Cat No.74104,Qiagen),glycogen(10901393001,Roche),基因组提取试剂盒(Tiangen)。
1.1.3 主要仪器显微注射器,P-97微电极拉制仪(Sutter),毛细玻璃管(Sutter),PCR仪(Bio-Rad),琼脂糖凝胶电泳相关仪器,体视显微镜(Zeiss)。
1.2 突变体模型的制备 1.2.1 gRNA靶位点及鉴定引物设计① gRNA靶位点的设计。根据靶位点预测网站http://chopchop.cbu.uib.no/选择了6个符合靶位点要求的gRNA(表 1),采用T7E1酶切筛选突变体;若邻近靶位点PAM区含有单一特异性酶切位点还可采用限制性内切酶酶切鉴定筛选突变体。② 鉴定引物设计。对6个靶位点的上下游200 bp处分别设计了6对鉴定突变引物(表 2),从准备用于打靶的成鱼中PCR扩增靶点及附近序列,酶切及测序筛选出满足靶点要求的靶位点。若靶位点含有潜在的脱靶位点(off-target),应同样对脱靶位点设计鉴定引物。
| 靶位点 | 序列 | 位置 |
| gRNA1 | GGTGGTAAGACACTATTTGTTGG | 2号外显子与下游内含子交界 |
| gRNA2 | GGTTTTGATTTCACCTGCAGTGG | 5号外显子与上游内含子交界 |
| gRNA3 | GGCACTGTTAAATGGGGAGAGGG | 7号外显子与上游内含子交界 |
| gRNA4 | GGGCACTGTTAAATGGGGAGAGG | 7号外显子与上游内含子交界 |
| gRNA5 | GGAGTGATACCACAAGAAAACGG | 3号外显子上 |
| gRNA6 | 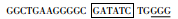 |
6号外显子上 |
| □:EcoR Ⅴ酶切位点;_:PAM序列 | ||
| 靶位点 | 前引物(5′→3′) | 后引物(5′→3′) | 片段大小(bp) |
| gRNA1 | CCCACCCATCGTTATACCTG | CCACAGAATAAAGTGCCAGAT | 603 |
| gRNA2 | GAGGCGGGTCAATAAGGAAAC | ATGCCAACTAATCGTAAGCAAA | 429 |
| gRNA3 | GGAAGTGCCCTCTAGTGGATT | TCTCACCTCCTGGATGGTTTT | 592 |
| gRNA4 | GGAAGTGCCCTCTAGTGGATT | TCTCACCTCCTGGATGGTTTT | 592 |
| gRNA5 | ATCCAGTTATGGGTAGTTGCT | TCTAATCAGATGAGCAGGCAC | 543 |
| gRNA6 | TTGGTCCAGTTGTTTCTC | CATGCCTTCTATGGATGA | 610 |
1.2.2 体外转录合成gRNA及Cas9 mRNA
① gRNA的制备。用前引物pT7-gRNAn(n=1、2、3、4、5、6,表 3)和后引物pT7-gRNA5′-AAAAAAAGCACCGACTCGGTGCCAC-3′,以pT7-gRNA质粒作为模板进行PCR,切胶回收纯化得到gRNA体外转录模板,使用MAXIscript T7试剂盒进行体外转录,1%琼脂糖凝胶电泳确认gRNA被转录出,通过苯酚氯仿异戊醇抽提纯化方法得到可用于显微注射的gRNA原液。② Cas9 mRNA的制备。使用限制性内切酶XbaⅠ线性化pXT7-Cas9质粒,琼脂糖凝胶电泳确认线性化完全后,过柱纯化得到Cas9 mRNA体外转录模板,使用mMESSAGE mMACHINE® T7试剂盒进行体外转录,琼脂糖凝胶电泳确认gRNA被转录出后通过RNeasy® Mini试剂盒纯化得到可用于显微注射的Cas9 mRNA原液。
| 靶位点 | 前引物pT7-gRNAn(n=1、2、3、4、5、6,5′→3′) |
| gRNA1 | TAATACGACTCACTATAGGGTGGTAAGACACTATTTGTGTTTTAGAGCTAGAAATAGC |
| gRNA2 | 靶位点附近含Poly T结构,打滑现象无法准确测序靶位点,该位点不可用 |
| gRNA3 | TAATACGACTCACTATAGGGCACTGTTAAATGGGGAGAGTTTTAGAGCTAGAAATAGC |
| gRNA4 | TAATACGACTCACTATAGGGCACTGTTAAATGGGGAGGTTTTAGAGCTAGAAATAGC |
| gRNA5 | TAATACGACTCACTATAGGAGTGATACCACAAGAAAAGTTTTAGAGCTAGAAATAGC |
| gRNA6 | TAATACGACTCACTATAGGCTGAAGGGGCGATATCTGGTTTTAGAGCTAGAAATAGC |
| _:靶位点序列 | |
1.2.3 显微注射制备F0代斑马鱼并检测gRNA效果
将Cas9 mRNA分别与gRNAn两两混合,终浓度分别为300、120 ng/μL。实验组:显微注射Cas9 mRNA和gRNAn的野生型斑马鱼单细胞胚胎100枚;对照组:未注射的野生型斑马鱼胚胎50枚。受精后第48小时各组分别取30枚胚胎提基因组,利用鉴定引物PCR扩增靶位点及其附近序列,EcoR Ⅴ酶切鉴定gRNA6注射组突变情况,T7E1酶切鉴定gRNA1、gRNA3、gRNA4、gRNA5注射组突变情况,1%琼脂糖凝胶电泳初步检测突变结果。将发生突变的PCR产物送测序,靶位点位置开始出现套峰者即确认靶位点发生突变,从而得到有效的gRNA。
1.2.4 斑马鱼突变体的筛选① 将有效的gRNA和Cas9 mRNA混合物显微注射入200枚野生型斑马鱼单细胞胚胎中,饲养至1个月(F0),剪尾鳍提基因组、PCR扩增靶位点、酶切鉴定出突变的斑马鱼,将其基因组PCR,TA克隆送测序,检测每条鱼体细胞主要的突变类型并在体式显微镜下观察表型并记录。② 将体细胞含有移码突变的F0代斑马鱼个体饲养至性成熟,与野生型斑马鱼交配产生F1代,于受精后48 h提取胚胎基因组(每条F0所产F1代取20枚,10枚/管),PCR扩增靶位点并酶切鉴定,TA克隆送测序,检测可遗传的主要突变类型并在体式显微镜下观察并记录表型。
1.3 阿尔新蓝染色及常见头颅软骨变化指标的测量头颅软骨观察采用阿尔新蓝染色[14];常见头颅软骨变化指标包括梅克尔软骨面积、舌角骨夹角、舌角骨长度、舌角骨尖-侧鳍长度、头颅长度(即梅克尔软骨最前端至侧鳍最前端的距离)。
1.4 数据处理及统计学分析软骨变化指标测量均采用Image J软件分析;各组计量资料以x±s表示,采用SPSS 21.0统计软件进行单因素方差分析。检验水准:α=0.05。
2 结果 2.1 成功制备gRNA和Cas9 mRNA琼脂糖凝胶电泳结果证实,gRNA1、gRNA3、gRNA4、gRNA5、gRNA6(图 1A)以及Cas9 mRNA(图 1B)被转录出,Nanodrop测gRNA浓度分别为300、240、198、230、240 ng/μL;Cas9 mRNA浓度为600 ng/μL。

|
| A:faf1 gRNA1、gRNA3、gRNA4、gRNA5、gRNA6体外转录产物M:DNA标准(DL 2000),1:gRNA1,2:gRNA3,3:gRNA4,4:gRNA5,5:gRNA6;B:Cas9 mRNA体外转录产物M:DNA标准(DL 2000),1:Cas9 mRNA 图 1 琼脂糖凝胶电泳实验鉴定faf1 gRNA和Cas9 mRNA的制备 |
2.2 酶切鉴定及测序筛出gRNA6可导致faf1发生突变
收集经显微注射的F0斑马鱼胚胎及野生型对照组胚胎,提基因组、PCR扩增靶位点、EcoR Ⅴ酶切鉴定gRNA6有效(图 2A),PCR产物测序进一步确认gRNA6有效(图 2B);T7E1酶切鉴定gRNA1、gRNA3、gRNA4、gRNA5均无效。gRNA6排除脱靶后,大量显微注射gRNA6和Cas9 mRNA复合物并收集300枚胚胎,饲养至1个月剪尾鳍,TA克隆筛选出有移码突变的F0代斑马鱼并检测体细胞主要的突变类型,显示靶位点附近有插入、缺失及单个碱基突变(图 2C)。

|
| A:EcoR Ⅴ酶切鉴定gRNA 6靶位点发生突变M:DNA标准(DL 2000),1:野生型斑马鱼组靶位点PCR产物,2:酶切野生型斑马鱼组靶位点PCR产物,3:gRNA6/Cas9 mRNA复合物注射组靶位点PCR产物,4:酶切RNA6/Cas9 mRNA复合物注射组靶位点PCR产物;B:基因组DNA PCR产物测序峰WT:野生型斑马鱼组,MU(MIX):gRNA6/Cas9 mRNA复合物注射组;黑色方框标记gRNA6靶位点序列,黑色虚线标记基因组序列开始突变的位置;C:代表性体细胞突变类型下划线表示靶位点序列;绿色表示PAM序列,红色虚线表示缺失碱基,蓝色表示插入碱基,黄色表示单个突变后的碱基 图 2 测序及EcoR Ⅴ酶切检测gRNA6的作用 |
2.3 获得可遗传的F1代faf1+/-斑马鱼及基因组DNA水平的检测
将体细胞含有移码突变的F0代斑马鱼与野生型外交产F1代,将产生表型的胚胎随机抽取20枚(10枚/组)提取基因组,EcoR Ⅴ酶切鉴定(图 3A)及TA克隆测序发现其基因组DNA均缺失2个碱基(△2)(图 3B),命名为MU1,能够导致下游阅读框移码改变,使6号外显子上编码第199位天冬氨酸D的密码子突变成酪氨酸Y,并于下游225位氨基酸提前出现终止密码子(图 3C),造成整个蛋白质翻译提前终止,从而实现基因敲除的目的。

|
| A:EcoR Ⅴ酶切鉴定F1代有表型组DNA水平M:DNA标准(DL 2000),1:野生型斑马鱼组,2:酶切阳性对照组,模板为F0代测序确认发生突变的基因组,3:酶切有表型组(MU1),右侧箭头标记靶位点目的片段,表示发生突变的条带;B:TA克隆检测F1代有表型组DNA突变类型WT:野生型斑马鱼组,MU1:随机抽取20枚有表型的胚胎提基因组PCR, TA克隆阳性结果(共5个)测序均为该种突变类型;黑色方框标记靶位点序列,蓝色虚线标记基因组序列突变起始位置;C:faf1 MU1突变型氨基酸序列及其所在蛋白序列位置WT:野生型斑马鱼组,MU1:可遗传突变型1,蓝色方框标记氨基酸序列突变起始位置 图 3 鉴定所获得的可遗传faf1突变斑马鱼 |
2.4 faf1突变斑马鱼形态发育
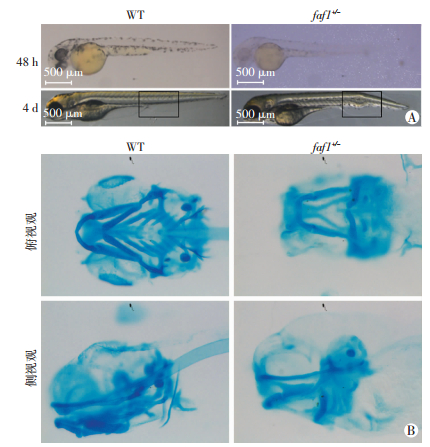
连续对比观察faf1突变型和野生型斑马鱼发现,具有上述突变类型MU1的faf1杂合子斑马鱼受精后30 h之前有明显的发育延迟,达到相同发育时期较野生型斑马鱼晚2~3 h。受精后30 h开始MU1突变型杂合体斑马鱼的色素沉着程度明显小于野生型,受精后48 h眼睛部位未观察到色素沉积(图 4A);受精后4 d发现该种突变型97.3%的F1代斑马鱼躯干邻近尾部出现“结节”,而野生型没有“结节”(图 4A);受精后5 d阿尔新蓝染色显示faf1杂合突变组较野生型组的头颅梅克尔软骨面积缩小,舌角骨夹角增大,舌角骨长度、舌角骨尖-侧鳍长度、头颅长度均缩短(图 4B,表 4);8~9 d,该种突变型斑马鱼死亡。
|
| A:faf1杂合突变型MU1受精48 h及其4 d表型,黑色方框内为“结节”改变部位;B:阿尔新蓝染色显示受精5 d faf1 MU1突变型软骨变化 图 4 faf1杂合突变斑马鱼形态学变化 |
| 组别 | 梅克尔软骨面积(cm2) | 舌角骨夹角(°) | 舌角骨尖侧鳍距离(cm) | 舌角骨长度(cm) | 头颅距离(cm) |
| 野生型组 | 2.45±0.15 | 71.37±0.27 | 5.22±0.14 | 3.75±0.18 | 7.64±0.13 |
| faf1杂合突变型MU1组 | 1.83±0.24a | 126.95±14.69a | 2.11±0.94a | 2.44±0.37a | 5.42±0.72a |
| a:P < 0.01,与野生型组比较 | |||||
3 讨论
本研究采用近年来最高效、可靠的CRISPR/Cas9基因组编辑技术,选择与人类高度同源的斑马鱼作为模式生物,在faf1基因第6个外显子上设计6个gRNA靶点,并最终筛选出1个有效的gRNA。排除gRNA脱靶效应后, 通过特异性酶切及测序筛选出有移码突变的F0代斑马鱼并与野生型斑马鱼外交,获得了可遗传的faf1移码突变F1代杂合体斑马鱼, 其中仅有MU1突变型出现表型并于8~9 d死亡。
对突变体基因型检测发现,每条发生突变的F0代斑马鱼体细胞都至少有2种以上突变类型,即马赛克突变[15];成功敲除的F0代斑马鱼未发现明显的表型,原因很可能在于F0是马赛克突变,未造成明显的faf1表达异常。将发生突变的F0代成鱼与WT外交产生F1代,出现小头、色素沉着延迟、躯干尾部有结节、软骨发育异常的表型,对出现该表型的F1代斑马鱼胚胎随机抽取20枚胚胎提取基因组TA克隆发现基因型确实发生改变,且均为MU1。
对MU1突变型杂合体斑马鱼形态观察发现,除了与之前文献[5-6]报道的利用MO敲低faf1出现的斑马鱼颅面软骨整体偏小、舌骨角角度明显增大的表型类似外,还出现色素沉积迟缓、尾部肌节部位出现“结节型”的肌节异常变化。通过文献[16]总结,在斑马鱼中,神经嵴细胞从神经上皮向外迁移进入咽弓分别形成软骨、神经元、神经胶质和色素细胞。据此推测,faf1可能影响了神经嵴细胞迁移分化成色素细胞的过程,从而导致色素沉积延迟;而结节样表型从未在斑马鱼相关研究中发现过,发生原因有待进一步研究[6]。
综上所述,faf1在胚胎发育过程中是至关重要的。相对于采用MO敲低faf1造成的颅面软骨变化,利用CRISPR/Cas9敲除该基因产生了新的表型,为探究faf1基因功能奠定了基础,也为唇腭裂发病机制的研究提供了新线索。
| [1] | MENGES C W, ALTOMARE D A, TESTA J R. FAS-associated factor 1 (FAF1): diverse functions and implications for oncogenesis[J]. Cell Cycle, 2009, 8(16): 2528–2534. DOI:10.4161/cc.8.16.9280 |
| [2] | CHEN J, GE L, LIU A, et al. Identification of pathways related to FAF1/H. pylori-associated gastric carcinogenesis through an integrated approach based on iTRAQ quantification and literature review[J]. J Proteomics, 2016, 131: 163–176. DOI:10.1016/j.jprot.2015.10.026 |
| [3] | KANG H J, MOON H S, CHUNG H W. The expression of FAS-associated factor 1 and heat shock protein 70 in ovarian cancer[J]. Obstet Gynecol Sci, 2014, 57(4): 281–290. DOI:10.5468/ogs.2014.57.4.281 |
| [4] | BEA S, SALAVERRIA I, ARMENGOL L, et al. Uniparental disomies, homozygous deletions, amplifications, and target genes in mantle cell lymphoma revealed by integrative high-resolution whole-genome profiling[J]. Blood, 2009, 113(13): 3059–3069. DOI:10.1182/blood-2008-07-170183 |
| [5] | ADHAM I M, KHULAN J, HELD T, et al. Fas-associated factor(FAF1) is required for the early cleavage-stages of mouse embryo[J]. Mol Hum Reprod, 2008, 14(4): 207–213. DOI:10.1093/molehr/gan009 |
| [6] | GHASSIBE-SABBAGH M, DESMYTER L, LANGENBERG T, et al. FAF1, a gene that is disrupted in cleft palate and has conserved function in zebrafish[J]. Am J Hum Genet, 2011, 88(2): 150–161. DOI:10.1016/j.ajhg.2011.01.003 |
| [7] | ZHANG L, ZHOU F, LI Y, et al. Fas-associated factor 1 is a scaffold protein that promotes beta-transducin repeat-containing protein (beta-TrCP)-mediated beta-catenin ubiquitination and degradation[J]. J Biol Chem, 2012, 287(36): 30701–30710. DOI:10.1074/jbc.M112.353524 |
| [8] | ZHANG L, ZHOU F, VAN LAAR T, et al. Fas-associated factor 1 antagonizes Wnt signaling by promoting beta-catenin degradation[J]. Mol Biol Cell, 2011, 22(9): 1617–1624. DOI:10.1091/mbc.E10-12-0985 |
| [9] | CHANG N, SUN C, GAO L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos[J]. Cell Res, 2013, 23(4): 465–472. DOI:10.1038/cr.2013.45 |
| [10] | MARESCA M, LIN V G, GUO N, et al. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining[J]. Genome Res, 2013, 23(3): 539–546. DOI:10.1101/gr.145441.112 |
| [11] | O'CONNELL M R, OAKES B L, STERNBERG S H, et al. Programmable RNA recognition and cleavage by CRISPR/Cas9[J]. Nature, 2014, 516(7530): 263–266. DOI:10.1038/nature13769 |
| [12] | RAN F A, HSU P D, WRIGHT J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8(11): 2281–2308. DOI:10.1038/nprot.2013.143 |
| [13] | KIMMEL C B, BALLARD W W, KIMMEL S R, et al. Stages of embryonic development of the zebrafish[J]. Dev Dyn, 1995, 203(3): 253–310. DOI:10.1002/aja.1002030302 |
| [14] | WALKER M B, KIMMEL C B. A two-color acid-free cartilage and bone stain for zebrafish larvae[J]. Biotech Histochem, 2007, 82(1): 23–28. DOI:10.1080/10520290701333558 |
| [15] | BROCAL I, WHITE R J, DOOLEY C M, et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish[J]. BMC Genomics, 2016, 17: 259. DOI:10.1186/s12864-016-2563-z |
| [16] | SCHILLING T F, KIMMEL C B. Segment and cell type lineage restrictions during pharyngeal arch development in the zebrafish embryo[J]. Development, 1994, 120(3): 483–494. |



