2. 400037 重庆,第三军医大学新桥医院:胸外科;
3. 400037 重庆,第三军医大学新桥医院:全军呼吸内科研究所,全军呼吸病研究重点实验室
2. Department of Thoracic Surgery, Xinqiao Hospital, Third Military Medical University, Chongqing, 400037, China;
3. Institute of Respiratory Diseases, Key Laboratory of Respiratory Diseases, Xinqiao Hospital, Third Military Medical University, Chongqing, 400037, China
根据世界卫生组织最新公布的数据显示,肺癌无论是发病率 (180万/年) 还是死亡率 (159万/年) 均位于全世界肿瘤首位[1]。肺癌也是我国发病率和死亡率首位的恶性肿瘤,成为危害健康的主要疾病负担[2]。肺癌中的非小细胞肺癌 (non-small cell cancer,NSCLC) 约占肺癌总数的80%[3]。虽然吸烟是肺癌发生的主要风险因素,但是仅有不超过20%的吸烟者会发生肺癌[4],且具有癌症家族史的非吸烟者发生肺癌的风险也显著增加[5],提示包括遗传在内的其他因素也可能是肺癌发生的风险因素。许多研究表明遗传因素在肺癌的发生、发展中也发挥重要作用[3, 6-7]。单核苷酸多态性 (single nucleotide polymorphism,SNP) 是人类基因组中最常见的可遗传变异[8],人类千人基因组项目已经发现8 500多万个SNPs,其中900多万个SNPs在东亚人群中属于常见变异 (最小等位基因频率,minor allele frequency,MAF>0.01)。
大量基于候选基因的关联研究和全基因组关联研究 (genome-wide association study,GWAS) 已经确证了许多与肺癌风险相关的遗传易感性位点[7, 9],比如CYP1A1 rs1048943,CLPTM1L rs401681,COX-2 rs5275,CHRNA3 rs1051730和AGPHD1 rs8034191位点等。然而,这些研究主要报道的是位于蛋白编码基因上的SNPs位点,对包括微小RNA (microRNA,miRNA) 在内的非蛋白编码基因上的SNPs研究的还较少,绝大多数非蛋白编码基因上SNPs位点尚未进行发掘[10]。
近年来,相关研究发现位于miRNA基因区域内的多态性位点可以影响miRNA的成熟以及miRNA与靶基因的结合能力,并可能与包括肺癌在内的多种癌症的发生及预后相关[10-11]。但是,目前有关miRNA基因多态性与肺癌易感性关系的研究不多。最近有meta分析发现目前只报道了20个位于miRNA基因上的SNPs位点与肺癌发生或预后相关,并且主要关注miRNA-146a rs2910164和miRNA-196a rs11614913位点与肺癌的关系[12]。
为了进一步探讨miRNA基因多态性与NSCLC遗传易感性的关系,本研究基于生物信息学分析系统地筛选位于miRNA基因区域内的潜在功能SNPs位点,并结合病例-对照研究设计,评估所筛选的SNPs位点与NSCLC发生风险之间的关联。
1 资料与方法 1.1 研究对象及样本收集病例组:来源于2012年10月到2015年10月在第三军医大学新桥医院就诊并经病理组织学确诊的626例新发NSCLC患者,年龄 (58.4±9.5) 岁,其中患有其他肿瘤或有其他肿瘤史,或有其他器官合并症者被排除。对照组:736例,年龄 (58.6±12.6) 岁,来自医院同一时期门诊体检人群,纳入对象的性别、年龄 (±5岁) 和居住地 (重庆) 分布与病例组频数匹配,对照组排除标准为:患有肿瘤、肺部疾病史如支气管扩张、哮喘、间质性肺疾病或肺结核以及其他器官严重疾病者。纳入对象均完成了相应的流行病学调查,并抽取研究对象治疗前5 mL外周静脉血,分离单个核细胞,于-80 ℃冰箱保存待用。本研究经第三军医大学伦理委员会审核批准 (2012年),纳入研究对象均签署知情同意书。
1.2 流行病学资料收集研究对象的一般人口学特征和环境暴露史,采用统一设计的调查问卷并由经过专业培训的临床医生进行面访式调查。调查内容包括一般人口学资料、肿瘤家族史、疾病史、吸烟史、饮酒史、饮食史、职业史等。其中每天吸烟1支并持续1年以上者为吸烟者,其余定义为非吸烟者。病例组的临床特征信息来自医院的病案资料,包括诊断日期、病理类型、有无远处转移、临床分期、实验室检测等。
1.3 miRNA基因区域内SNPs位点筛选利用公共数据库结合已知的与肺癌发生及预后相关的miRNA基因,系统性的筛选位于miRNA基因区域内的潜在功能SNPs位点,筛选流程见图 1。基于千人基因组项目 (1000 Genome) 公共数据库 (http://browser.1000genomes.org/index.html) 第三阶段v5b版本提供的SNPs数据,提取东亚人群中常见 (MAF≥1%) SNPs数据信息,进而与Ensemble公共数据库 (http://asia.ensembl.org/index.html) 目前提供的所有miRNA基因数据信息比对,共发现位于miRNA基因区域内的1 470个SNPs位点。
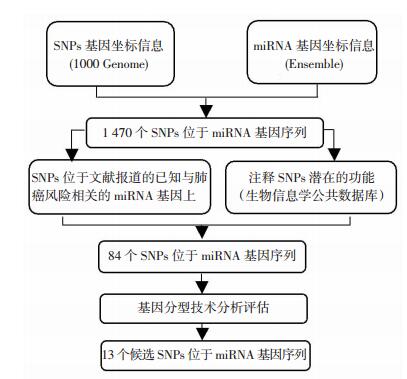
|
| 图 1 筛选位于miRNA基因序列上的单核苷酸多态性位点 |
进一步利用生物信息学资源对初步获取的SNPs位点进行功能注释:① GWAS研究报道的与肿瘤风险显著相关 (P≤10-5) 的SNPs位点以及与这些SNPs存在强连锁不平衡 (linkage disequilibrium,LD;相关系数r2≥0.5) 的SNPs位点 (基于NHGRI GWAS Catalog数据库[13]);② GWAS研究报道的与任何表型显著相关 (P≤10-8) 的SNPs位点 (基于NHGRI GWAS Catalog数据库[13]);③ 位于肺癌细胞株A549的启动子或增强子区域 (基于Encode数据库[14]);④ 位于肺癌细胞株A549的DNA酶I高敏感区 (基于Encode数据库[14]);⑤ 位于进化高度保守区 (基于PhastCons数据库[15]);⑥ 位于miRNA序列的种子区 (seed region,基于targetScanS数据库[16]);⑦ 位于转录因子结合位点 (基于Transfac Matrix数据库[17])。
根据功能注释信息,筛选到了84个具有至少两个潜在功能注释信息或者位于已知的与肺癌风险或预后相关的miRNA基因上的位点。进一步根据筛选位点的基因分型技术SNPscanTM分析评估结果,剔除其中不适合进行进一步基因分型的位点,最终选取13个SNPs位点进行后续的多态性检测。
1.4 基因组DNA的提取应用血液基因组DNA抽提试剂盒DP319(北京天根生化科技有限公司) 从外周血细胞中抽提基因组DNA,定量分装后,于-80 ℃冰箱保存待用。
1.5 miRNA基因区域内的SNPs位点基因分型采用上海天昊生物科技有限公司研发的SNPscanTM技术对筛选的13个SNPs位点进行基因分型。该技术基本原理是采用连接酶连接反应的高特异性实现对SNP位点等位基因的识别,然后通过在连接探针末段引入不同长度的非特异序列以及通过连接酶加接反应获得位点对应的不同长度连接产物,利用标记荧光的通用引物对连接产物进行PCR扩增,通过荧光毛细管电泳对扩增产物进行电泳分离。使用GeneMapper 4.1(Applied Biosystems,USA) 软件读取各位点基因分型原始数据信息。本研究检测的1 362份DNA样本,13个位点的基因分型成功率均为99%,基因分型实验设置的阴性对照组未出现条带,且随机抽取10%样本进行重复检测,结果100%一致。
1.6 统计学分析检查并核对收集的流行病学调查资料和病例资料信息,使用EpiData 3.02软件进行双人、双轨录入数据,建立数据库。采用Stata 12.0软件 (StataCorp 2011,TX,USA) 进行统计分析。病例组和对照组之间人口统计学资料分布差异比较,采用卡方检验 (计数资料) 或t检验 (计量资料)。采用拟合优度χ2检验评估各SNP位点在对照人群中基因型频率分布是否满足哈迪-温伯格平衡 (Hardy-Weinberg equilibrium,HWE),检验分析各SNP位点在病例组与对照组之间的等位基因频率及其基因型频率分布差异。在不同遗传模型 (加性、隐性和显性模型) 下,使用多因素Logistic回归分析计算比值比 (Odds ratio,OR) 及其95%可信区间 (confidence interval,CI),评估各SNP位点与NSCLC发生风险的关联,并校正年龄、性别和吸烟混杂因素。进一步根据病理学类型和吸烟状态分层,分析在不同亚组中各SNP位点与NSCLC风险的关联。所有统计检验均为双侧概率检验,P < 0.05表示差异具有统计学意义。
1.7 SNP位点的连锁不平衡分析鉴于在线LDlink数据库可以进行LD分析,能够对来自1000 Genomes数据库的不同人种样本进行选择,并且可以显示LD plot图,提供具体的关联系数 (r2) 值以及可以关联UCSC公共数据库 (http://genome.ucsc.edu/) 查看关联区域内的相关基因信息。因此,本研究采用LDlink在线分析数据库 (https://analysistools.nci.nih.gov/LDlink/) 筛查与关联分析识别的阳性位点 (具有统计学意义的位点) 存在LD的位点,进一步查看关联区域内的相关基因信息。
2 结果 2.1 研究对象的基本信息本研究包括了626例肺癌患者 (病例组) 和736例健康对照 (对照组),表 1显示了两组研究对象的基本信息。其中年龄和性别在病例组和对照组中分布差异无统计学意义 (P>0.05)。病例组中吸烟者比例显著高于对照组 (P < 0.001)。病例组中肺癌组织学类型包括腺癌356例,鳞癌251例,腺鳞癌17例以及大细胞癌2例。
| 变量 | 病例组 (n=626) | 对照组 (n=736) | P值 |
| 年龄 (岁) | 0.196 | ||
| < 60 | 343(50.16) | 395(53.67) | |
| ≥60 | 366(49.84) | 341(46.33) | |
| 性别 | 0.439 | ||
| 男 | 445(71.09) | 509(69.16) | |
| 女 | 181(28.91) | 227(30.84) | |
| 肺癌家族史 | < 0.001 | ||
| 有 | 47(7.56) | 7(0.95) | |
| 无 | 575(92.44) | 729(99.05) | |
| 吸烟状态 | < 0.001 | ||
| 吸烟 | 445(71.43) | 310(42.12) | |
| 不吸烟 | 178(28.57) | 426(57.88) |
2.2 SNPs位点筛选结果及哈迪-温伯格平衡检验
基于生物信息学分析选取了13个潜在功能的SNPs位点进行后续的关联分析。表 2显示了这些SNPs位点的基本信息,包括rs号、所在miRNA基因、染色体位置、等位基因频率等。对照组位点的基因型分布频率均符合HWE平衡 (P>0.05,表 2)。
| SNP ID | miRNA基因a | 染色体位置 | 碱基改变 | MAFb | HWE检验 | ||
| 病例组 | 对照组 | 1000 Genome数据库 | |||||
| rs11597888 | AL391839.1 | 10p11.22 | G>A | 0.24 | 0.23 | 0.21 | 0.141 |
| rs12803915 | MIR612 | 11q13.1 | G>A | 0.14 | 0.14 | 0.14 | 0.819 |
| rs16867808 | AL021918.2 | 6p22.1 | T>C | 0.09 | 0.10 | 0.11 | 0.164 |
| rs2292879 | MIR6811 | 2q37.3 | A>G | 0.31 | 0.31 | 0.31 | 0.674 |
| rs45530340 | MIR6084 | 1p36.12 | C>T | 0.16 | 0.14 | 0.13 | 0.539 |
| rs5997893 | MIR3928 | 22q12.2 | G>A | 0.51 | 0.49 | 0.50 | 0.407 |
| rs61747536 | AC109460.1 | 16p11.2 | C>T | 0.26 | 0.25 | 0.24 | 0.479 |
| rs62085660 | AC145343.1 | 17q24.2 | C>G | 0.28 | 0.30 | 0.29 | 0.232 |
| rs6464546 | MIR6892 | 7q34 | G>A | 0.18 | 0.18 | 0.17 | 0.704 |
| rs6717413 | MIR7845 | 2q33.3 | A>G | 0.13 | 0.14 | 0.10 | 0.142 |
| rs7247237 | MIR3188 | 19p13.11 | C>T | 0.44 | 0.44 | 0.46 | 0.946 |
| rs745666 | MIR3615 | 17q25.1 | G>C | 0.49 | 0.46 | 0.40 | 0.364 |
| rs999665 | AC021066.1 | 12q13.13 | G>A | 0.30 | 0.29 | 0.31 | 0.615 |
| a:5个新的miRNA基因对应的Ensemble数据库 (http://asia.ensembl.org/index.html) 基因编号:AL391839.1(ENSG00000222309)、AL021918.2(ENSG00000280950)、AC109460.1(ENSG00000281146)、AC145343.1(ENSG00000278219)、AC021066.1(ENSG00000281415);b:MAF (minor allele frequency, 最小等位基因频率):其中病例组和对照组的MAF数据来自研究所检测的样本人群;千人基因组 (1000 Genome) 数据库的MAF数据来自东亚人种 | |||||||
2.3 筛选的SNPs位点基因型分布频率及其与NSCLC易感性的关系
分析所选取的SNPs位点的基因型分布及其与NSCLC风险的关系。结果显示,病例组与对照组中各SNP位点基因型频率分布差异无统计学意义 (P>0.05)。进一步分别基于加性、显性和隐性遗传模型,采用多因素Logistics回归分析并校正年龄、性别和吸烟因素,评估所检测的SNPs位点与肺癌发生风险之间的关联。结果显示,所检测的位于miRNA基因上的13个SNPs位点与肺癌整体上发生风险均无显著性关联 (P>0.05),见表 3。
| SNP ID | 碱基改变 (A1>A2)a | 基因型分布b | P值 | 加性模型 (A1 vs A2) | 显性模型 (A1/A2+A2/A2 vs A1/A1) | 隐性模型 (A2/A2 vs A1/A2+A1/A1) | ||||||
| 病例组 | 对照组 | OR(95%CI) | P值 | OR(95%CI) | P值 | OR(95%CI) | P值 | |||||
| rs11597888 | G>A | 359/222/35 | 445/244/45 | 0.554 | 1.03(0.86~1.25) | 0.720 | 1.09(0.87~1.37) | 0.452 | 0.86(0.53~1.38) | 0.524 | ||
| rs12803915 | G>A | 453/154/9 | 539/180/14 | 0.810 | 1.01(0.80~1.28) | 0.914 | 1.05(0.81~1.35) | 0.731 | 0.71(0.29~1.72) | 0.447 | ||
| rs16867808 | T>C | 519/89/8 | 596/127/11 | 0.336 | 0.82(0.63~1.07) | 0.149 | 0.81(0.60~1.09) | 0.171 | 0.67(0.26~1.75) | 0.417 | ||
| rs2292879 | A>G | 297/260/59 | 345/320/69 | 0.876 | 0.95(0.80~1.13) | 0.594 | 0.91(0.73~1.14) | 0.410 | 1.04(0.71~1.53) | 0.827 | ||
| rs45530340 | C>T | 437/160/17 | 536/184/13 | 0.410 | 1.09(0.87~1.36) | 0.460 | 1.05(0.82~1.35) | 0.692 | 1.67(0.78~3.60) | 0.189 | ||
| rs5997893 | G>A | 142/323/151 | 187/378/169 | 0.555 | 1.13(0.96~1.33) | 0.154 | 1.20(0.92~1.56) | 0.174 | 1.14(0.87~1.48) | 0.343 | ||
| rs61747536 | C>T | 330/247/39 | 404/286/44 | 0.859 | 1.07(0.89~1.28) | 0.499 | 1.08(0.86~1.35) | 0.527 | 1.10(0.69~1.76) | 0.683 | ||
| rs62085660 | C>G | 324/237/55 | 361/297/75 | 0.431 | 0.88(0.74~1.04) | 0.133 | 0.84(0.67~1.05) | 0.119 | 0.87(0.59~1.28) | 0.479 | ||
| rs6464546 | G>A | 414/182/19 | 492/216/26 | 0.900 | 0.96(0.78~1.18) | 0.696 | 0.96(0.76~1.22) | 0.742 | 0.90(0.48~1.69) | 0.744 | ||
| rs6717413 | A>G | 474/127/15 | 546/166/19 | 0.627 | 0.87(0.69~1.09) | 0.227 | 0.86(0.66~1.11) | 0.244 | 0.79(0.38~1.63) | 0.520 | ||
| rs7247237 | C>T | 190/302/122 | 230/362/141 | 0.955 | 1.00(0.85~1.17) | 0.986 | 0.99(0.78~1.26) | 0.926 | 1.01(0.76~1.34) | 0.939 | ||
| rs745666 | G>C | 158/318/140 | 207/377/150 | 0.443 | 1.08(0.92~1.28) | 0.330 | 1.09(0.85~1.41) | 0.485 | 1.13(0.86~1.49) | 0.374 | ||
| rs999665 | G>A | 299/261/56 | 367/308/59 | 0.743 | 0.99(0.83~1.18) | 0.915 | 0.97(0.77~1.21) | 0.776 | 1.06(0.71~1.58) | 0.792 | ||
| a:A1为主要等位基因;A2为次要等位基因 (即在对照组中等位基因频率相对较小的等位基因);b:野生型纯合子/杂合子/变异型纯合子 (A1A1/A1A2/A2A2) | ||||||||||||
2.4 筛选的SNPs位点与NSCLC发生风险关联的分层分析
根据病理类型进行分层分析,发现rs62085660(C/G) 位点的等位基因G具有显著降低肺鳞癌发生风险的效应 (校正OR=0.79,95%CI=0.62~1.00,P=0.049),其中与CC基因型者相比,等位基因G携带者 (即CG和GG基因型者) 罹患肺癌的风险降低28%(校正OR=0.72,95%CI=0.53~0.98,P=0.035),见表 4。但未观察到该位点与肺腺癌发生风险相关。其他位点在两个肺癌病理亚型中,均未观察到与肺癌发生风险相关。
| SNP ID (A1>A2)a | 对照组b | 腺癌 | ||||||||
| 病例组b | 加性模型 (A1 vs A2) | 显性模型 (A1/A2+A2/A2 vs A1/A1) | 隐性模型 (A2/A2 vs A1/A2+A1/A1) | |||||||
| 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | |||||
| rs11597888(G>A) | 445/244/45 | 194/135/20 | 1.16(0.93~1.44) | 0.202 | 1.28(0.97~1.69) | 0.076 | 0.89 (0.50~1.58) | 0.685 | ||
| rs12803915(G>A) | 539/180/14 | 263/81/5 | 0.94(0.71~1.25) | 0.662 | 0.95(0.70~1.31) | 0.771 | 0.70 (0.24~2.08) | 0.524 | ||
| rs16867808(T>C) | 596/127/11 | 296/49/4 | 0.76(0.55~1.05) | 0.100 | 0.75(0.52~1.09) | 0.132 | 0.51(0.15~1.72) | 0.277 | ||
| rs2292879(A>G) | 345/320/69 | 166/150/33 | 0.94(0.76~1.16) | 0.549 | 0.91(0.69~1.19) | 0.476 | 0.97(0.61~1.56) | 0.910 | ||
| rs45530340(C>T) | 536/184/13 | 254/86/9 | 1.06(0.81~1.39) | 0.675 | 1.02(0.75~1.38) | 0.915 | 1.72(0.68~4.35) | 0.253 | ||
| rs5997893(G>A) | 187/378/169 | 71/189/89 | 1.18(0.97~1.44) | 0.107 | 1.33(0.96~1.85) | 0.086 | 1.15(0.84~1.59) | 0.377 | ||
| rs61747536(C>T) | 404/286/44 | 186/141/22 | 1.09(0.88~1.36) | 0.435 | 1.12(0.85~1.47) | 0.433 | 1.11 (0.63~1.94) | 0.719 | ||
| rs62085660(C>G) | 361/297/75 | 178/138/33 | 0.92(0.75~1.13) | 0.418 | 0.89(0.68~1.17) | 0.405 | 0.91(0.57~1.44) | 0.691 | ||
| rs6464546(G>A) | 492/216/26 | 237/102/10 | 0.93(0.72~1.20) | 0.575 | 0.94(0.71~1.26) | 0.700 | 0.76(0.35~1.66) | 0.488 | ||
| rs6717413(A>G) | 546/166/19 | 266/75/8 | 0.87(0.66~1.15) | 0.330 | 0.85(0.62~1.17) | 0.328 | 0.83(0.35~1.99) | 0.677 | ||
| rs7247237(C>T) | 230/362/141 | 106/170/73 | 1.03(0.75~1.25) | 0.754 | 1.01(0.75~1.36) | 0.932 | 1.08 (0.77~1.51) | 0.651 | ||
| rs745666(G>C) | 207/377/150 | 90/180/79 | 1.10(0.90~1.33) | 0.358 | 1.13(0.83~1.53) | 0.440 | 1.13(0.81~1.57) | 0.472 | ||
| rs999665(G>A) | 367/308/59 | 167/151/31 | 1.04(0.84~1.29) | 0.717 | 1.04(0.79~1.37) | 0.760 | 1.07 (0.66~1.74) | 0.778 | ||
| SNP ID (A1>A2)a | 鳞癌 | |||||||||
| 病例组b | 加性模型 (A1 vs A2) | 显性模型 (A1/A2+A2/A2 vs A1/A1) | 隐性模型 (A2/A2 vs A1/A2+A1/A1) | |||||||
| 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | |||||
| rs11597888(G>A) | 154/80/14 | 1.00(0.78~1.28) | 0.990 | 1.01(0.74~1.38) | 0.930 | 0.93(0.49~1.78) | 0.834 | |||
| rs12803915(G>A) | 175/69/4 | 1.11(0.82~1.49) | 0.514 | 1.17(0.84~1.64) | 0.355 | 0.70(0.22~2.21) | 0.543 | |||
| rs16867808(T>C) | 206/38/4 | 0.84(0.60~1.20) | 0.340 | 0.82(0.55~1.22) | 0.325 | 0.83(0.25~2.71) | 0.756 | |||
| rs2292879(A>G) | 119/104/25 | 0.98(0.78~1.24) | 0.871 | 0.93(0.69~1.26) | 0.631 | 1.12(0.68~1.86) | 0.651 | |||
| rs45530340(C>T) | 169/69/8 | 1.20(0.90~1.61) | 0.210 | 1.18(0.85~1.64) | 0.323 | 1.84(0.72~4.70) | 0.202 | |||
| rs5997893(G>A) | 67/122/59 | 1.08(0.87~1.34) | 0.496 | 1.04(0.74~1.48) | 0.803 | 1.17(0.82~1.67) | 0.383 | |||
| rs61747536(C>T) | 133/98/17 | 1.08(0.84~1.38) | 0.539 | 1.08(0.80~1.46) | 0.624 | 1.18(0.65~2.17) | 0.586 | |||
| rs62085660(C>G) | 140/89/19 | 0.79(0.62~1.00) | 0.049 | 0.72(0.53~0.98) | 0.035 | 0.80(0.46~1.38) | 0.424 | |||
| rs6464546(G>A) | 165/74/8 | 0.96(0.73~1.26) | 0.763 | 0.95(0.69~1.31) | 0.760 | 0.95(0.41~2.20) | 0.905 | |||
| rs6717413(A>G) | 192/49/7 | 0.85(0.62~1.15) | 0.296 | 0.83(0.58~1.19) | 0.317 | 0.75(0.29~1.96) | 0.556 | |||
| rs7247237(C>T) | 80/122/45 | 0.92(0.74~1.15) | 0.465 | 0.91(0.66~1.25) | 0.548 | 0.89(0.60~1.31) | 0.554 | |||
| rs745666(G>C) | 65/125/58 | 1.10(0.88~1.36) | 0.407 | 1.13(0.80~1.58) | 0.499 | 1.13(0.79~1.63) | 0.502 | |||
| rs999665(G>A) | 123/100/25 | 0.95(0.75~1.20) | 0.661 | 0.86(0.64~1.17) | 0.346 | 1.18(0.71~1.98) | 0.520 | |||
| a:A1为主要等位基因;A2为次要等位基因 (即在对照组中等位基因频率相对较小的等位基因);b:研究对象基因型分布:野生型纯合子/杂合子/变异型纯合子 (A1A1/A1A2/A2A2) | ||||||||||
根据吸烟状态进行分层分析,结果显示在吸烟人群中,rs11597888(G/A) 位点与肺癌的发生风险显著相关 (P=0.033)。其中,与携带GG基因型者比较,携带至少1个A等位基因 (即GA和GG基因型) 个体罹患肺癌的风险增加42%(95%CI=1.03~1.96)。在吸烟者中,rs16867808(T/C) 位点在加性模型 (校正OR=0.58,95%CI=0.40~0.84,P=0.004) 和显性模型 (校正OR=0.53,95%CI=0.35~0.81,P=0.004) 下都与肺癌的易感性显著相关,且具有降低肺癌发生风险的效应 (表 5)。然而,在非吸烟者中,均未观察到这两个位点与肺癌易感性显著相关。其他位点在吸烟人群和非吸烟人群中,与肺癌易感性之间的关联均无统计学意义。
| SNP ID (A1>A2)a | 病例组b | 对照组b | 相加模型 (A1 vs A2) | 显性模型 (A1/A2+A2/A2 vs A1/A1) | 隐性模型 (A2/A2 vs A1/A2+A1/A1) | |||||
| 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | 校正OR(95%CI) | P值 | |||||
| rs11597888(G>A) | 242/165/30 | 197/94/18 | 1.29(0.99~1.67) | 0.058 | 1.42(1.03~1.96) | 0.033 | 1.17(0.61~2.24) | 0.635 | ||
| rs12803915(G>A) | 330/101/6 | 226/74/8 | 0.83(0.60~1.15) | 0.275 | 0.85(0.59~1.23) | 0.388 | 0.51(0.16~1.67) | 0.266 | ||
| rs16867808(T>C) | 377/53/7 | 240/62/7 | 0.58(0.40~0.84) | 0.004 | 0.53(0.35~0.81) | 0.004 | 0.48(0.13~1.71) | 0.259 | ||
| rs2292879(A>G) | 211/185/41 | 135/146/28 | 0.88(0.69~1.12) | 0.302 | 0.82(0.59~1.12) | 0.210 | 0.96(0.55~1.68) | 0.896 | ||
| rs45530340(C>T) | 298/124/13 | 224/80/4 | 1.35(0.99~1.83) | 0.059 | 1.30(0.92~1.84) | 0.134 | 2.91(0.92~9.23) | 0.070 | ||
| rs5997893(G>A) | 98/234/105 | 87/157/65 | 1.17(0.93~1.47) | 0.191 | 1.26(0.88~1.81) | 0.205 | 1.18(0.80~1.73) | 0.401 | ||
| rs61747536(C>T) | 231/180/26 | 175/116/18 | 1.15(0.89~1.49) | 0.280 | 1.21(0.88~1.66) | 0.242 | 1.11(0.57~2.15) | 0.754 | ||
| rs62085660(C>G) | 230/166/41 | 143/137/28 | 0.82(0.64~1.05) | 0.108 | 0.75(0.55~1.03) | 0.074 | 0.86(0.49~1.52) | 0.612 | ||
| rs6464546(G>A) | 289/136/11 | 202/95/12 | 0.90(0.67~1.20) | 0.474 | 0.95(0.68~1.32) | 0.742 | 0.52(0.20~1.35) | 0.181 | ||
| rs6717413(A>G) | 333/94/10 | 230/66/11 | 0.86(0.63~1.18) | 0.358 | 0.86(0.59~1.24) | 0.415 | 0.72(0.29~1.81) | 0.486 | ||
| rs7247237(C>T) | 135/213/87 | 93/154/62 | 1.01(0.80~1.26) | 0.963 | 0.98(0.70~1.38) | 0.907 | 1.04(0.71~1.54) | 0.830 | ||
| rs745666(G>C) | 103/239/95 | 88/154/67 | 1.07(0.85~1.34) | 0.569 | 1.24(0.87~1.78) | 0.239 | 0.94(0.64~1.38) | 0.757 | ||
| rs999665(G>A) | 209/187/41 | 134/144/31 | 0.86(0.67~1.09) | 0.214 | 0.80(0.58~1.10) | 0.176 | 0.88(0.52~1.51) | 0.649 | ||
| a:A1为主要等位基因;A2为次要等位基因 (即在对照组中等位基因频率相对较小的等位基因);b:研究对象基因型分布:野生型纯合子/杂合子/变异型纯合子 (A1A1/A1A2/A2A2) | ||||||||||
2.5 SNP位点间连锁不平衡分析
通过LDlink在线数据库,基于1000 Genomes数据库中东亚人群样本的基因分型数据,识别与本研究关联分析发现的3个阳性关联SNPs位点 (rs62085660 C/G,rs11597888 G/A和rs16867808 T/C位点) 存在强连锁不平衡的位点 (r2 ≥ 0.8)。其中与rs62085660位点存在强连锁不平衡的位点所在基因坐标区域对应的基因是LINC00674和KPNA2基因。与rs11597888位点存在强连锁不平衡的位点所在基因坐标区域对应的基因是EPC1和LOC102031319。与rs16867808位点存在强连锁不平衡的位点所在基因坐标区域对应的基因主要是ZNF391、ZNF184、PR1-153G14.4和TRNAI6基因。这些基因既包括蛋白编码基因也包括非蛋白编码基因。
3 讨论miRNA是一类内源性长约22个核苷酸 (nucleotide,nt) 的非蛋白编码RNA分子[18]。其在基因表达调控网络中起着重要作用,通过与特定的靶信使RNA (mRNA) 结合,有效地抑制mRNA的翻译或者使mRNA降解,进而调控基因的表达[18]。miRNAs在恶性肿瘤 (包括肺癌) 发生、发展中的作用越来越受到关注,目前已发现许多与肺癌的发生、发展及预后相关的miRNAs生物标志物[19]。
近年来,位于miRNA基因序列上的多态性位点也报道与肺癌的易感性和预后相关。Xie等[20]发现has-mir-30a rs763354 SNP位点和位于miR-200b/200a/429基因簇上的rs9660710 SNP位点与NSCLC的发生风险显著相关。有研究发现pri-let-7a-2 rs1143770位点影响NSCLC患者的生存[21]。研究报道,has-mir-196a2 rs11614913位点与肺癌的易感性和预后均相关[22-23]。has-mir-499 rs3746444 SNP位点影响肺癌患者的预后,进一步的体内外实验表明该位点影响肿瘤细胞的增殖和肿瘤组织的生长[24]。这些研究提示,miRNA基因上的功能性SNP可能在肺癌的发生发展中起作用,然而,目前发现的与肺癌易感性相关的miRNA基因上的SNPs位点尚属于少数,值得进一步研究。
本研究将生物信息学分析与病例-对照研究设计结合,系统性的筛选位于miRNAs基因序列上具有潜在功能的SNPs位点并探索其与NSCLC易感性的关联。最终识别了3个新的与NSCLC遗传易感性相关的SNPs位点,分别是rs62085660(C/G),rs11597888(G/A) 和rs16867808(T/C) 位点。然而所观察到的关联效应只局限于特定的亚组人群。
rs62085660(C/G) 位点的等位基因G只降低肺鳞癌的发生风险,而与肺腺癌的发生风险无关。肺鳞癌和肺腺癌是NSCLC的两种主要病理组织学类型,两者在临床表征以及流行病学特征上存在很大的差异。之前文献报道[25]两种肺癌病理亚型之间的遗传学背景也存在差异,本研究的结果与之一致。rs62085660(C/G) 位点位于miRNA基因AC145343.1上,该基因位于染色体17q24区域,包括102碱基。rs62085660(C/G) 位点之前没有报道过,且基因AC145343.1是由芯片预测出的新miRNA基因,因而还不清楚所观察到的关联潜在的生物学机制。我们通过生物信息学功能数据库注释,发现该位点位于启动子和增强子区,推测该位点可能影响miRNA基因AC145343.1的表达量。
rs11597888(G/A) 位点和rs16867808(T/C) 位点与肺癌易感性的关联局限于吸烟人群。提示该位点可能只影响吸烟相关性肺癌的发生风险,通过其遗传效应调控吸烟机体对肺癌的易感性。这与流行病学调查报道的吸烟个体之间罹患肺癌风险差异的结果一致,说明多态性位点可能影响个体对烟草暴露物的敏感性,参与吸烟引起肺癌的生物学过程,在吸烟人群肺癌发生中扮演着重要的作用。再次体现了在肺癌的发生中,除了主要风险因素吸烟以外,遗传因素也起到重要作用。
rs11597888(G/A) 位点位于miRNA基因AL391839.1上,该基因位于染色体10p11区域,包括83个碱基。通过检索UCSC公共数据库 (http://genome.ucsc.edu/),发现多梳增强子 (EPC1) 基因的外显子的序列包含了miRNA基因AL391839.1的序列。进一步查询dbSNP公共数据库 (https://www.ncbi.nlm.nih.gov/snp/),发现rs11597888也位于EPC1基因的外显子区域,引起第4号密码子亮氨酸的同义改变。EPC1基因属于组蛋白修饰相关基因,是NuA4组蛋白乙酰转移酶复合物的组成成分,与组蛋白乙酰转移酶的活动有关,可以对转录活动起到激活和抑制的作用。鉴于该rs11597888(G/A) 位点没有引起所编码氨基酸的改变,推测它可能通过改变miRNA基因的功能影响肺癌的发生。
rs16867808(T/C) 位点位于miRNA基因AL021918.2上,该基因位于染色体6p22.1区域,该染色体区域已被GWAS研究识别为肺癌遗传易感区域[26]。rs16867808(T/C) 位点也位于ZNF184基因的5′端调控区,ZNF184基因可以影响核酸序列的结合,参与基因表达调控通路。最近一篇文献通过整合之前的GWAS数据资料,在ZNF184基因上也识别了一个新的与肺癌的易感性显著相关的SNP位点 (ZNF184 rs6904596)[27]。LDlink在线数据库分析未发现该位点与本研究识别的阳性关联位点rs16867808(T/C) 之间存在连锁不平衡关系。生物信息学功能数据库注释发现,该位点可以影响染色质重塑,位于基因调控元件 (启动子和增强子) 区。而且该位点也位于A549肺腺癌细胞DNA酶Ⅰ高敏感区域,具有潜在调控基因表达的功能。
由于本研究中识别的3个NSCLC易感性位点是新的位点,且所在的miRNA基因是由之前的芯片预测的新的miRNA基因,因而还不清楚这些位点是否影响所在miRNA基因的功能以及所观察到的关联潜在的生物学机制。研究报道位于miRNA基因上的疾病易感SNPs位点影响miRNA的功能可能基于以下三种机制:改变初始miRNA (pri-miRNA) 的转录,影响pri-miRNA转化为前体miRNA (pre-miRNA) 的过程;以及影响miRNA与靶mRNA之间的交互作用[28]。
本研究通过生物信息学数据探索了可能与NSCLC易感性相关的位于miRNA基因上的潜在功能性SNP位点;筛选miRNAs基因序列上的SNPs位点的范围较广,不同于以往的研究大多局限于一些反复验证的肺癌相关的miRNAs基因去探索肺癌易感性相关的SNPs位点。然而本研究也存在一些局限性:第一,本研究不是基于miRNA基因的已知功能信息,而是基于生物信息学分析识别易感性的miRNA基因单核苷酸多态性位点,所以对识别的肺癌风险相关的miRNA基因的生物学功能知道的并不多,尽管我们综合多个生物信息学数据库也不能很好的解释所观察到的关联效应的潜在生物学机制。因此,所观察到的关联效应还需要在功能机制研究中验证。第二,应用以医院为基础的病例-对照研究设计,可能存在选择偏倚,但是我们严格遵循对象的纳入与排除标准,对病例组和对照组的年龄、性别和地区进行了匹配,并在统计分析中校正混杂因素的影响,尽可能降低偏倚。第三,本研究人群比较单一,还需要在多个人群中进行验证。
综上所述,本研究采用生物信息学分析和病例-对照关联分析的方法,探索了miRNA基因序列上的单核苷酸多态性与NSCLC易感性之间的关联,发现了rs62085660(C/G) 位点与肺鳞癌的发生风险相关,rs11597888(G/A) 和rs16867808(T/C) 位点与吸烟相关性NSCLC的易感性相关。本研究为识别新的肺癌遗传易感性位点以及深入研究miRNA基因在肺癌发生中的作用提供了重要线索,有待后续大样本、多人群以及功能研究的验证和解释。
| [1] | Ferlay J, Soerjomataram I, Dikshit R, et al. Cancer incidence and mortality worldwide: sources, methods and major patterns in GLOBOCAN 2012[J]. Int J Cancer, 2015, 136(5): E359–386. DOI:10.1002/ijc.29210 |
| [2] |
陈万青, 张思维, 邹小农. 中国肺癌发病死亡的估计和流行趋势研究[J].
中国肺癌杂志, 2010, 13(5): 488–493.
Chen W Q, Zhang S W, Zou X N. Estimation and projection of lung cancer incidence and mortality in China[J]. Chin J Lung Cancer, 2010, 13(5): 488–493. DOI:10.3779/j.issn.1009-3419.2010.05.20 |
| [3] | Rom W N, Hay J G, Lee T C, et al. Molecular and genetic aspects of lung cancer[J]. Am J Respir Crit Care Med, 2000, 161(4 Pt 1): 1355–1367. DOI:10.1164/ajrccm.161.4.9908012 |
| [4] | Wu C, Hu Z, Yu D, et al. Genetic variants on chromosome 15q25 associated with lung cancer risk in Chinese populations[J]. Cancer Res, 2009, 69(12): 5065–5072. DOI:10.1158/0008-5472.can-09-0081 |
| [5] | Brownson R C, Alavanja M C, Caporaso N, et al. Family history of cancer and risk of lung cancer in lifetime non-smokers and long-term ex-smokers[J]. Int J Epidemiol, 1997, 26(2): 256–263. DOI:10.1093/ije/26.2.256 |
| [6] | Zienolddiny S, Skaug V. Single nucleotide polymorphisms as susceptibility, prognostic, and therapeutic markers of nonsmall cell lung cancer[J]. Lung Cancer: Targets Ther, 2012, 3: 1–14. DOI:10.2147/LCTT.S13256 |
| [7] | Timofeeva M N, Hung R J, Rafnar T, et al. Influence of common genetic variation on lung cancer risk: meta-analysis of 14 900 cases and 29 485 controls[J]. Hum Mol Genet, 2012, 21(22): 4980–4995. DOI:10.1093/hmg/dds334 |
| [8] | Seng K C, Seng C K. The success of the genome-wide association approach: a brief story of a long struggle[J]. Eur J Hum Genet, 2008, 16(5): 554–564. DOI:10.1038/ejhg.2008.12 |
| [9] | Zhang Q, Jin H, Wang L, et al. Lung cancer risk and genetic variants in East Asians: a meta-analysis[J]. Tumour Biol, 2014, 35(6): 5173–5179. DOI:10.1007/s13277-014-1671-0 |
| [10] | Pipan V, Zorc M, Kunej T. MicroRNA Polymorphisms in Cancer: A Literature Analysis[J]. Cancers (Basel), 2015, 7(3): 1806–1814. DOI:10.3390/cancers7030863 |
| [11] | Hrdlickova B, de Almeida RC, Borek Z, et al. Genetic variation in the non-coding genome: Involvement of micro-RNAs and long non-coding RNAs in disease[J]. Biochim Biophys Acta, 2014, 1842(10): 1910–1922. DOI:10.1016/j.bbadis.2014.03.011 |
| [12] | Chen Z, Xu L, Ye X, et al. Polymorphisms of microRNA sequences or binding sites and lung cancer: a meta-analysis and systematic review[J]. PLoS One, 2013, 8(4): e61008. DOI:10.1371/journal.pone.0061008 |
| [13] | Welter D, MacArthur J, Morales J, et al. The NHGRI GWAS Catalog, a curated resource of SNP-trait associations[J]. Nucleic Acids Res, 2014, 42(Database issue): D1001–1006. DOI:10.1093/nar/gkt1229 |
| [14] | Consortium E P. The ENCODE (ENCyclopedia Of DNA Elements) Project[J]. Science, 2004, 306(5696): 636–640. DOI:10.1126/science.1105136 |
| [15] | Siepel A, Bejerano G, Pedersen J S, et al. Evolutionarily conserved elements in vertebrate, insect, worm, and yeast genomes[J]. Genome Res, 2005, 15(8): 1034–1050. DOI:10.1101/gr.3715005 |
| [16] | Lewis B P, Burge C B, Bartel D P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120(1): 15–20. DOI:10.1016/j.cell.2004.12.035 |
| [17] | Matys V, Kel-Margoulis O V, Fricke E, et al. TRANSFAC and its module TRANSCompel: transcriptional gene regulation in eukaryotes[J]. Nucleic Acids Res, 2006, 34(Database issue): D108–110. DOI:10.1093/nar/gkj143 |
| [18] | Di Leva G, Garofalo M, Croce C M. MicroRNAs in cancer[J]. Annu Rev Pathol, 2014, 9: 287–314. DOI:10.1146/annurev-pathol-012513-104715 |
| [19] | Del Vescovo V, Grasso M, Barbareschi M, et al. MicroRNAs as lung cancer biomarkers[J]. World J Clin Oncol, 2014, 5(4): 604–620. DOI:10.5306/wjco.v5.i4.604 |
| [20] | Xie K, Wang C, Qin N, et al. Genetic variants in regulatory regions of microRNAs are associated with lung cancer risk[J]. Oncotarget, 2016. DOI:10.18632/oncotarget.10299 |
| [21] | Shin K M, Jung D K, Hong M J, et al. The pri-let-7a-2 rs1143770C>T is associated with prognosis of surgically resected non-small cell lung cancer[J]. Gene, 2016, 577(2): 148–152. DOI:10.1016/j.gene.2015.11.036 |
| [22] | Tian T, Shu Y, Chen J, et al. A functional genetic variant in microRNA-196a2 is associated with increased susceptibility of lung cancer in Chinese[J]. Cancer Epidemiol Biomarkers Prev, 2009, 18(4): 1183–1187. DOI:10.1158/1055-9965.EPI-08-0814 |
| [23] | Hu Z, Chen J, Tian T, et al. Genetic variants of miRNA sequences and non-small cell lung cancer survival[J]. J Clin Invest, 2008, 118(7): 2600–2608. DOI:10.1172/JCI34934 |
| [24] | Qiu F, Yang L, Ling X, et al. Sequence variation in mature microRNA-499 confers unfavorable prognosis of lung cancer patients treated with platinum-based chemotherapy[J]. Clin Cancer Res, 2015, 21(7): 1602–1613. DOI:10.1158/1078-0432.ccr-14-1174 |
| [25] | Pei J, Balsara B R, Li W, et al. Genomic imbalances in human lung adenocarcinomas and squamous cell carcinomas[J]. Genes Chromosomes Cancer, 2001, 31(3): 282–287. DOI:10.1002/gcc.1145 |
| [26] | Walsh K M, Gorlov I P, Hansen H M, et al. Fine-mapping of the 5p15.33, 6p22.1-p21.31, and 15q25.1 regions identifies functional and histology-specific lung cancer susceptibility loci in African-Americans[J]. Cancer Epidemiol Biomarkers Prev, 2013, 22(2): 251–260. DOI:10.1158/1055-9965.epi-12-1007-t |
| [27] | Zuber V, Marconett C N, Shi J, et al. Pleiotropic Analysis of Lung Cancer and Blood Triglycerides[J]. J Natl Cancer Inst, 2016, 108(12): djw167. DOI:10.1093/jnci/djw167 |
| [28] | Sun G, Yan J, Noltner K, et al. SNPs in human miRNA genes affect biogenesis and function[J]. RNA, 2009, 15(9): 1640–1651. DOI:10.1261/rna.1560209 |



