急性肺损伤(acute lung injury,ALI)以及其严重阶段的急性呼吸窘迫综合征(acute respiratory distress syndrome,ARDS)是由以肺泡毛细血管膜弥漫性损伤、严重低氧血症为特征的呼吸危重症,其病因多样,病死率高,目前仍是临床上的棘手问题[1]。其中,由革兰阴性杆菌感染引起的脓毒血症是导致ALI/ARDS的重要原因,而脂多糖(lipopolysaccharides,LPS)作为革兰阴性杆菌的胞壁成分是导致脓毒症的主要因素[2]。
活化转录因子3(acting transcription factor 3,ATF3)属于ATF/CREB家族成员。ATF3作为调节中性粒细胞招募聚集的转录因子,是应激早期快反应基因。既往研究表明ATF3广泛参与各器官的炎症反应,与机体稳态维持、肿瘤发生[3]等多方面密切相关。目前大部分研究均表明,ATF3在多种炎症反应过程中,扮演了负性调控下游炎症因子的角色,对炎症反应起刹车作用[4-7]。根据文献[8]及我们的前期研究结果显示,ATF3在LPS导致的急性肺损伤肺组织中是呈高表达的,且对炎症损伤具有保护作用。但ATF3在急性肺损伤中是如何发挥其炎症保护作用的,其具体的分子调控机制尚不清楚。
基因芯片(gene chip)又称为DNA芯片或生物芯片,其测定原理是杂交测序法,是指将大量探针分子与样品分子进行杂交,通过检测杂交信号的强弱从而获取样品分子的数量和序列信息,可以一次性对样品进行高通量筛选与检测分析,主要用于基因测序工作[9]。本研究采用基因芯片技术,检测被LPS处理后的野生型小鼠(C57小鼠)和ATF3敲基因小鼠肺组织之间基因表达谱的差异,希望寻找与ATF3炎症调控相关的差异基因,从而为探索ATF3在LPS导致的急性肺损伤中起炎症保护作用的潜在分子机制奠定理论基础。
1 材料与方法 1.1 材料野生型C57小鼠购自第三军医大学新桥医院实验动物中心,实验选取1只6~8周龄雄性小鼠,体质量18~22 g;ATF3敲基因小鼠由美国俄亥俄州立大学的TsonwinHai教授馈赠,放置于新桥医院实验动物中心进行传代繁育,选取1只6~8周龄雄性且体质量18~22 g的小鼠用于本实验。在基因芯片实验中,分别选取C57小鼠和ATF3敲基因小鼠各1只,在PCR实验中,先后选取C57小鼠和ATF3敲基因小鼠各6只。LPS(2880)购自美国Sigma公司,TRIzol试剂购自美国Invitrogen公司,PCR反转录试剂盒以及Taq聚合酶均 购自日本TaKaRa公司,GeneChip ® Mouse Transcr-iptome Assay 1.0购自上海其明生物有限公司。
1.2 方法 1.2.1 小鼠模型的建立按照15 mg/kg的比例计算小鼠所需的LPS液的量,并用1 mL 无菌注射器抽取相应量的LPS液。然后用左手拇指与食指抓住小鼠的颈部皮肤,以固定小鼠头部,并用左手小指固定小鼠尾巴,将小鼠腹部朝上,右手持注射器,使注射器与皮肤呈45°角刺入小鼠左下腹,针尖通过腹肌后抵抗消失,回抽确认无血或无尿液后,再将LPS液注入腹腔内,因小鼠皮肤较薄,故注射时需观察是否有液体外漏。LPS腹腔注射2 h后,处死小鼠,取出肺组织置于液氮中冻存过夜。
1.2.2 肺组织RNA提取将肺组织放入灭菌灭酶的EP管中,加入TRIzol液1 mL,用匀浆器研磨肺组织;每管TRIzol组织混悬液中加入氯仿200 L,颠倒混匀,冰上静置20 min;低温离心20 min,12 000 r/min,4 ℃;取上清液放入新的 EP管中,加入异丙醇300 L,颠倒混匀,冰上静置15 min;低温离心15 min,12 000 r/min,4 ℃;弃上清,加入75%的乙醇1 mL,颠倒混匀,冰上静置10 min;低温离心15 min,12 000 r/min,4 ℃;弃上清,倒置在滤纸上,晾干10 min;加入灭菌灭酶水 50 mL,充分混匀,可置于-80 ℃冻存或进行下一步实验。
1.2.3 基因芯片检测采用Affymetrix WT芯片推荐试剂盒标准操作流程对总RNA进行放大、标记和纯化,获得带有生物素标记的cDNA。按照Affymetrix表达谱芯片配套的杂交标准流程在滚动杂交炉中45 ℃、16 h滚动杂交,杂交完成后再在洗涤工作站上按照Affymetrix提供的标准操作流程进行芯片的洗涤。芯片结果采用GeneChip-Scanner 3000进行扫描,用Command Console Software 4.0读取原始数据,指控合格的数据采用Expression Console进行归一化处理。每组样本需有3个以上生物学重复,差异筛选主要利用SAM方法,在预先设定的分组下筛选具有显著差异的基因。首先对每个基因通过构造1个统计量来衡量其在相应的分组情况下基因表达量与设定分组间的相关程度,然后通过一系列(1 000次)置换的方法来模拟上述统计量在随机状态(随机置换样本)下的分布。考虑低丰度基因方差的影响以及基因间的相关性,通过适当的方法调整统计量的计算与显著性评判方法来得到基因的显著性水平。最后,为了调整多重检验的P值,我们通过构造一系列离散化拒绝域的方法计算Q值用来控制误判率。
1.2.4 荧光定量PCR(RT-PCR)验证采用RT-PCR对重点差异基因在野生型C57小鼠和ATF3敲基因小鼠肺组织中的表达水平进行进一步验证,观察其检测结果与基因芯片结果是否一致。检测基因引物序列由金斯瑞公司设计合成。配制20 L的反应体系,其中上游引物0.5 L,下游引物0.5 L,SYRB Enzyme 10 L,cDNA 1 L,无酶水8 L。反应条件,预变性:95 ℃,30 s;退火-延伸:95 ℃,5 s,60 ℃,30 s,40个循环;熔解曲线:70~95 ℃,每一步上升1 ℃,第1步等待45 s,以后每一步等待5 s。
1.3 统计学分析所有数据采用SPSS 13.0统计软件进行分析,用单因素方差分析来进行多组间比较,组间的两两比较采取SNK检验,计量资料以x±s来表示,P<0.05认为差异具有统计学意义。
2 结果 2.1 提取总RNA结果提取野生型C57小鼠和ATF3敲基因小鼠肺组织的总RNA,经凝胶电泳检测,结果显示,肺组织总RNA电泳条带清晰,两组肺组织总RNA的光密度值D(260)/D(280)均为2.02,C57小鼠肺组织总RNA浓度为280.2 ng/μL,总量为7.01 μg; ATF3敲基因小鼠肺组织总RNA浓度为332 ng/μL,总量为10.80 μg,样品纯度、总量及完整性均符合表达谱芯片实验要求。
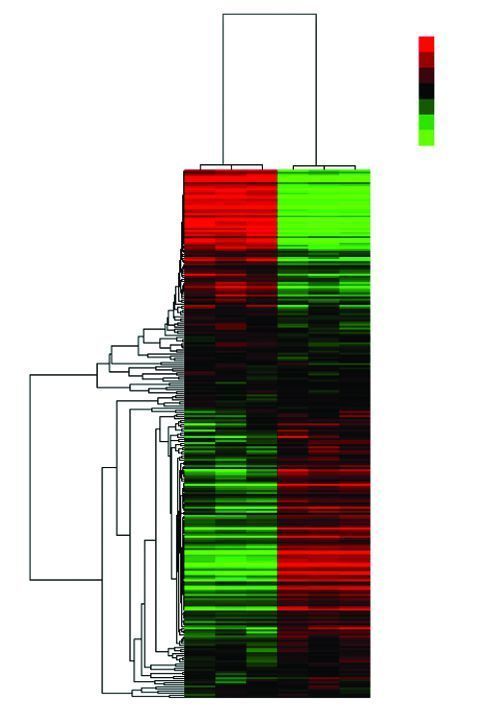
2.2 芯片结果聚类图根据每个基因在样本中的信号值来分别对基因和样本进行聚类分析,可以从样本的聚类结果中发现样本组内和组间的相关性情况,从基因的聚类结果中可以发现聚为一类的基因群在样本中具有类似的表达情况,可能具有相似的功能。与野生型C57小鼠比较,ATF3敲基因小鼠共筛选出1 692个差异基因,其中有1 038个基因是表达下调的,656个基因是表达上调的(图 1)。
|
| 上调基因用红色表示,下调基因用绿色表示 图 1 差异基因表达的聚类图 |
2.3 芯片检测结果分析
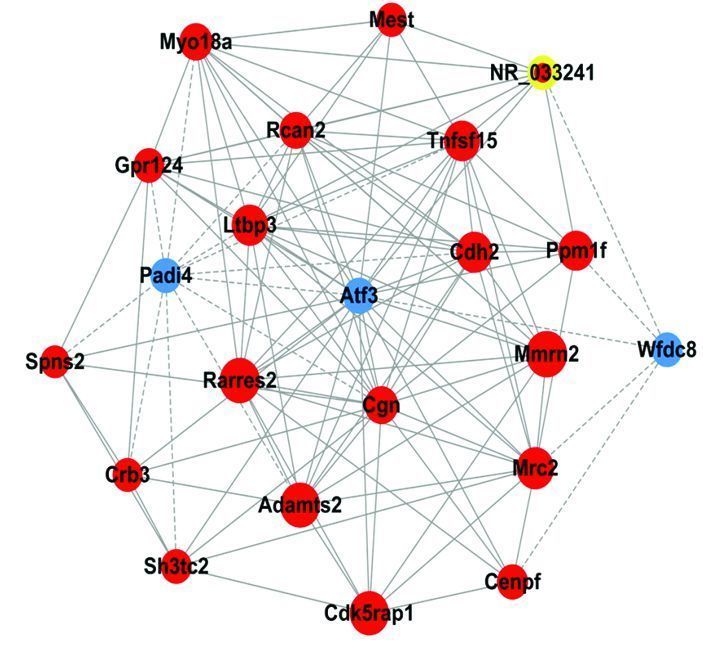
与野生型C57小鼠相比,ATF3敲基因小鼠肺组织中有21个基因表达差异显著(图 2),其中1个为非编码序列;在可编码基因中,上调基因18个,下调基因2个。进一步对基因GO的信息进行分析,结果显示这些差异基因主要与应激反应、炎症因子分泌调控、血 管生成、蛋白及RNA修饰、转录转运等密切相关(表 1)。
|
| 红色圆点表示该基因表达是上调的,蓝色圆点表示该基因是表达下调的,黄色圆点表示该基因为非编码序列 图 2 与ATF3敲基因小鼠有关的差异基因表达 |
| 基因名称 | 差异倍数 | GO名称 |
| Rcan2 | 1.84 | 应激反应 |
| Mest | 1.60 | 维甲酸应答 |
| Mmrn2 | 1.43 | 血管形成 |
| Tnfsf15 | 1.37 | 促进细胞因子的分泌 |
| Cenpf | 1.34 | 蛋白转运 |
| Cdk5rap1 | 1.33 | tRNA修饰 |
| Spns2 | 1.30 | 跨膜转运 |
| Cgn | 1.30 | 紧密连接的装配 |
| Sh3tc2 | 1.28 | 表皮生长因子受体信号通路的调节 |
| Gpr124 | 1.27 | 血脑屏障的建立 |
| Mrc2 | 1.26 | 胞吞作用 |
| Adamts2 | 1.25 | 肺发育 |
| Ltbp3 | 1.24 | 激活转录生长因子 |
| Cdh2 | 1.24 | 细胞紧密连接 |
| Myo18a | 1.22 | 细胞迁移 |
| Rarres2 | 1.22 | 炎症应答 |
| Ppm1f | 1.21 | 凋亡过程 |
| Crb3 | 1.21 | 细胞-细胞连接组织 |
| Wfdc8 | -1.46 | 生物过程 |
| Padi4 | -1.22 | 免疫系统过程 |
2.4 RT-PCR验证结果
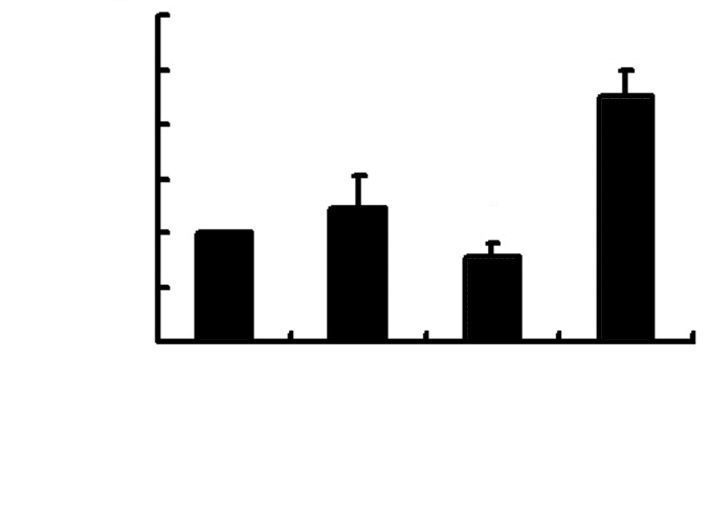
根据芯片分析结果,通过查阅文献的方式我们将上述 21个差异显著的基因进行了进一步筛查。最终将目标基因锁定在Tnfsf15,其又名肿瘤坏死因子样配体1A(TNF-like cytokine 1A,TL1A)。我们对该基因进行了RT-PCR技术验证,结果显示,在ATF3敲基因小鼠肺组织中,该基因是表达显著上调的,变化趋势与基因芯片结果一致(图 3),说明基因芯片结果可靠。
|
| 1:野生型C57小鼠;2:AFT3敲基因小鼠;3:LPS处理后的野生型C57小鼠;4:LPS处理后的AFT3敲基因小鼠 a:P<0.05,与野生型C57小鼠比较;b:P<0.01,与LPS处理后的野生型C57小鼠比较 图 3 RT-PCR验证TL1A在野生型C57小鼠和AFT3敲基因小鼠肺组织中的表达 |
3 讨论
ALI和ARDS作为呼吸系统的危重症,目前仍有接近40%的病死率[10]。在ALI/ARDS的病理生理发生过程中,有许多因素参与其中,包括炎症细胞的激活、炎症介质的释放、免疫功能的失调,甚至包括microRNA的炎症调控等。总的来说,炎症反应的失控是导致疾病发生、发展的重要因素[11-12]。目前,临床上针对ALI/ARDS的治疗主要以对症支持治疗为主[1],且病死率仍然居高不下,针对其病理机制仍缺乏有效的救治策略。因此,对ALI/ARDS的病理分子机制研究就显得尤为重要。
ATF3作为一个快速应激反应基因,在众多炎症反应过程中起调控作用。在发热反应中,热休克转录因子1(heatshocktranscriptionfactor 1,HSF1)诱导ATF3的表达,从而抑制了IL-6的生成[13]。ATF3能减轻由卵清蛋白引起的肺部过敏性炎症,降低其气道高反应性[14]。在呼吸机导致的肺损伤模型中,有研究证明了ATF3敲基因小鼠的肺部损伤更重,细胞渗出及炎症因子的表达均高于野生对照组,说明ATF3对呼吸机相关性肺损伤有保护作用,能抗衡环切力和高通气带来的炎症损害[15]。进一步的研究表明,这一保护作用可能是通过抑制了Nrf2的降解达成的[16]。本课题组的前期研究结果也证实了ATF3在LPS导致的ALI小鼠 模型中呈早期高表达,且对炎症反应具有保护效应,但其机制并不明确。为进一步探究其潜在的分子机制,我们采取了基因芯片的技术方法,通过基因芯片来筛查被LPS处理后的野生型小鼠和ATF3敲基因小鼠肺组织中的差异基因表达,为后期分子机制研究奠定了基础。
本研究通过基因芯片技术,在LPS导致的ALI小鼠模型中,成功筛选出了在LPS导致的ALI小鼠模型中与ATF3相关的差异基因表达。经过筛选,与野生型小鼠比较,ATF3敲基因小鼠共筛选出1 692个差异基因,有21个与炎症调控相关的基因表达差异明显,其中,Myo18a、Mest、Gpr124、Rcan2、Tnfsf15、Ltbp3、Cdh2、Ppm1f、Spns2、Rarres2、Cgn、Mmrn2、Crb3、Adamts2、Mrc2、Sh3tc2、Cdk5rap1、Cenpf共18个编码基因在ATF3敲基因小鼠肺组织中表达明显上调;Padi4、Wfdc8共2个编码基因表达明显下调。通过GO分析,可以看到在这些差异基因中,大部分与炎症调控是相关的,比如参与应激反应、上调细胞因子分泌、炎症应答、蛋白转运、RNA修饰、细胞粘附等生物学过程。
根据基因芯片筛选结果,针对上述21个差异显著的基因,我们通过查阅文献的方式进行进一步筛查,寻 找到重点基因,并用RT-PCR进行验证。根据文献[17-21] 报道,Tnfsf15又名TL1A,是肿瘤坏死因子家族成员之一,表达于单核细胞、巨噬细胞、树突状细胞、滑膜成纤维细胞以及内皮细胞等多种细胞中,对各种微生物、免疫复合物、细胞因子等刺激产生应答。研究发现,TL1A与其功能性受体DR3结合后能诱导NF-κB的激活,从而促进组织器官的炎症反应[17, 22-23]。在多种炎症疾病模型中证实了其是一个具有促炎效应的分子[18, 24-25]。虽然TL1A在炎症过程中的调控作用是明确的,但ATF3与TL1A在调控LPS所致的急性肺损伤中的作用关系尚不清楚。
本研究通过基因芯片和RT-PCR阐明了ATF3对LPS导致的急性肺损伤炎症保护机制可能是通过下调TL1A的表达实现的,但缺乏从分子蛋白水平的进一步验证,而且ATF3与TL1A之间是直接作用还是有其他分子通路参与,也不明确。这些都是我们后续需要进一步研究的内容。总之,本研究结果为下一步探究ATF3在急性肺损伤中的潜在调控分子机制打下了坚实的基础,以进一步揭示ARDS的潜在防治靶点。
| [1] | Cutts S, Talboys R, Paspula C, et al. Adult respiratory distress syndrome[J]. Ann R Coll Surg Engl,2016 : 1 –5. DOI:10.1308/rcsann.2016.0238 |
| [2] | Chen H, Bai C, Wang X. The value of the lipopolysaccharide-induced acute lung injury model in respiratory medicine[J]. Expert Rev Respir Med,2010, 4 (6) : 773 –783. DOI:10.1586/ers.10.71 |
| [3] | Bar J, Hasim M S, Baghai T, et al. Induction of Activating Transcription Factor 3 Is Associated with Cisplatin Responsiveness in Non-Small Cell Lung Carcinoma Cells[J]. Neoplasia,2016, 18 (9) : 525 –535. DOI:10.1016/j.neo.2016.07.004 |
| [4] | Gilchrist M, Thorsson V, Li B, et al. Systems biology approaches identify ATF3 as a negative regulator of Toll-like receptor4[J]. Nature,2006, 441 (7090) : 173 –178. DOI:10.1038/natrure04768 |
| [5] | Khuu C H, Barrozo R M, Hai T, et al. Activating transcription factor 3 (ATF3) represses the expression of CCL4 in murine macrophages[J]. Mol Immunol,2007, 44 (7) : 1598 –1605. DOI:10.1016/j.molimm.2006.08.006 |
| [6] | De Nardo D, Labzin L I, Kono H, et al. High-density lipoprotein mediates anti-inflammatory reprogramming of macrophages via the transcriptional regulator ATF3[J]. Nat Immunol,2014, 15 (2) : 152 –160. DOI:10.1038/ni.2784 |
| [7] | Hellmann J, Tang Y, Zhang M J, et al. Atf3 negatively regulates Ptgs2/Cox2 expression during acute inflammation[J]. Prostaglandins Other Lipid Mediat,2015, 116-117 : 49 –56. DOI:10.1016/j.prostaglandins.2015.01.001 |
| [8] | Lai P F, Cheng C F, Lin H, et al. ATF3 Protects against LPS-Induced Inflammation in Mice via Inhibiting HMGB1 Expression[J]. Evid Based Complement Alternat Med,2013, 2013 (7) : 716481 . DOI:10.1155/2013/716481 |
| [9] | Marshall A, Hodgson J. DNA chips: an array of possibilities[J]. Nat Biotechnol,1998, 16 (1) : 27 –31. DOI:10.1038/nbt0198-27 |
| [10] | Diaz J V, Brower R, Calfee C S, et al. Therapeutic strategies for severe acute lung injury[J]. Crit Care Med,2010, 38 (8) : 1644 –1650. DOI:10.1097/CCM.0b013e3181e795ee |
| [11] | Dolinay T, Kim Y S, Howrylak J, et al. Inflammasome-regulated cytokines are critical mediators of acute lung injury[J]. Am J Respir Crit Care Med,2012, 185 (11) : 1225 –1234. DOI:10.1164/rccm.201201-0003OC |
| [12] | Rajasekaran S, Pattarayan D, Rajaguru P, et al. MicroRNA Regulation of Acute Lung Injury and Acute Respiratory Distress Syndrome[J]. J Cell Physiol,2016, 231 (10) : 2097 –2106. DOI:10.1002/jcp.25316 |
| [13] | Takii R, Inouye S, Fujimoto M, et al. Heat shock transcription factor 1 inhibits expression of IL-6 through activating transcription factor3[J]. J Immunol,2010, 184 (2) : 1041 –1048. DOI:10.4049/jimmunol.0902579 |
| [14] | Gilchrist M, Henderson W R, Clark A E, et al. Activating transcription factor 3 is a negative regulator of allergic pulmonary inflammation[J]. J Exp Med,2008, 205 (10) : 2349 –2357. DOI:10.1084/jem.20072254 |
| [15] | Akram A, Han B, Masoom H, et al. Activating transcription factor 3 confers protection against ventilator-induced lung injury[J]. Am J Respir Crit Care Med,2010, 182 (4) : 489 –500. DOI:10.1164/rccm.200906-0925OC |
| [16] | Shan Y, Akram A, Amatullah H, et al. ATF3 protects pulmonary resident cells from acute and ventilator-induced lung injury by preventing Nrf2 degradation[J]. Antioxid Redox Signal,2015, 22 (8) : 651 –668. DOI:10.1089/ars.2014.5987 |
| [17] | Migone T S, Zhang J, Luo X, et al. TL1A is a TNF-like ligand for DR3 and TR6/DcR3 and functions as a T cell costimulator[J]. Immunity,2002, 16 (3) : 479 –492. DOI:10.1016/S1074-7613(02)00283-2 |
| [18] | Cassatella M A, Pereira-da-Silva G, Tinazzi I, et al. Soluble TNF-like cytokine (TL1A) production by immune complexes stimulated monocytes in rheumatoid arthritis[J]. J Immunol,2007, 178 (11) : 7325 –7333. DOI:10.4049/jimmunol.178.11.7325 |
| [19] | Prehn J L, Thomas L S, Landers C J, et al. The T cell costimulator TL1A is induced by FcgammaR signaling in human monocytes and dendritic cells[J]. J Immunol,2007, 178 (7) : 4033 –4038. DOI:10.4049/jimmunol.178.7.4033 |
| [20] | Shih D Q, Kwan L Y, Chavez V, et al. Microbial induction of inflammatory bowel disease associated gene TL1A (TNFSF15) in antigen presenting cells[J]. Eur J Immunol,2009, 39 (11) : 3239 –3250. DOI:10.1002/eji.200839087 |
| [21] | Zhang J, Wang X, Fahmi H, et al. Role of TL1A in the pathogenesis of rheumatoid arthritis[J]. J Immunol,2009, 183 (8) : 5350 –5357. DOI:10.4049/jimmunol.0802645 |
| [22] | Aiba Y, Nakamura M. The role of TL1A and DR3 in autoimmune and inflammatory diseases[J]. Mediators Inflamm,2013, 2013 : 258164 . DOI:10.1155/2013/258164 |
| [23] | Pobezinskaya Y L, Choksi S, Morgan M J, et al. The adaptor protein TRADD is essential for TNF-like ligand 1A/death receptor3 signaling[J]. J Immunol,2011, 186 (9) : 5212 –5216. DOI:10.4049/jimmunol.1002374 |
| [24] | Bamias G, Siakavellas S I, Stamatelopoulos K S, et al. Circulating levels of TNF-like cytokine 1A (TL1A) and its decoy receptor 3 (DcR3) in rheumatoid arthritis[J]. Clin Immunol,2008, 129 (2) : 249 –255. DOI:10.1016/j.clim.2008.07.014 |
| [25] | Sun X, Zhao J, Liu R, et al. Elevated serum and synovial fluid TNF-like ligand 1A (TL1A) is associated with autoantibody production in patients with rheumatoid arthritis[J]. Scand J Rheumatol,2013, 42 (2) : 97 –101. DOI:10.3109/03009742.2012.727026 |



