乳腺癌是女性最常见的恶性肿瘤之一。据统计,2015年美国乳腺癌的发病率居于女性恶性肿瘤首位,在肿瘤相关疾病的死亡率中占第2位,严重威胁女性的生命健康[1]。我国乳腺癌发病率所占比例为15%,是女性发病率最高的恶性肿瘤,且其发病率及死亡率有明显上升趋势[2]。近年来,虽然以外科手术、放疗、化疗、内分泌治疗、靶向治疗为主的综合治疗在乳腺癌治疗中取得了重要进展,但患者的预后并未得到显著改善,乳腺癌发生、发展的分子机制有待深入探讨。
微小RNAs(microRNAs,miRNAs)是含有19~22个核苷酸、进化上保守的非编码RNA,不仅在调控生物体正常发育中具有重要作用,而且也参与调控多种恶性肿瘤的病理过程[3]。miRNAs在乳腺癌发生、发展中的作用已有研究报道[4]。我们前期的研究从乳腺癌细胞株中分离得到乳腺癌干细胞,采用基因芯片技术获得其miRNAs表达谱,经实时定量PCR验证,该芯片的准确率达到80%[5]。在此基础上,本研究从宏观的角度,利用调控网络分析在基因芯片大数据研究中的独特优势,筛选调控乳腺癌的关键miRNAs,并验证其在乳腺癌侵袭及迁移中的作用。
1 材料与方法 1.1 细胞、主要试剂及仪器人乳腺癌细胞系MCF-7、T47D及正常乳腺上皮细胞MCF-10A购自第三军医大学中心实验室。Gibco胎牛血清、Gibco DMEM-H培养基、RPMI1640培养基购自重庆衍庆生物技术有限公司,青霉素和链霉素购自上海碧云天生物技术公司,胰酶购自HyClone公司,miRNA negative control、miRNA agomir、miRNA antagomir购自广州市锐博生物技术有限公司,逆转录试剂盒及qRT-PCR试剂盒购自宝生物工程有限公司,Transwell小室(8 μm孔径聚碳酸酯膜)和Matrigel购自美国Corning Costar公司,梯度PCR仪及荧光定量PCR仪购自美国Applied公司,倒置相差显微镜购自德国Leica公司。
1.2 相关特异miRNAs的靶基因预测利用miRNAs靶基因预测的在线网络TargetScan(http://www.targetscan.org/)和miRanda(http://www.microrna.org/microrna/home.do)等工具对乳腺癌干细胞miRNAs进行靶基因预测,得到显著差异miRNAs调控的潜在靶基因。
1.3 miRNAs-gene-network和miRNAs-GO-network以人乳腺癌和乳腺癌干细胞样细胞的miRNAs芯片为研究对象[5],对筛选出的靶基因进行显著靶向性功能(gene ontology,GO)分析,得到具有显著性、低误判率、靶向性的GO功能富集以及对应的靶基因,构建差异miRNAs与靶基因的作用网络miRNAs-gene-network以及差异miRNAs与靶基因显著性功能之间的作用网络miRNAs-GO-network。利用图论的方法对miRNAs在两个网络中的调控地位进行评价,评价标准以miRNAs在网络中的维度(Degree)来衡量,用维度表示网络中的miRNAs调控靶基因的数目和功能多少,维度越大代表miRNAs调控的靶基因数目越多、功能越强。
1.4 MCF-7及T47D细胞培养及基因转染采用含10%胎牛血清、1%双抗的DMEM-H或RPMI1640培养基,于37 ℃、5% CO2孵箱中分别培养MCF-7和T47D细胞,倒置相差显微镜观察细胞形态,贴壁达到70%~80%,胰酶消化后,进行传代。实验分3组:Negative control组、miR-106a-5p agomir组、miR-106a-5p antagomir组。取对数生长期的细胞,5 mL PBS清洗3次,1 mL胰酶消化后加入含10% 胎牛血清的完全培养基4 mL重悬、计数,24孔板每孔接种1×105个细胞,37 ℃、5% CO2 孵箱中培养,过夜。去除培养基,PBS清洗3次后每孔加入DMEM或RPMI1640 完全培养基500 μL,再分别加入1 μL Negative control、miR-106a-5p agomir、miR-106a-5p antagomir,轻轻晃动24孔板混匀,37 ℃、5% CO2孵箱中培养24~48 h。
1.5 实时荧光定量PCR 检测miR-106a-5p表达水平检测过表达或敲低miR-106a-5p后MCF-7和T47D细胞中miR-106a-5p的表达水平,将细胞接种在6孔板,分为3组:Negative control组、miR-106a-5p agomir组、miR-106a-5p antagomir组,转染48 h采用TRIzol法提取总RNA,miR-106a-5p特异引物进行反转录,获得相应cDNA,以cDNA为模板进行实时荧光定量PCR检测,以U6 RNA为内参对照校正PCR模板的拷贝数,其相对表达量用2-ΔΔCt值计算。引物序列如下,hsa-miR-106a-5p反转录引物: 5′-TCGTATCCAGTGCAGG-GTCCGAGGTATTCGCACTGGATACGACctacc-3′;hsa-miR-106a-5p上游引物: 5′-AAAAGTGCTTACAGTGC-AGGTAG-3′;下游引物: 5′-GGAAAAGTGCTTACAGTGCAGGT-3′。
1.6 过表达及敲低miR-106a-5p对MCF-7和T47D细胞侵袭能力的影响4 ℃ 复温基质胶和高压消毒后的PBS溶液,当基质胶呈液态时,基质胶和PBS按1:8比例配置成混合液,每个小室上室缓慢均匀加入80 μL混合液,避免有气泡产生,37 ℃孵育过夜;选对数生长的细胞,胰酶消化,计数。下室加入800 μL 含15% FBS的完全培养基,按照分组上室分别加入200 μL用无血清DMEM-H或RPMI1640培养基重悬的细胞,细胞数为2×105个。37 ℃、5% CO2孵箱中培养48 h;1 mL 95% 乙醇溶液固定10 min,PBS清洗3次,1% 结晶紫染色10 min,小水流清洗、棉签轻轻擦除上室结晶紫和基质胶,倒置显微镜细胞观察、计数。
1.7 过表达及敲低miR-106a-5p对MCF-7和T47D细胞迁移能力的影响选对数生长的细胞,胰酶消化,计数。将小室放入24孔板中,下室加入800 μL含15% 胎牛血清的DMEM-H或RPMI1640完全培养基,按照分组上室分别加入200 μL无血清DMEM-H或者RPMI1640培养基重悬的细胞,细胞数为4×104个。37 ℃、5% CO2孵箱中培养24 h;清除培养基后,以1 mL 95% 乙醇溶液固定10 min。PBS清洗3次,1% 结晶紫染溶液色10 min。小水流清洗上室结晶紫,棉签轻轻擦净。倒置显微镜观察,拍照,细胞计数。
1.8 细胞划痕实验先用标记笔在6孔板背面垂直划3~4条直线,按照分组进行细胞种板,采用无双抗10%胎牛血清的DMEM-H或RPMI1640培养基培养MCF-7或T47D细胞株;待细胞融合度达到60%~70%时,加入丝裂霉素干预处理2 h(终浓度10 ng/mL,减少细胞侵袭对迁移的影响),PBS清洗3次,垂直背后标记横线用 100 μL移液枪头轻轻作划痕。PBS清洗除去脱落细胞,倒置显微镜下拍照,并记录拍照位置;24 h后重复在记录位置拍照。0 h和24 h分别在划痕的上中下3个位置,用Image J工具测量划痕间距,得到3次的均值L0和L24,并计算L=L0-L24,得到细胞平均迁移距离。
1.9 统计学分析数据以x±s表示,采用SPSS 17.0统计软件进行独立样本t检验。P<0.05为差异具有统计学意义。
2 结果 2.1 miRNAs-gene-network的建立对乳腺癌和乳腺癌干细胞进行miRNAs靶基因预测、乳腺组织特异性筛选和显著靶向性功能分析,建立miRNAs与调节基因的网络miRNAs-gene-network,见图 1。miRNAs调控的Gene越多,其面积就越大;同理,Gene受到miRNAs的调控越多,其面积也越大。以调控靶分子的数目(Degree)来衡量miRNAs在网络中的调控地位,Degree值较高的miRNAs见表 1。
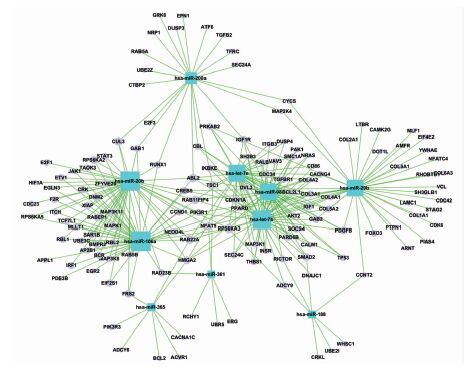
|
| 矩形为下调miRNAs;圆形为gene;直线表示miRNAs与gene的调控关系 图 1 miRNAs-gene-network |
| miRNAs | Degree |
| miR-106a-5p | 48 |
| miR-20b-5p | 48 |
| let-7b-5p | 42 |
| let-7e-5p | 39 |
| miR-29b-3p | 39 |
| miR-98-5p | 39 |
2.2 miRNAs-GO-network的建立
对乳腺癌和乳腺癌干细胞样细胞进行miRNAs和显著靶向性功能分析,建立miRNAs与基因功能的调控网络miRNAs-GO-network,见图 2。miRNAs调控的GO越多,其面积就越大;同理,GO受到miRNAs的调控越多,其面积也越大。以调控靶分子功能的数目(Degree)来衡量miRNAs在网络中的调控地位,Degree值较高的miRNAs见表 2。
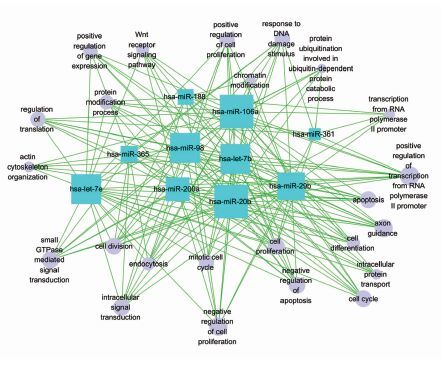
|
| 矩形为miRNAs;圆形为GO;直线表示miRNAs与GO的调控关系 图 2 miRNAs-GO-network |
| miRNAs | Degree |
| miR-106a-5p | 36 |
| miR-20b-5p | 36 |
| let-7b-5p | 33 |
| let-7e-5p | 33 |
| miR-29b-3p | 33 |
| miR-98-5p | 33 |
2.3 关键miRNA的筛选
在两个网络中调控维度最高的miRNAs基本一致,miR-106a-5p是调控维度最高的miRNAs之一,可能是调控乳腺癌恶性表型的关键miRNAs之一。
2.4 实时荧光定量PCR检测miR-106a-5p的表达结果表明miR-106a-5p在乳腺癌细胞中的表达下调8.866倍(t=3.827,P=0.019,图 3A);过表达或敲低miR-106a-5p后实时荧光定量PCR检测miR-106a-5p在乳腺癌MCF-7及T47D细胞中的表达水平,结果显示,在MCF-7和T47D细胞中miR-106a-5p agomir 可明显使miR-106a-5p过表达,分别上调3.480倍和11.811倍(t=-7.872,P=0.001;t=-4.772,P=0.009,图 3B、C);而miR-106a-5p antagomir可明显抑制miR-106a-5p的表达,分别下调2.841倍和1.949倍(t=6.732,P=0.003;t=3.259,P=0.031,图 3B、C)。
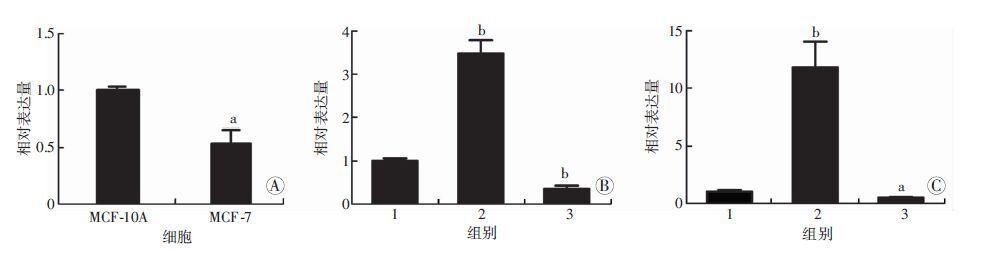
|
| A:人正常乳腺上皮细胞MCF-10A和乳腺癌细胞MCF-7中miR-106a-5p的相对表达量 a:P<0.05,与MCF-10A比较;B、C: 分别为乳腺癌MCF-7、T47D细胞中miR-106a-5p的相对表达量 1:Negative control组; 2: miR-106a-5p agomir组; 3: miR-106a-5p antagomir组;a:P<0.05,b:P<0.01,与Negative control组比较 图 3 实时荧光定量PCR检测miR-106a-5p的相对表达量 |
2.5 转染miR-106a-5p对MCF-7和T47D细胞侵袭能力的影响
处理48 h后,MCF-7细胞的穿膜细胞数:Negative control组(72.67±4.73)、miR-106a-5p agomir组(57.00±3.61)、miR-106a-5p antagomir组(179.67±3.06);T47D细胞的穿膜细胞数:Negative control组(67.33±2.52)、miR-106a-5p agomir组(52.67±5.13)、miR-106a-5p antagomir组(164.33±4.04)。结果显示,过表达miR-106a-5p可抑制MCF-7 (t=4.565,P=0.010)和T47D (t=4.445,P=0.011)细胞的侵袭能力;抑制miR-106a-5p的表达可以促进MCF-7 (t=-32.934,P<0.001)和T47D (t=-35.289,P<0.001)细胞的侵袭能力(图 4)。
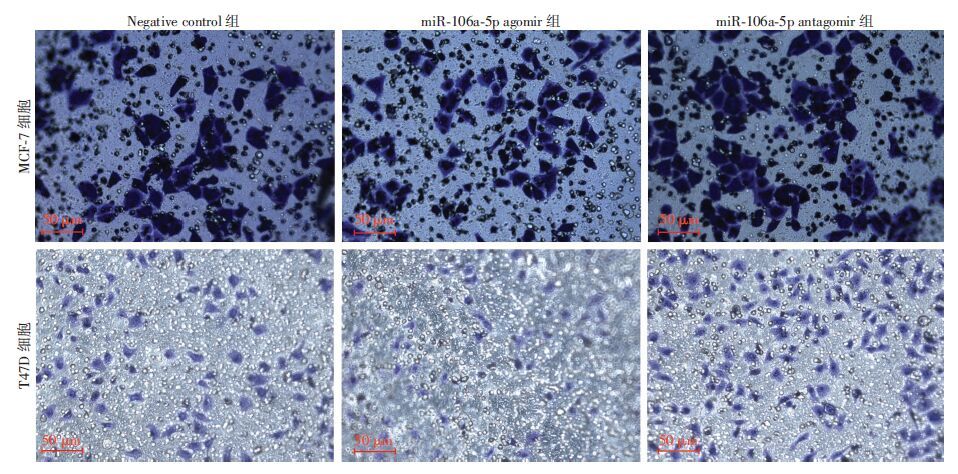
|
| 图 4 结晶紫染色观察miR-106a-5p对MCF-7和T47D细胞侵袭能力的影响 |
2.6 转染miR-106a-5p对MCF-7和T47D细胞迁移能力的影响
处理24 h后,MCF-7细胞的穿膜细胞数:Negative control组(83.33±2.08)、miR-106a-5p agomir组(65.67±2.08)、miR-106a-5p antagomir组(166.67±2.52);T47细胞的穿膜细胞数:Negative control组(20.33±3.51)、miR-106a-5p agomir组(13.33±2.08)、miR-106a-5p antagomir 组(2.67±2.08)。结果显示,过表达miR-106a-5p可抑制MCF-7 (t=10.394,P<0.001)和T47D (t=2.970,P=0.041)细胞的迁移能力,抑制miR-106a-5p的表达可以促进MCF-7 (t=-44.194,P<0.001)和T47D (t=-5.223,P=0.006)细胞的迁移能力(图 5)。
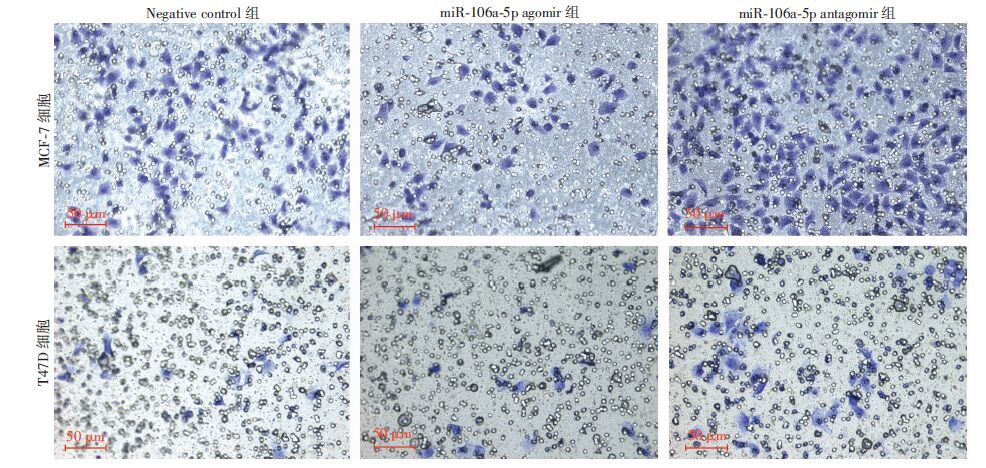
|
| 图 5 结晶紫染色观察miR-106a-5p对MCF-7和T47D细胞迁移能力的影响 |
2.7 转染miR-106a-5p对MCF-7和T47D细胞划痕实验的影响
细胞划痕实验中,MCF-7细胞平均迁移距离:Negative control组(161.33±6.51)μm、miR-106a-5p agomir组(142.67±4.51) μm、miR-106a-5p antagomir组(288.67±8.08) μm;T47D细胞平均迁移距离:Negative control组(106.33±4.73) μm、miR-106a-5p agomir 组(55.67±4.04) μm、miR-106a-5p antagomir组(254.67±5.01) μm。结果显示,过表达miR-106a-5p可抑制MCF-7 (t=4.084,P=0.015)和T47D (t=14.113,P<0.001)细胞的爬行能力,抑制miR-106a-5p的表达则显著增加MCF-7 (t=-21.255,P<0.001)和T47D (t=-35.402,P<0.001)细胞的爬行能力(图 6)。

|
| A:MCF-7细胞;B:T47D细胞 图 6 miR-106a-5p对MCF-7和T47D细胞爬行能力的影响 |
3 讨论
miRNAs是由发夹状miRNAs前体经过RNaseⅢ酶家族中的Drosha酶和Dicer酶剪切加工而来。它通过与特定地靶基因mRNA的3′-UTR非翻译区不完全互补配对,在转录或转录后水平下调靶基因的表达,参与疾病的发生、发展[6]。miRNAs的生物学功能包括区分癌症的表型、药物治疗靶点或者作为药物本身[7]。最新研究表明,在酸性的肿瘤微环境中,通过核酸肽运输抗miRNAs药物(反义寡肽),可实现miRNAs 相关的抗肿瘤治疗[8]。Li等[9]报道血浆miRNAs可用于肝癌的早期诊断。可见,miRNAs在癌症的发生、发展、诊断、维持治疗、预后等方面具有重要的作用。与传统的单个miRNA研究相比,通过miRNAs与靶基因及GO分析建立的网络,系统筛选肿瘤相关关键miRNAs,可以提高miRNAs调控肿瘤恶性表型研究的准确性及可靠性[10]。本研究通过构建乳腺癌相关的miRNAs-gene-network和miRNAs-GO-network调控网络,筛选出miR-106a-5p、miR-20b-5p、let-7b-5p、let-7e-5p、miR-29b-3p、miR-98-5p等6个调控乳腺癌的关键miRNAs,其中miR-106a-5p是乳腺癌调控维度最高的miRNAs之一。它定位于人X染色体(Xq26.2)上,属于miR-106a-363基因簇,该基因簇在肿瘤的发生、发展中具有重要作用[11]。研究发现miR-106a在乳腺癌中具有重要地位[12]。然而,近年来关于miR-106a在乳腺癌中的作用报道不尽相同[13-14]。miR-106a有作为促癌miRNAs的相关报道:通过抑制ZBTB4/Sp/EZH2信号轴促进乳腺癌的发生、发展,而这种促癌作用可被Betulinic acid(白桦脂酸)抑制[15]。相反,Guo等[14]发现miR-106a在雌激素受体阴性的乳腺癌患者中表达下降,具有抑癌的作用,抑制miR-106a的表达可上调毛细血管扩张性共济失调症突变基因(ATM)的表达,而ATM的高表达与ER阴性的患者术后或放疗后复发正相关。因此,miR-106a在乳腺癌中的确切调控作用仍然不清楚,本研究拟探讨其对乳腺癌细胞侵袭及迁移功能的作用。
本研究比较乳腺癌细胞MCF-7与乳腺正常上皮细胞MCF-10A中miR-106a-5p的表达水平,发现miR-106a-5p在MCF-7中表达较低,提示miR-106a-5p可能具有抑癌作用;通过Transwell侵袭实验、Transwell迁移实验以及划痕实验验证,miR-106a-5p确实显著抑制了乳腺癌细胞的侵袭和迁移能力,起着抑癌基因的作用。研究报道在胶质瘤及胃癌中miR-106a可通过分别抑制靶基因E2F1和TIMP2而抑制肿瘤的增殖、侵袭、迁移[16-17],与本研究miR-106a-5p可抑制侵袭、迁移的生物学作用的结论相似。但是,本研究仅对miR-106a-5p的生物学功能进行了探讨,其调控机制尚不清,其调控的靶基因不明,对乳腺癌干细胞的调控作用也未阐明,有待进一步探讨。
此外,本研究还筛查到多个调控维度较高的miRNAs,如miR-20b-5p、let-7b-5p等。既往研究显示,这些miRNAs可能与乳腺癌发生、发展有关,如Xu等[18]发现let-7b与乳腺癌干细胞的干性维持密切相关。因此,我们将逐个探讨这些miRNAs对乳腺癌的调控作用及其确切机制。miRNAs-GO-network初筛表明miR-106a-5p在乳腺癌的多种生物学功能中均有调控作用,而本研究仅选取了侵袭、迁移两个方面进行研究,拟在今后的研究中继续探讨其对乳腺癌的增殖、分化、干性调控等方面的作用。
| [1] | Siegel R L, Miller K D, Jemal A. Cancer statistics, 2016[J]. CA Cancer J Clin,2016, 66 (1) : 7 –30. DOI:10.3322/caac.21332 |
| [2] | Chen W, Zheng R, Baade P D, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin,2016, 66 (2) : 115 –132. DOI:10.3322/caac.21338 |
| [3] | Fang Y X, Gao W Q. Roles of microRNAs during prostatic tumorigenesis and tumor progression[J]. Oncogene,2014, 33 (2) : 135 –147. DOI:10.1038/onc.2013.54 |
| [4] | Aydogdu E, Katchy A, Tsouko E, et al. MicroRNA-regulated gene networks during mammary cell differentiation are associated with breast cancer[J]. Carcinogenesis,2012, 33 (8) : 1502 –1511. DOI:10.1093/carcin/bgs161 |
| [5] | Sun J G, Liao R X, Qiu J, et al. Microarray-based analysis of microRNA expression in breast cancer stem cells[J]. J Exp Clin Cancer Res,2010, 29 : 174 . DOI:10.1186/1756-9966-29-174 |
| [6] | Bartel D P. MicroRNAs: genomics, biogenesis, mechanism, and function[J]. Cell,2004, 116 (2) : 281 –297. DOI:10.1016/S0092-8674(04)00045-5 |
| [7] | Lujambio A, Lowe S W. The microcosmos of cancer[J]. Nature,2012, 482 (7385) : 347 –355. DOI:10.1038/nature10888 |
| [8] | Cheng C J, Bahal R, Babar I A, et al. MicroRNA silencing for cancer therapy targeted to the tumour microenvironment[J]. Nature,2015, 518 (7537) : 107 –110. DOI:10.1038/nature13905 |
| [9] | Li L, Chen J, Chen X, et al. Serum miRNAs as predictive and preventive biomarker for pre-clinical hepatocellular carcinoma[J]. Cancer Lett,2016, 373 (2) : 234 –240. DOI:10.1016/j.canlet.2016.01.028 |
| [10] | Buffa F M, Camps C, Winchester L, et al. microRNA-associated progression pathways and potential therapeutic targets identified by integrated mRNA and microRNA expression profiling in breast cancer[J]. Cancer Res,2011, 71 (17) : 5635 –5645. DOI:10.1158/0008-5472.CAN-11-0489 |
| [11] | Landais S, Landry S, Legault P, et al. Oncogenic potential of the miR-106-363 cluster and its implication in human T-cell leukemia[J]. Cancer Res,2007, 67 (12) : 5699 –5707. DOI:10.1158/0008-5472.CAN-06-4478 |
| [12] | Mishra S, Srivastava A K, Suman S, et al. Circulating miRNAs revealed as surrogate molecular signatures for the early detection of breast cancer[J]. Cancer Lett,2015, 369 (1) : 67 –75. DOI:10.1016/j.canlet.2015.07.045 |
| [13] | Kim K, Chadalapaka G, Lee SO, et al. Identification of oncogenic microRNA-17-92/ZBTB4/specificity protein axis in breast cancer[J]. Oncogene,2012, 31 (8) : 1034 –1044. DOI:10.1038/onc.2011.296 |
| [14] | Guo X, Yang C, Qian X, et al. Estrogen receptor alpha regulates ATM Expression through miRNAs in breast cancer[J]. Clin Cancer Res,2013, 19 (18) : 4994 –5002. DOI:10.1158/1078-0432.CCR-12-3700 |
| [15] | Yang W S, Chadalapaka G, Cho S G, et al. The transcriptional repressor ZBTB4 regulates EZH2 through a MicroRNA-ZBTB4-specificity protein signaling axis[J]. Neoplasia,2014, 16 (12) : 1059 –1069. DOI:10.1016/j.neo.2014.09.011 |
| [16] | Zhu M, Zhang N, He S, et al. MicroRNA-106a targets TIMP2 to regulate invasion and metastasis of gastric cancer[J]. FEBS Lett,2014, 588 (4) : 600 –607. DOI:10.1016/j.febslet.2013.12.028 |
| [17] | Yang G, Zhang R, Chen X, et al. MiR-106a inhibits glioma cell growth by targeting E2F1 independent of p53 status[J]. J Mol Med (Berl),2011, 89 (10) : 1037 –1050. DOI:10.1007/s00109-011-0775-x |
| [18] | Xu L Z, Li S S, Zhou W, et al. p62/SQSTM1 enhances breast cancer stem-like properties by stabilizing MYC mRNA[J]. Oncogene, 2016. [Epub ahead of print]. DOI: 10.1038/onc.2016.202 |



