2. 400038 重庆,第三军医大学基础医学部分子遗传学教研室
成纤维细胞生长因子23(FGF23)是一种在磷酸盐和维生素D代谢中扮演重要角色的骨源性激素样物质。近年研究报道显示血清FGF23浓度升高可增加冠心病(CHD)患病风险,其相关作用机制与FGF23能在血管内皮细胞和平滑肌细胞中促进活性氧簇(ROS)产生引起炎性反应,并诱导血管平滑肌细胞收缩迁移有关[1, 2]。目前发现FGF23基因rs7955866(p.T239M)等部分位点与血清FGF23浓度及生理活性改变相关[3],并因此促进佝偻病、前列腺癌及卵巢癌等临床疾病的发生和发展[4, 5]。而FGF23基因是否亦存在与冠心病发病的相关性,目前尚未见文献报道。冠心病是一种与遗传因素密切相关的多因素疾病,其易感基因的研究发现对于解析CHD发病的分子遗传学机制以及高危人群的筛查和预测、临床早期诊疗等具有重要的科学价值[6]。因此本课题组针对FGF23基因与冠心病的相关性,依托重庆地区汉族人群开展了本次病例对照研究。
1 对象与方法 1.1 伦理声明该项研究符合赫尔辛基宣言并经第三军医大学大坪医院野战外科研究所伦理委员会审核批准,所有参与该试验的人群均知情同意。
1.2 试验对象该试验研究人员筛选2014年5月至2015年3月在我科住院首次接受冠脉造影检查的重庆汉族人群431例,划分为CHD组与对照组进行病例对照研究。CHD组诊断依据采纳1979年WHO颁布的缺血性心脏病诊断标准,并经冠脉造影检查明确诊断(以左主干、左前降支、回旋支及右冠状动脉4支血管中至少1支血管病变狭窄程度≥50%且排除冠脉痉挛、肌桥、创伤、先天畸形为诊断依据),而冠脉造影无异常的非CHD人群划分为对照组[7]。本研究共纳入CHD组231例样本,其中男性140例,女性91例,年龄(63.9±9.6)岁,对照组纳入200例样本,其中男性97例,女性103例,年龄(62.2±10.7)岁。本次入选样本均为相互无血缘关系的重庆地区汉族人群,排除罹患肿瘤、血液病、甲状腺及甲状旁腺疾病、肝炎、肝功能不全(ALT>2 ULN;1ULN=40 U/L)、肾功能不全(Cr≥133 μmol/L)及自身免疫病者。
1.3 试验方法 1.3.1 临床资料采集使用调查问卷的形式采集受试者性别、年龄、吸烟史、饮酒史、用药史、既往史及家族史信息,其中每年吸烟≥ 100支定义为吸烟者[8],每年饮酒≥ 12次定义为饮酒者[9]。实地测量受试者身高、体重并计算体重指数(BMI),采用已校准的水银柱血压计测量受试者坐位上臂血压,以柯氏音第1音及消失音(第5音)确定收缩压和舒张压水平。受试者空腹12 h后采集肘前静脉血,使用BECKMAN DXC800全自动生化分析仪检测甘油三酯(TG)、总胆固醇(TC)、低密度脂蛋白胆固醇(LDL-C)、高密度脂蛋白胆固醇(HDL-C)、糖化血红蛋白(HbA1c)、空腹血糖(FPG)、血肌酐(Scr)、尿酸(UA),罗氏 cobas e601电化学发光法检测受试者血清C肽(CP)、胰岛素浓度(INS),并同法检测受试者餐后2 h血糖浓度(2hPBG)。
1.3.2 DNA提取清晨由肘前静脉采集2 mL的空腹静脉血放置于EDTA抗凝管,-20 ℃冷冻保存。采用天根血液基因组提取试剂盒DP332(北京,中国)对血液样本进行DNA提取,所得DNA样本于-70 ℃条件下保存待用。
1.3.3 基因位点筛选首先从国际人类基因组单体型图计划网站上检索并下载中国汉族人群FGF23基因SNP基因型资料(http://hapmap.ncbi.nlm.nih.gov/),并在国家生物技术信息中心SNP数据库及英国Sanger研究所生物信息库中核对其资料信息(http://www.ncbi.nlm.nih.gov/snp,http://www.ensembl.org/index.html)。然后采用Haploview 4.2软件进行标签SNP(TagSNP)的选取,采取的标准为r2≥0.8和中国汉族人群最小基因频率MAF(CHB)及全球最小基因频率MAF(GLO)均>0.1。按照上述方法共选取FGF23基因的3个标签TagSNP位点,其位点信息见表 1。
| SNPs | MAF(CHB) | MAF(GLO) | 位置 | 功能区 |
| rs7955866 | 0.19 | 0.1474 | Chr12:4349810 | missense |
| rs13312756 | 0.387 | 0.244 | Chr12:4357858 | intron variant |
| rs3812822 | 0.201 | 0.1597 | Chr12:4359496 | upstream variant-487A>G |
本次试验采用Sequenom公司出品的Massarray系统进行基因分型,该系统具有灵敏度高(准确度> 99.7%)、误差小、可同时多位点多样本检测的特点[10],技术服务由 北京六合华大基因科技有限公司提供(深圳,中国)。首先采用AssayDesigner3.1 软件进行引物设计,再通过使用384孔板点样完成PCR扩增反应、SAP反应、延伸反应后去盐,上述反应引物序列见表 2。使用Typer4.0软件输入样本信息及建板,再次点样运行Mass ARRAY系统,完成最后一步CHIP扫描后使用Typer4.0软件进行基因型分析。
| SNPs | PCR引物 | Tm (℃) | 延伸引物 | 方向 |
| rs7955866 | 正向引物:5′- ACGTTGGATGAGCGACCCTAGATGAACTTG-3′ | 55.6 | 5′- TGCGCAGGGCCGGCT-3′ | 反向延伸 |
| 反向引物:5′- ACGTTGGATGTCGAGTGAACACGCACGCTG-3′ | ||||
| rs13312756 | 正向引物:5′- ACGTTGGATGCTTTGAGGTGTTTTCTCACG-3′ | 45 | 5′-TAGATATTGAAAAAGACTGGAT-3′ | 正向延伸 |
| 反向引物:5′- ACGTTGGATGGTAATGTGGTGAAAGCAGGG-3′ | ||||
| rs3812822 | 正向引物:5′- ACGTTGGATGGCTCACAGATTAAGCACTGG-3′ | 46.3 | 5′- TCCTGTCGAGTTTACTTATT-3′ | 正向延伸 |
| 反向引物:5′- ACGTTGGATGAGGCAATTAATCTCCTGTCG-3′ |
首先采用标准χ2检验或者Fisher精确检验的方法对每个SNP位点分型结果进行CHD组及对照组Hardy-Weinberg平衡检验。使用SPSS18.0软件进行独立样本t检验、χ2检验或非参数检验来确定年龄、血压、血脂、血糖及胰岛素等临床信息是否具有组间统计学差异。在线使用SHEsis软件进行基因型、等位基因、单体型的病例对照分析及连锁不平衡(LD)分析(http://analysis.bio-x.cn/myAnalysis.php),采用非条件Logistic回归方法对阳性位点进行单因素及多因素回归分析。上述分析结果具有统计学差异的界点均定义为P < 0.05,SPSS 18.0软件及SHEsis软件分析中均定义95%的可信区间(95%CI),且频率 < 0.03的单体型会在SHEsis软件单体型分析中舍去。
2 结果 2.1 临床基本资料组间分析统计分析结果显示,该病例对照试验中年龄、BMI、饮酒史、收缩压、舒张压及空腹C肽浓度不存在CHD与对照组的组间统计学差异(P>0.05),而性别、高血压病史、吸烟史、心血管病家族史、TC、TG、HDL-C、LDL-C、ApoA1、ApoB、HbA1c、FPG、2 hPBG、INS在CHD组及对照组间存在统计学差异(P < 0.05),具体结果见表 3。
| 组别 | n | 男/女(例) | 年龄(岁, x±s) | BMI(kg/m 2, x±s) | 高血压病史[例(%)] | 吸烟史[例(%)] | 饮酒史[例(%)] |
| CHD组 | 231 | 140/91 a | 63.9±9.6 | 24.4±3.3 | 150(54.9%) a | 81(35.1%) a | 54(23.4%) |
| 对照组 | 200 | 97/103 | 62.2±10.7 | 24.1±3.2 | 108(54%) | 50(25%) | 32(16%) |
| 组别 | 心血管病家族史[例(%)] | SBP(mmHg, x±s) | DBP(mmHg, x±s) | TC(mmol/L, x±s) | TG(mmol/L, x±s) | HDL-C(mmol/L, x±s) | LDL-C(mmol/L, x±s) |
| CHD组 | 57(24.7%) a | 128.8±18.4 | 75.6±10.7 | 4.5±1.1 a | 1.8±1.2 a | 1.1±0.3 a | 2.9±0.9 a |
| 对照组 | 31(15.5%) | 126.2±16.8 | 74.7±10.1 | 4.2±1.0 | 1.5±0.9 | 1.2±0.3 | 2.7±0.8 |
| 组别 | ApoA1(g/L, x±s) | ApoB(g/L, x±s) | HbA1c(%, x±s) | FPG(mmol/L, x±s) | 2 h PBG(mmol/L, x±s) | CP(ng/mol, x±s) | INS(pmol/L, x±s) |
| CHD组 | 1.2±0.2 a | 0.9±0.3 a | 5.9±0.6 a | 5.5±1.1 a | 9.7±2.9 a | 2.8±1.8 | 76.0±72.9 a |
| 对照组 | 1.3±0.2 | 0.8±0.3 | 5.8±0.9 | 5.3±1.3 | 8.4±2.8 | 2.4±0.7 | 54.7±28.7 |
| a: P < 0.05,与对照组比较 | |||||||
本实验3个SNP位点在对照组的基因频率分布经标准χ2检验或Fisher精确检验后均符合哈温平衡(P>0.05),具有群体代表性。
2.3 等位基因及基因型组间频率分布使用SHEsis在线分析发现CHD组rs7955866 A等位基因和rs3812822 C等位基因频率明显高于对照组(P < 0.001,表 4),两位点组间基因型分布也存在明显差异(P < 0.01,表 4),然而rs13312756位点等位基因及基因型分布未发现统计学差异(P>0.05)。单因素Logistic回归分析示rs7955866 GA基因型携带者患病风险为GG基因型的2.146倍,具有统计学意义(P < 0.05),rs3812822 CT基因型与TT基因型携带者患病风险亦具有统计学差异(P < 0.05)。多因素非条件Logistic回归分析调整性别、年龄、BMI、吸烟饮酒史、高血压病史、高胆固醇血症等混杂因素后,结果显示rs7955866及rs3812822位点均与CHD发病独立相关(P < 0.001),见表 5。
| SNPs | 等位基因(1/2) | 组别 | 基因型[例(%)] | P | 等位基因[例(%)] | OR(95% CI) | P | |||
| 1/1 | 1/2 | 2/2 | 1 | 2 | ||||||
| rs7955866 | A/G | CHD组 | 5(0.022) | 84(0.364) | 142(0.615) | <0.001 * | 94(0.203) | 368(0.797) | 2.015(1.372~2.959) | <0.001 * |
| 对照组 | 1(0.005) | 43(0.215) | 156(0.780) | 45(0.113) | 355(0.887) | |||||
| rs13312756 | C/G | CHD组 | 140(0.606) | 82(0.355) | 9(0.039) | 0.229 | 362(0.784) | 100(0.216) | 1.305(0.954~1.786) | 0.096 |
| 对照组 | 107(0.535) | 80(0.400) | 13(0.065) | 294(0.735) | 106(0.265) | |||||
| rs3812822 | C/T | CHD组 | 8(0.035) | 92(0.398) | 131(0.567) | 0.002 | 108(0.234) | 354(0.766) | 1.836(1.289~2.615) | <0.001 * |
| 对照组 | 3(0.015) | 51(0.255) | 146(0.730) | 57(0.142) | 343(0.858) | |||||
| SNPs | 单因素非条件Logistic回归分析 | 多因素非条件Logistic回归分析 | |||||
| 组别 | P < /td> | OR | 95% CI | P < /td> | OR | 95% CI | |
| rs7955866 | AG:GG | 0.01 | 2.146 | 1.393~3.306 | <0.001 | 2.478 | 1.613~3.806 |
| rs3812822 | CT:TT | 0.01 | 2.010 | 1.327~3.046 | <0.001 | 2.123 | 1.439~3.132 |
使用SHEsis在线分析软件首先进行连锁不平衡(LD)分析,参照图 1中分析结果,上述3位点间|D′|均≥0.9,可选择 rs7955866-rs13312756-rs3812822组成FGF23基因单体型。进一步单体型分析结果显示携带ACC单体型的人群CHD发病风险上升到2.074倍,该单体型在CHD组分布频率明显高于对照组(P < 0.001),见表 6。
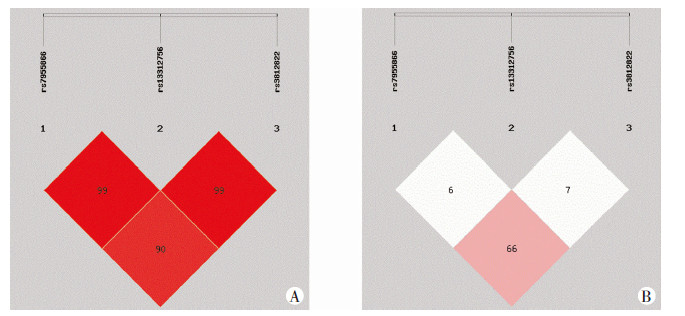 |
| A:LD分析: |D′|×100(图示数值);B:LD分析:R2×100 (图示数值) 图 1 FGF23基因分型结果连锁不平衡(LD)分析 [配色方案:|D′|(R2) < 0.5: 白色, 0.5 < |D′|(R2)≤ 0.7:粉色,0.7 < |D′|(R2)≤ 0.9:浅红,|D′|(R2)>0.9:深红] |
| 单体型 | 单体型分布频率 | P < /td> | OR | 95% CI | |
| CHD组 | 对照组 | ||||
| A C C | 0.190 | 0.102 | <0.001 | 2.074 | 1.391~3.091 |
| G C C | 0.044 | 0.040 | 0.796 | 1.092 | 0.560~2.129 |
| G C T | 0.536 | 0.582 | 0.191 | 0.834 | 0.635~1.095 |
| G G T | 0.216 | 0.265 | 0.100 | 0.768 | 0.561~1.052 |
FGF23作为成纤维细胞生长因子家族的最新成员,是一种来源于骨细胞和成骨细胞的激素样物质,其经典作用为调节机体磷酸盐和1α,25一羟基维生素D[1,25(OH)D]平衡,而目前研究发现其同时与诱导炎性反应,引起细胞增殖迁移有重要联系[11]。目前多项基础及临床研究表明FGF23浓度及活性的改变与冠心病、颈动脉斑块、缺血性脑卒中等动脉粥样硬化性疾病的发病风险有关,而机体FGF23基因多态性的差别能引起FGF23浓度及功能的改变,Takeo等[6]研究发现rs7955866位点等位基因差异可导致血清FGF23浓度改变进而与肺气肿发病相关,此外,Minna Pekkinen等[12]尽管在其试验中未发现该基因差异可引起FGF23浓度改变,但其与血清甲状旁腺素(P-PTH) 及尿磷/尿肌酐(U-Pi/U-Crea)相关,也证明了rs7955866是一个具有病理生理功能的位点。而上述研究均提示FGF23基因多态性与冠心病相关的可能性。
本次临床试验中rs7955866、rs13312756、rs3812822最小基因频率分别为0.161、0.239及0.191,该实验结果与人类基因组单体型图计划网站上公布的中国汉族人群SNP最小基因频率结果接近,且本实验对照组基因频率分布符合哈温平衡,具有重庆地区汉族人群群体代表性。通过本次病例对照研究发现rs7955866、rs3812822基因型及等位基因差异与冠心病发病独立相关,rs7955866-rs13312756-rs3812822单体型ACC可增加中国汉族人群冠心病患病风险。rs7955866和rs3812822位点分别位于FGF23基因的外显子和启动子区域,其中rs7955866(p.T239M)尚是该基因片段中的错义位点,其等位基因的改变引起翻译过程中苏氨酸(Thr)与蛋氨酸(Met)的转变[13],Minna Pekkinen等[12]的临床研究均证实此种错义改变可影响血清FGF23的表达程度及生物活性,rs7955866单核苷酸多态性可能亦因此与冠心病相关。启动子区rs3812822位点目前未见文献报道,其致病机制尚不清楚,Hoogendoorn等[14]调查研究显示启动子区域大约1/3的SNP位点可显著影响基因编码产物水平,这与启动子区域控制基因转录的起始时间和表达程度相关,rs3812822 C等位基因可能亦因此导致外周血FGF23水平升高而进一步促进动脉粥样硬化性疾病的发生与发展。人类基因组单体型相对于单个等位基因往往具有更好地个体代表性,能更显著地反应该基因多态性的致病风险[15, 16]。本次试验中,单体型分析结果显示rs7955866-rs13312756-rs3812822单体型ACC的致病风险(OR=2.074)高于rs7955866 A或rs3812822 C单个等位基因导致冠心病的发病风险(OR=2.015,OR=1.836),能更准确的预测冠心病的高危人群。
2015年大样本综合GWAS meta分析(n=185 000) 结果显示,目前已鉴定出50多个与CHD关联的基因和位点,而上述基因位点多与血脂浓度、脂蛋白代谢、高血压等CHD传统危险因素相关,FGF23等CHD非传统危险因素的研究仍需在冠心病分子遗传学研究领域进一步补充[17]。此外该次研究中大多数为欧裔人群,东亚人群只占6%,而样本种族来源可以严重影响研究结果。例如PHACTR1 rs12526463位点尽管在欧洲临床研究中证明是CHD易感基因,然而由于其最小基因频率(MAF)过低,在中国汉族人群中无法得到相应结论,再如中国汉族人群CHD易感基因位点rs6903956因其在欧裔人群中MAF明显升高一直无法得到欧裔人群的阳性结果,足见易感基因分析的种族特异性[18]。本次病例对照研究具有明显的种族性和地域性,可真实有效地反应重庆地区汉族人群的基因分布特点且消除他区域他种族的混杂影响。
综上所述,此次临床研究发现FGF23基因多态性与重庆地区汉族人群冠心病发病存在相关性,在遗传流行病学角度可以将FGF23基因作为冠心病发病的易感基因。据笔者所知,目前国内外尚未见FGF23基因与冠心病等动脉粥样硬化性疾病发病相关的报道,此次研究发现为冠心病等动脉粥样硬化性疾病的基因诊断及个体化治疗提供了更宝贵的分子遗传学依据。然而本次病例对照试验仅为证实FGF23基因多态性与冠心病相关性的研究,在后续的研究方案中,本课题组将拓展临床试验规模,在重庆汉族人群中进一步探讨FGF23基因多态性与血清FGF23浓度的相关性,并借助细胞及动物模型深入探讨FGF23基因多态性致病的相关作用机制,更全面深入的了解FGF23基因多态性致病机理。
| [1] | Six I, Okazaki H, Gross P, et al. Direct, acute effects of Klotho and FGF23 on vascular smooth muscle and endothelium. PLoS One, 2014, 9(4): e93423.DOI:10.1371/journal.pone.0093423 |
| [2] | di-Giuseppe R, Kuhn T, Hirche F, et al. Plasma fibroblast growth factor 23 and risk of cardiovascular disease: results from the EPIC-Germany case-cohort study. Eur J Epidemiol, 2015, 30(2): 131-141.DOI:10.1007/s10654-014-9982-4 |
| [3] | Ishii T, Gemma A, Kida K. Senescence is involved in the pathogenesis of chronic obstructive pulmonary disease through effects on telomeres and the anti-aging molecule fibroblast growth factor 23. Geriatr Gerontol Int, 2015, 15(7): 827-833.DOI:10.1111/ggi.12354 |
| [4] | Kim H J, Kim K H, Lee J, et al. Single nucleotide polymorphisms in fibroblast growth factor 23 gene, FGF23, are associated with prostate cancer risk. BJU Int, 2014, 114(2): 303-310.DOI:10.1111/bju.12396 |
| [5] | Meng Q H, Xu E, Hildebrandt M A, et al. Genetic variants in the fibroblast growth factor pathway as potential markers of ovarian cancer risk, therapeutic response, and clinical outcome. Clin Chem, 2014, 60(1): 222-232. |
| [6] | Miller C L, Assimes T L, Montgomery S B, et al. Dissecting the causal genetic mechanisms of coronary heart disease. Curr Atheroscler Rep, 2014, 16(5): 406.DOI:10.1007/s11883-014-0406-4 |
| [7] | Yu C, Yan Q, Fu C, et al. CYP4F2 genetic polymorphisms are associated with coronary heart disease in a Chinese population. Lipids Health Dis, 2014, 13: 83. |
| [8] | Fang H, Luo X, Wang Y, et al. Correlation between single nucleotide polymorphisms of the ACTA2 gene and coronary artery stenosis in patients with type 2 diabetes mellitus. Exp Ther Med, 2014, 7(4): 970-976.DOI:10.3892/etm.2014.1510 |
| [9] | Galan I, Valencia-Martin J L, Guallar-Castillon P, et al. Alcohol drinking patterns and biomarkers of coronary risk in the Spanish population. Nutr Metab Cardiovasc Dis, 2014, 24(2): 189-197. |
| [10] | Yin L, Cai W J, Liu C X, et al. Analysis of PTEN methylation patterns in soft tissue sarcomas by MassARRAY spectrometry. PLoS One, 2013, 8(5): e62971. |
| [11] | Ky B, Shults J, Keane M G, et al. FGF23 modifies the relationship between vitamin D and cardiac remodeling. Circ Heart Fail, 2013, 6(4): 817-824.DOI:10.1161/CIRCHEARTFAILURE.112.000105 |
| [12] | Pekkinen M, Laine C M, Makitie R, et al. FGF23 gene variation and its association with phosphate homeostasis and bone mineral density in Finnish children and adolescents. Bone, 2015, 71: 124-130.DOI:10.1016/j.bone.2014.10.013 |
| [13] | Tangri N, Alam A, Wooten E C, et al. Lack of association of Klotho gene variants with valvular and vascular calcification in Caucasians: a candidate gene study of the Framingham Offspring Cohort. Nephrol Dial Transplant, 2011, 26(12): 3998-4002. |
| [14] | Hoogendoorn B, Coleman S L, Guy C A, et al. Functional analysis of human promoter polymorphisms. Hum Mol Genet, 2003, 12(18): 2249-2254. |
| [15] | Xu H, Guan Y. Detecting local haplotype sharing and haplotype association. Genetics, 2014, 197(3): 823-838.DOI:10.1534/genetics.114.164814 |
| [16] | Alavi-Naini R, Salimi S, Sharifi-Mood B, et al. Association between the CD14 gene C-159T polymorphism and serum soluble CD14 with pulmonary tuberculosis. Int J Tuberc Lung Dis, 2012, 16(10): 1383-1387.DOI:10.5588/ijtld.11.0827 |
| [17] | CARDIoGRAMplusC4D Consortium . A comprehensive 1000 Genomes-based genome-wide association meta-analysis of coronary artery disease. Nat Genet, 2015, 47(10): 1121-1130.DOI:10.1038/ng.3396 |
| [18] | Wang F, Xu CQ, He Q, et al. Genome-wide association identifies a susceptibility locus for coronary artery disease in the Chinese Han population. Nat Genet, 2011, 43(4): 345-349.DOI:10.1038/ng.783 |



