利奈唑胺作为第1个恶唑烷酮类抗生素,能与细菌核糖体23S rRNA结合,阻止翻译起始物的形成,对革兰阳性球菌引起的感染,包括耐甲氧西林金黄色葡萄球菌(methicillin resistant Staphylococcus aureus,MRSA)和耐万古霉素肠球菌(vancomycine resistant enterococci,VRE)具有良好的抗菌活性。迄今为止,已明确的肠球菌利奈唑胺耐药机制包括23S rRNA的V区突变,L3、L4核糖体蛋白氨基酸突变以及位于质粒,可使核糖体发生甲基化的多重耐药cfr基因介导的耐药[1]。其中23S rRNA的V区G2576T突变为最常见的耐药相关突变,且耐药程度与突变拷贝数有关[2]。然而,近年来国内外均有文献报道有部分肠球菌对利奈唑胺呈现低水平耐药表型[最小抑菌浓度(minimal inhibitory concentration,MIC)4~16 μg/mL],并缺乏上述已知的耐药机制[3, 4, 5]。此部分肠球菌的MIC值是否能在利奈唑胺压力下增加,以及利奈唑胺压力是否能使其V区发生突变目前少见相关报道。
本研究利用临床分离的5株耐药机制未明的利奈唑胺耐药粪肠球菌(MIC 4~16 μg/mL),持续给予利奈唑胺进行体外诱导,观察其MIC值在抗生素压力下的变化,同时分析其耐药改变与23S rRNA的V区突变的联系,探讨利奈唑胺耐药肠球菌的相关耐药机制。
1 材料与方法 1.1 菌株来源从2011年8月至2013年11月,收集从重庆医科大学附属第一医院检验科临床标本分离的5株利奈唑胺耐药粪肠球菌,经Vitek 2 Compact全自动细菌鉴定和药敏检测系统进行菌种鉴定和药敏实验,纸片扩散法和微量肉汤稀释法测定利奈唑胺药敏结果。ATCC 29212(MIC 2 μg/mL)为本室保存。菌株经分离后纯培养,采用纸片法冷冻保存于-80 ℃。
采用PCR方法扩增5株菌的相关耐药基因,包括23S rRNA的V区基因,编码L3、L4核糖体蛋白基因及多重耐药cfr基因[6, 7]。扩增片段经测序后与标准野生株ATCC 29212基因序列进行对比分析。经检测,5株耐药菌与标准野生菌的23S rRNA的V区核酸以及L3、L4核糖体蛋白氨基酸序列一致,未检出cfr基因,表明5株菌不存在任何已知的利奈唑胺耐药机制。菌株基本情况见表 1。
| 细菌编号 | 标本类型 | 分离病区 | 利奈唑胺MIC (μg/mL) |
| N0551 | 尿 | 泌尿外科 | 8 |
| P4992 | 引流液 | 肝胆外科 | 8 |
| N4289 | 尿 | 妇科 | 16 |
| N2504 | 尿 | 妇科 | 16 |
| N8774 | 尿 | 泌尿外科 | 8 |
PCR扩增仪、凝胶成像仪(美国Bio-Rad 公司),细菌基因组DNA提取试剂盒(北京天根公司),质粒提取试剂盒(德国Qiagen公司),Mueller Hinton Broth (MHB)培养基、脑心浸液肉汤(Brain Heart Infusion,BHI)培养基(青岛日水生物公司),MH琼脂平板、血平板(郑州安图公司),利奈唑胺(美国Pfizer公司),利奈唑胺药敏纸片(英国Oxoid公司),利奈唑胺E-test试条(法国bioMérieux公司),BfaⅠ限制性内切酶(美国New England Biolabs公司)。
1.3 利奈唑胺体外诱导实验5株耐药菌经复苏后调制0.5麦氏单位接种于MH琼脂平板,贴利奈唑胺药敏纸片37 ℃孵育16~20 h,刮取纸片边缘细菌重新接种于新鲜MH琼脂平板并重复以上步骤。实验过程中采用利奈唑胺E-test试条检测细菌利奈唑胺MIC值变化。为检测诱导耐药的稳定性,诱导完成后细菌接种于无抗生素血平板传代10次,再次用E-test试条测定其利奈唑胺MIC值有无变化。
1.4 诱导菌23S rRNA的V区突变及L3、L4核糖体蛋白氨基酸突变分析将诱导完成后的细菌接种至血平板,37 ℃孵育16~20 h。挑取单个菌落接种于BHI肉汤,增菌后按照细菌基因组DNA提取试剂盒提取细菌基因组DNA,通过PCR扩增及测序方法检测23S rRNA的V区突变和L3、L4核糖体蛋白氨基酸突变。为进一步证实G2576T突变的发生,当23S rRNA的V区发生G2576T突变后,会产生新的酶切位点(C|UAG)[8]。利用PCR扩增诱导菌V区片段(429 bp)进行切胶纯化,纯化产物经酶切后将分别产生2个新的酶切片段(335 bp和94 bp)。取10 μL PCR纯化产物,1 μL BfaⅠ限制性内切酶,2 μL酶切反应液,7 μL无菌纯水,总体系20 μL 37 ℃过夜,最后取5 μL酶切产物采用30 mg/mL 琼脂糖电泳检测目的条带。
1.5 23S rRNA的V区G2576T 突变拷贝分析粪肠球菌含有4条不同的23S rRNA基因拷贝(rrlA~rrlD)[9]。将经测序及酶切证实含有G2576T突变的诱导菌利用PCR分别扩增4条不同的23S rRNA基因拷贝,所用引物包括1条通用上游引物5′-GGCGCTGGTGGGATACTA-3′和4条不同的下游引物rrlA: 5′-GGACGGTTATGAGCCGTC-3′,rrlB:5′-GCGA-TCTCCTGCGTGAC-3′,rrlC:5′-CCCTTCTTCAAGCTTA-TC-3′和rrlD:5′-CCACAGTGATTTTGCCCA-3′。随后将上述扩增产物切胶纯化回收,再次利用PCR技术扩增上述片段中所包含的V583区域,扩增产物经纯化后双向测序,与标准野生株进行序列对比分析。
2 结果 2.1 利奈唑胺诱导结果经过利奈唑胺多步体外诱导后,5株利奈唑胺耐药菌MIC值显著增高。其中有2株菌初始MIC值分别为8 μg/mL和16 μg/mL,在经过12、10代传代后E-test试条测定其MIC值超过256 μg/mL;其余3株菌经不同代数诱导后最低利奈唑胺MIC值为64 μg/mL。诱导基本情况见表 2。
| 细菌编号 | 初始MIC (μg/mL) | 传代次数 | 诱导后MIC (μg/mL) |
| N0551 | 8 | 12 | >256 |
| P4992 | 8 | 27 | 64 |
| N4289 | 16 | 10 | >256 |
| N2504 | 16 | 31 | 96 |
| N8774 | 8 | 17 | 192 |
通过PCR扩增和测序分析,结果显示N551、 N4289以及N8774发生了G2576T突变,未发现V区其他位点突变。P4992及N2504未发现G2576T及其他23S rRNA的V区突变。5株诱导菌均未发现核糖体蛋白L3、L4氨基酸改变。Bfa Ⅰ 酶切G2576T突变结果与测序结果一致,5株诱导菌G2576T酶切结果见图 1。
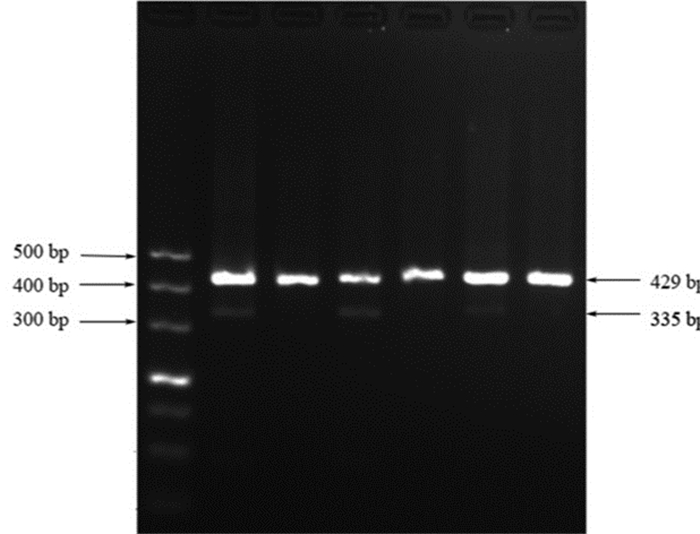
|
| M: 标准;1~6:分别为诱导后N551、P4992、N4289、N2504、N8774和ATCC 29212;1、3、5:G2576T突变株 图 1 粪肠球菌23S rRNA的V区扩增产物BfaⅠ酶切 |
通过PCR分别扩增粪肠球菌4条不同的23S rRNA基因拷贝,测序分析N551、N4289以及N8774的G2576T突变的拷贝数。测序结果发现粪肠球菌4个23S rRNA基因拷贝(rrlA~rrlD)均可发生G2576T突变。 5株菌中N0551和N4289的MIC值超过256 μg/mL,2株菌除了在rrlD拷贝的23S rRNA基因发生了1个完整的G2576T突变,还分别在rrlC和rrlA的23S rRNA基因存在野生型及突变型混合的现象,测序图显示为G/T完全重叠混合双峰。而MIC为192 μg/mL 的N8774诱导菌仅在rrlB的23S rRNA基因发生了 G2576T突变。测序结果见图 2,突变拷贝数变化见表 3。
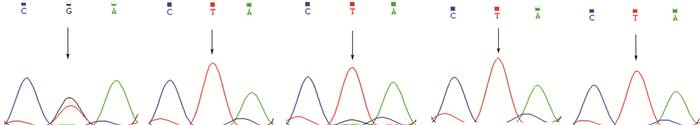
|
| A:N0551 rrlC的2 576位测定,G/T混合双峰;B:N0551 rrlD的2576位测定,T单峰;C: N4289 rrlA的2576位测定,G/T混合双峰;D:N4289 rrlA的2576位测定, T单峰;E: N8774 rrlB的2576位测定,T单峰 ↑:示细菌23S rRNA的2576位核苷酸 图 2 肠球菌 4条23S rRNA 基因拷贝2576位测序结果 |
| 菌株 | 利奈唑胺MIC | 23S rRNA拷贝基因在2 576位置检测结果 | G2576T突变的总拷贝数目 | |||
| rrlA | rrlB | rrlC | rrlD | |||
| N0551 | >256 μg/mL | G | G | G/T | T | 1.5/4 |
| P4992 | 64 μg/mL | NT | NT | NT | NT | 0/4 |
| N4289 | >256 μg/mL | G/T | G | G | T | 1.5/4 |
| N2504 | 96 μg/mL | NT | NT | NT | NT | 0/4 |
| N8774 | 192 μg/mL | G | T | G | G | 1/4 |
| NT:未测定 | ||||||
近年来,肠球菌已成为医院获得性感染的主要病原菌之一,其耐药性也不断增加。利奈唑胺的主要抑菌机制是与肠球菌23S rRNA结合,通过抑制细菌蛋白合成而产生抑菌作用,其中V583区域的点突变可以直接导致利奈唑胺对核糖体的亲和力降低而形成细 菌耐药。国内外常见的V区突变点报道包括了G2576T 、T2504C、G2505T以及C2610G等点突变[1],且其中G2576T突变与利奈唑胺细菌耐药密切相关。然而近年来,多地区均有文献报道有部分肠球菌对利奈唑胺产生耐药却缺乏明确的耐药机制,其共同的特点是利奈唑胺MIC值均不超过16 μg/mL[3, 4, 5]。
有体外诱导利奈唑胺敏感肠球菌显示随着利奈唑胺抗生素压力的增大,细菌利奈唑胺MIC值将增高并产生耐药,且耐药主要与23S rRNA的V区突变有关[10]。然而对于缺乏明确耐药机制,且MIC值不超过16 μg/mL的利奈唑胺耐药粪肠球菌是否会在利奈唑胺的持续作用下发生MIC值的改变,以及产生变化的耐药机制目前还缺乏深入的研究。
本研究通过体外诱导5株利奈唑胺耐药机制不明的耐药粪肠球菌(MIC 8~16 μg/mL),测定其MIC改变发现随着利奈唑胺抗生素压力的累积,耐药菌的MIC值迅速增加,所有诱导菌突破了原有的8 μg/mL和16 μg/mL耐药水平,且其中最高MIC值可超过256 μg/mL。该结果提示长期、持续利奈唑胺作用可诱导低水平、耐药机制未明的粪肠球菌MIC值迅速增加,从而产生更高水平的耐药。
与此同时,随着细菌MIC值增加,诱导菌23S rRNA的V区耐药基因检测结果显示细菌在该域发生了G2576T突变。有研究表明,在肠球菌利奈唑胺耐药中,多个23S rRNA基因拷贝可在利奈唑胺压力下发生V区的突变。通过基因同源重组,突变型将不断替代野生型从而产生抗生素耐药,且细菌耐药程度与突变拷贝数目有关[11]。本研究通过分别扩增粪肠球菌4条不同的23S rRNA基因发现随着细菌利奈唑胺MIC值的升高,V区G2576T突变拷贝数也在同步增加。MIC值超过256 μg/mL的细菌比MIC值为192 μg/mL 的细菌分别在rrlA以及rrlC拷贝多1条突变型和野生型混合的23S rRNA基因拷贝,显示为对抗抗生素压力,细菌正在突变以产生更高级更稳定的耐药。而更低MIC值细菌则未发现G2576T突变。表明利奈唑胺高水平耐药主要与23S rRNA基因V区G2576T突变相关,且突变拷贝数量与MIC值增高呈正相关。
利奈唑胺可结合到肠球菌转肽中心(PTC),通过影响转肽中心的结构及稳定性发挥抑菌作用。除了V583区域,与转肽中心距离相近位置的L3、L4核糖体蛋白同样可以通过突变引起氨基酸序列改变从而造成利奈唑胺耐药[12, 13]。文献[14]报道核糖体蛋白L3、L4的突变主要与肠球菌低MIC(4~8 μg/mL)耐药相关。本研究5株利奈唑胺耐药菌经体外诱导成高MIC耐药后并没有发生核糖体蛋白L3、L4氨基酸突变,结果与文献报[14]道一致,进一步提示利奈唑胺高水平耐药主要与23S rRNA基因V区突变有关。
然而,在诱导过程中,有2株耐药菌MIC被诱导至64 μg/mL和96 μg/mL后依然缺乏明确的耐药机制,提示可有其他未明确的耐药机制负责细菌耐药。文献[15]报道利奈唑胺耐药除了靶位点基因突变及cfr基因的介导耐药外,还可能与细菌外排泵有关。在研究中,我们曾采用4种常见的外排泵抑制剂:利血平、羰基氰氯苯腙(CCCP)、维拉帕米以及兰索拉唑处理耐药菌,用以观察利奈唑胺MIC值的变化。结果发现外排泵抑制剂使用前后细菌MIC值无明显变化。该现象提示耐药菌可能并不具有能外排利奈唑胺的外排泵,或者如果耐药菌存在可以外排利奈唑胺的外排泵,该外排泵将不能被以上抑制剂所抑制。
目前肠球菌利奈唑胺耐药机制尚未完全明确,深入探索并明确其耐药机制对于感染的防控和治疗具有重要意义。本研究通过体外诱实验发现利奈唑胺可诱导耐药机制未明的利奈唑胺耐药粪肠球菌MIC值迅速增加,且其耐药改变与V区G2576T突变密切相关。采用外膜蛋白提取并进行比较蛋白质组分析将有望筛选出潜在的外排泵蛋白,从而发现新的耐药相关靶标。
| [1] | Long K S, Vester B. Resistance to linezolid caused by modifications at its binding site on the ribosome[J]. Antimicrob Agents Chemother, 2012, 56(2): 603-612. DOI: 10.1128/AAC.05702-11 |
| [2] | Mendes R E, Deshpande L M, Jones R N. Linezolid update: stable in vitro activity following more than a decade of clinical use and summary of associated resistance mechanisms[J]. Drug Resist Updat, 2014, 17(1/2): 1-12. DOI: 10.1016/j.drup.2014.04.002 |
| [3] | Jones R N, Ross J E, Bell J M, et al . Zyvox Annual Appraisal of Potency and Spectrum program: linezolid surveillance program results for 2008[J]. Diagn Microbiol Infect Dis, 2009, 65(4): 404-413. DOI: 10.1016/j.diagmicrobio.2009.10.001 |
| [4] | Patel S N, Memari N, Shahinas D, et al . Linezolid resistance in Enterococcus faecium isolated in Ontario, Canada[J]. Diagn Microbiol Infect Dis, 2013, 77(4): 350-353. DOI: 10.1016/j.diagmicrobio.2013.08.012 |
| [5] | Ross J E, Farrell D J, Mendes R E, et al . Eight-year (2002-2009) summary of the linezolid (Zyvox(R) Annual Appraisal of Potency and Spectrum; ZAAPS) program in European countries [J]. J Chemother, 2011, 23(2): 71-76. |
| [6] | Bourgeois-Nicolaos N, Massias L, Couson B, et al . Dose dependence of emergence of resistance to linezolid in Enterococcus faecalis in vivo [J]. J Infect Dis, 2007, 195(10): 1480-1488. |
| [7] | Diaz L, Kiratisin P, Mendes R E, et al . Transferable plasmid-mediated resistance to linezolid due to cfr in a human clinical isolate of Enterococcus faecalis [J]. Antimicrob Agents Chemother, 2012, 56(7): 3917-3922. DOI: 10.1128/AAC.00419-12 |
| [8] | Qi C, Zheng X, Obias A, et al . Comparison of testing methods for detection of decreased linezolid susceptibility due to G2576T mutation of the 23S rRNA gene in Enterococcus faecium and Enterococcus faecalis [J]. J Clin Microbiol, 2006, 44(3): 1098-1100. |
| [9] | Paulsen I T, Banerjei L, Myers G S, et al . Role of mobile DNA in the evolution of vancomycin-resistant Enterococcus faecalis [J]. Science, 2003, 299(5615): 2071-2074. |
| [10] | 李多云, 余治健, 徐俊, 等. 利奈唑胺体外诱导粪肠球菌耐药株23S rRNA V区基因突变位点分析[J]. 中国病原生物学杂志, 2014, 9(4): 316-317,375. |
| [11] | Lobritz M, Hutton-Thomas R, Marshall S, et al . Recombination proficiency influences frequency and locus of mutational resistance tolinezolid in Enterococcus faecalis [J]. Antimicrob Agents Chemother, 2003, 47(10): 3318-3320. |
| [12] | Bosling J, Poulsen S M, Vester B, et al . Resistance to the peptidyl transferase inhibitor tiamulin caused by mutation of ribosomal protein l3[J]. Antimicrob Agents Chemother, 2003, 47(9): 2892-2896. |
| [13] | Locke J B, Hilgers M, Shaw K J. Novel ribosomal mutations in Staphylococcus aureus strains identified through selection with the oxazolidinones linezolid and torezolid (TR-700)[J]. Antimicrob Agents Chemother, 2009, 53(12): 5265-5274. DOI: 10.1128/AAC.00871-09 |
| [14] | Chen H, Wu W, Ni M, et al . Linezolid-resistant clinical isolates of enterococci and Staphylococcus cohnii from a multicentre study in China: molecular epidemiology and resistance mechanisms[J]. Int J Antimicrob Agents, 2013, 42(4): 317-321. DOI: 10.1016/j.ijantimicag.2013.06.008 |
| [15] | Floyd J L, Smith K P, Kumar S H, et al . LmrS is a multidrug efflux pump of the major facilitator superfamily from Staphylococcus aureus[J]. Antimicrob Agents Chemother, 2010, 54(12): 5406-5412. DOI: 10.1128/AAC.00580-10 |



