大肠埃希菌(Escherichia coli,E.coli)是临床分离率最高的革兰阴性菌,在医院获得性及社区获得性感染中较为常见。形成生物膜的大肠埃希菌可以逃避抗菌药物和宿主免疫系统的攻击,具有高度的耐药性,并可在生物膜解离后导致新的感染,这一过程使感染趋于慢性化且难于控制[1, 2]。H-NS是一种小组蛋白相似蛋白,广泛存在于革兰阴性菌中[3]。H-NS不仅参与基因组装配,而且在基因调控中也起关键作用[4, 5]。H-NS调控的许多基因参与细菌的调整来适应渗透性、pH等环境变化产生的应激[6]。H-NS可以抑制大肠埃希菌毒力基因或耐药性基因的转录,不同转录调控子通过减轻或抵消H-NS的作用来进行基因表达。有研究表明,H-NS能够温度依赖性地调节大肠埃希菌生物膜形成相关基因[7]。hns基因缺失能够促进胸膜炎肺炎放线杆菌生物膜形成[8],然而,H-NS在大肠埃希菌生物膜形成中的作用目前尚少见报道。因此,本研究以大肠埃希菌MG1655为研究对象,构建大肠埃希菌hns基因敲除株,并分析其细菌生物膜形成能力的变化。
1 材料与方法 1.1 实验材料 1.1.1 质粒与菌株大肠埃希菌BL21/pKD46由北京军事医学院病原微生物室周冬生馈赠。质粒pKD3和pCP20由上海意泓生物技术有限公司提供。大肠埃希菌MG1655为西南医院国家药物临床试验机构保存菌株。
1.1.2 试剂氨苄青霉素(ampicillin)、氯霉素(chloramphenicol)、DNA胶回收纯化试剂盒、PCR 产物回收试剂盒和DNA marker 购自北京天根生化科技有限公司,L-(+)-阿拉伯糖、琼脂糖购自上海生工生物工程有限公司,质粒提取试剂盒购自Promega公司,PCR引物合成和测序由上海英骏生物技术有限公司完成,2×Es Taq Master Mix购自北京康为世纪生物科技有限公司,结晶紫购自Sigma公司,冰醋酸购自重庆川东化工有限公司,苯酚购自北京鼎国生物公司,浓硫酸购自成都科龙化学试剂有限公司。
1.2 方法 1.2.1 E.coli MG1655/pKD46菌株的构建将携带pKD46质粒的大肠埃希菌实验室标准株BL21接种于5 mL LB肉汤(Amp终浓度为100 μg/mL)中,30 ℃过夜培养。然后用质粒提取试剂盒提取pKD46质粒DNA。取6 μL pKD46 DNA加入90 μL MG1655感受态细胞中,冰上预冷10 min后,加入到电击杯,于25 μF,200 Ω,2.5 kV的电击参数下电击。电击完后,立即加入900 μL的LB肉汤悬浮细胞,30 ℃培养1 h。取100 μL涂氨苄抗性LB平板,30 ℃培养至可见清晰单克隆。挑取单克隆转涂于氨苄抗性LB平板,培养适当时间后,刮取少量细菌,洗脱至无菌水中,于95 ℃变性10 min,10 000×g室温离心2 min,取5 μL作为模板进行PCR鉴定。pKD46质粒鉴定引物为:pKD46-正义链5′-GTCTGCTATGTGGTGCTATCTG-3′;pKD46-反义链5′-GCCAACTTACTTCTGACAACGA-3′。扩增阳性为重组克隆E. coli MG1655/pKD46。
1.2.2 打靶片段的扩增以pKD3质粒为模版进行打靶片段的扩增,扩增两侧含FRT 位点的氯霉素抗性基因点(FLP重组酶识别位点)。同源臂打靶序列引物:hns-正义链: 5′-AAAAAATCCCGCCGCTGGCGGGATTTTAAGCAAGTGCAATCTACAAAAGACATATGA-ATATCCTCCTTAGTTCCTATTC-3′;hns-反义链: 5′-AT-TTAAGGCTCTATTATTACCTCAACAAACCACCCCAAT-ATAAGTTTGAGATTACTACAGAGCTGCTTCGAAGTTC-CTA-3′。扩增条件:95 ℃ 5 min,(95 ℃ 30 s、65 ℃ 45 s、72 ℃ 60 s)×21个循环,(95 ℃ 30 s、55 ℃ 45 s、72 ℃ 60 s)×15个循环,72 ℃ 7 min。PCR产物经1%琼脂糖凝胶电泳,切割目的条带进行胶回收纯化。
1.2.3 hns突变株的构建将E.coli MG1655/pKD46接种于LB肉汤(含150 μg/mL Amp)中,30 ℃培养过夜。转接于100 mL的含氨苄抗性LB肉汤中,30 ℃培养至D(600)为0.6~0.8,并在培养终止前2 h加入L-(+)-阿拉伯糖,终浓度为1 mmol/L。4 ℃、4 000 r/min 离心5 min,收集菌体。用预冷10%甘油离心洗涤3次,浓缩100倍成 1 mL的感受态细胞,每管90 μL,于-70 ℃冻存备用。取100 ng纯化的打靶片段加入MG1655/pKD46感受态细胞中,轻微混匀,于冰上继续放置15 min。然后转入电击杯,于25 μF,200 Ω,2.5 kV的电击参数下电击。电击完后立即加入 900 μL的LB[含L-(+)-阿拉伯糖,终浓度1 mmol/L]。 于37 ℃、160 r/min培养2 h。取500 μL涂布LB平板(含34 μg/mL氯霉素)。37 ℃培养至可见清晰单克隆。然后用PCR筛选鉴定重组克隆,敲除株鉴定引物为: hns-out-正义链5′-CACCCTTGGCACGGAATTTAA-3′; hns-out-反义链5′-CCGCGTCTTTTCTGGCTAATTTT-3′。
1.2.4 MG1655/Δhns/Cm菌株中氯霉素抗性基因的删除DH5α/pCP20接种于5 mL的LB肉汤(含 100 μg/mL Amp,34 μg/mL氯霉素)中,30 ℃、220 r/min 培养过夜。然后用质粒提取试剂盒提取pCP20质粒。取1 μL pCP20质粒加入90 μL MG1655/Δhns::Cm感受态细胞中,冰上预冷10 min后,加入到电击杯,于25 μF,200 Ω,2.5 kV的电击参数下电击。电击完后,立即加入 900 μL的LB肉汤悬浮细胞,30 ℃、160 r/min 培养复苏2 h。取10 μL涂LB平板(含50 μg/mL Amp),30 ℃培养至单克隆形成,使质粒表达重组酶,删除氯霉素抗性基因。挑选生长良好的单个克隆划线接种LB平板(无抗性),42 ℃培养过夜,使质粒丢失。随机选取7个克隆,用hns-out-正义链和hns-out-反义链这对引物再次进行PCR扩增及测序鉴定。大肠埃希菌MG1655 hns基因敲除流程见图 1。
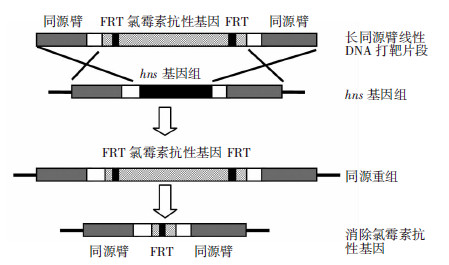 |
| 图 1 大肠埃希菌MG1655 hns基因敲除流程示意图 |
生物膜形成能力检测采用 96孔板结晶紫染色法。大肠埃希菌划线接种于LB平板培养过夜。挑单菌落于 10 mL LB培养基中,37 ℃、150 r/min培养约18 h。将过夜培养物稀释至0.5麦氏单位,再用LB肉汤稀释100倍。每孔加100 μL菌液和100 μL LB肉汤,每株菌设置3个复孔,37 ℃孵育 24 h。小心去除培养基,用灭菌 PBS液冲洗3次,风干后采用1%结晶紫染色10 min,自来水冲洗至空白对照孔未见明显颜色。通风阴凉处倒置自然晾干,用100 μL 30%醋酸溶解,酶标仪590 nm处测定光密度值[D(590)]。
1.2.6 大肠埃希菌hns基因敲除株胞外多糖含量测定采用苯酚-硫酸法检测细菌胞外多糖[9]。按1%接种量将大肠埃希菌过夜培养物转接于100 mL LB肉汤中,在37 ℃、150 r/min条件下培养8 h。然后将培养好的菌液倒入50 mL离心管,4 ℃、4 000 r/min下离心15 min,弃上清。蒸馏水重悬沉淀后加入300 μL 37%甲醛溶液,4 ℃保存3 h。然后加入20 mL 1 mol/L NaOH,4 ℃再保存3 h。4 ℃、12 000 r/min离心20 min,弃上清,留取沉淀待查。先用标准葡萄糖溶液为底物制作标准曲线,然后将上述沉淀稀释后按同样的方法进行显色,酶标仪490 nm处测定光密度值[D(490)],并根据标准曲线计算出相应的多糖含量。
1.3 统计学分析细菌生物膜形成和胞外多糖含量的数据均以 x±s表示,差异性采用SPSS 17.0统计软件进行t检验。检验水准α=0.01。
2 结果 2.1 MG1655/pKD46菌株的构建将pKD46质粒转化进入大肠埃希菌MG1655,氨苄抗性平板筛选阳性克隆。重组克隆通过PCR扩增进行鉴定。鉴定结果如图 2所示,挑取的重组克隆扩增结果均为阳性。
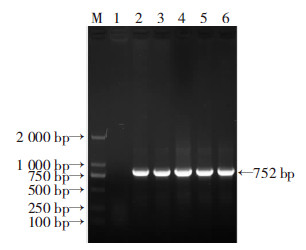 |
| M:DNA标准;1:阴性对照;2~6:MG1655/pKD46重组克隆 图 2 E.coli MG1655/pKD46重组克隆PCR扩增结果 |
以pKD3质粒为模板进行突变盒的扩增,获得氯霉素抗性基因片段,结果如图 3所示。
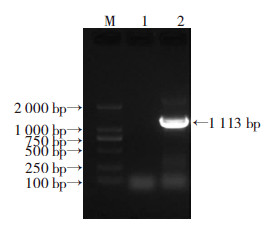 |
| M:DNA标准;1:阴性对照;2:hns基因打靶片段 图 3 氯霉素抗性基因片段PCR扩增结果 |
将打靶基因片段转入MG1655/pKD46,氯霉素抗性平板筛选出发生同源重组的克隆。hns基因敲除前扩增产物长度为774 bp,而hns被氯霉素基因替换后扩增长度变为1 364 bp。重组克隆通过PCR扩增进行鉴定。鉴定结果如图 4所示,挑取的克隆为正确发生重组的转化体。
 |
| M:DNA标准;1:重组克隆扩增结果;2:MG1655原始菌株扩增结果 图 4 E.coli MG1655 hns基因敲除PCR鉴定结果 |
随机选取7个克隆,用hns-out-正义链和hns-out-反义链这对引物再次进行PCR扩增鉴定,氯霉素抗性基因消除的克隆产物长度缩短为434 bp。结果如图 5所示,7个克隆全部为氯霉素抗性删除的阳性克隆,经测序验证序列正确。
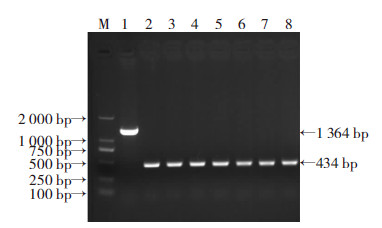 |
| M:DNA标准;1:阴性对照;2~8:氯霉素抗性基因消除克隆 图 5 E.coli MG1655/Δhns/Cm氯霉素抗性基因消除PCR鉴定结果 |
96孔板结晶紫染色法观察大肠埃希菌MG1655和hns基因缺失株生物膜形成能力结果如图 6所示。大肠埃希菌MG1655和MG1655 △hns菌株光密度值分别为(1.47±0.11)和(0.94±0.12)。MG1655 △hns株生物膜形成能力显著低于MG1655菌株(P<0.01)。
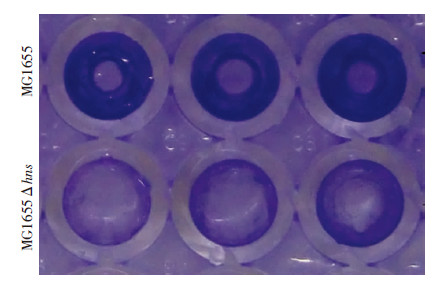 |
| 1~3: 3个平行复孔 图 6 结晶紫染色法观察大肠埃希菌MG1655和hns基因缺失株生物膜形成能力 |
大肠埃希菌MG1655和hns基因缺失株胞外多糖含量测定结果显示,大肠埃希菌MG1655和MG1655 △hns菌株胞外多糖含量分别为(363.98±16.22)μg和(283.07±18.52)μg。hns基因敲除后细菌产胞外多糖的能力显著减弱(P<0.01)。
3 讨论本研究MG1655 hns基因敲除株的构建采用Red 重组系统,该系统利用λ噬菌体3个蛋白质 Exo、Bet和Gam,Gam抑制大肠埃希菌的RecBCD核酸外切酶V活性,使外源线形DNA不至立即被降解,Exo和Bet引导线性片段与同源区发生重组置换,比传统的同源重组方法效率高几十倍[10]。本研究用含同源臂的 PCR片段,通过同源重组替换掉hns基因,构建了hns缺失的菌株,并用pCP20质粒重组酶删除氯霉素抗性基因,完成无痕敲除。该方法具有高效简便的特点,并且由于删除了抗性基因,对细菌的生物学性状的影响降到最小,有利于进一步研究的开展。
H-NS是一个小类核相关蛋白和转录抑制物,在肠道细菌中表达量丰富[11]。H-NS参与细菌拟核基因装配,H-NS与双链DNA分子结合,在细胞中紧密缠绕成较致密的不规则小体[3]。在大肠埃希菌中,H-NS是由134个氨基酸残基(大约15.4×103)组成的小蛋白。它由N-末端寡聚化结构域和C-末端DNA结合结构域通过接头连接组成[12]。H-NS在调节基因转录、沉默水平获得基因以及包装细菌类核中起关键作用[13, 14, 15]。在细菌进化的过程中,很多毒力基因或耐药性基因都是通过基因水平转移获得的,H-NS能有选择地结合到这些“外源基因”启动子区富含AT的碱基序列上,从而对靶基因的转录起抑制作用[13]。这种负调控机制避免了外源基因的过表达,也为这些外源遗传物质整合到细菌基因组并稳定遗传提供了先决条件。H-NS突变株缺乏寡聚化结构域不再能够抑制基因表达。hns突变株可以显示出多向性的表型,如生长滞后、运动性的缺失、增强对酸应激和渗透压的耐受性和降低毒性[11, 16],为研究H-NS对大肠埃希菌生物膜形成的影响,我们构建了大肠埃希菌hns基因缺失株。
本实验结果显示大肠埃希菌MG1655 hns缺失株生物膜形成能力显著低于野生株,与胸膜炎肺炎放线杆菌hns突变株生物膜形成能力增加结果相反[8],其原因可能是菌株的不同,调控的基因不同。研究发现大肠埃希菌的细胞外多糖减少,提示hns可能通过抑制胞外多糖合成基因icaA的表达而抑制大肠埃希菌生物膜形成。由于H-NS具有广泛的调节作用,所以我们推测H-NS可能还对调控生物膜形成的其他相关基因具有调节作用,而生物膜形成能力降低和胞外多糖的减少可能是H-NS调控生物膜相关基因整体的一个综合的结果,目前相关的研究报道较少,构建敲除株后将为进一步研究其机制奠定基础。
| [1] | Nguyen D, Joshi-Datar A, Lepine F, et al. Active starvation responses mediate antibiotic tolerance in biofilms and nutrient-limited bacteria. Science, 2011, 334(6058): 982-986. DOI:10.1126/science.1211037 |
| [2] | Kawarai T, Furukawa S, Narisawa N, et al. Biofilm formation by Escherichia coli in hypertonic sucrose media. J Biosci Bioeng, 2009, 107(6): 630-635. DOI:10.1016/j.jbiosc.2009.01.018 |
| [3] | Tendeng C, Bertin P N. H-NS in Gram-negative bacteria: a family of multifaceted proteins. Trends Microbiol, 2003, 11(11): 511-518. |
| [4] | Dorman C J. Horizontally acquired homologues of the nucleoid-associated protein H-NS: implications for gene regulation. Mol Microbiol, 2010, 75(2): 264-267. DOI:10.1111/j.1365-2958.2009.06996.x |
| [5] | Stoebel D M, Free A, Dorman C J. Anti-silencing: overcoming H-NS-mediated repression of transcription in Gram-negative enteric bacteria. Microbiology, 2008, 154(Pt 9): 2533-2545. DOI:10.1099/mic.0.2008/020693-0 |
| [6] | Hommais F, Krin E, Laurent-Winter C, et al. Large-scale monitoring of pleiotropic regulation of gene expression by the prokaryotic nucleoid-associated protein, H-NS. Mol Microbiol, 2001, 40(1): 20-36. |
| [7] | White-Ziegler C A, Davis T R. Genome-wide identification of H-NS-controlled, temperature-regulated genes in Escherichia coli K-12. J Bacteriol, 2009, 191(3): 1106-1110. DOI:10.1128/JB.00599-08 |
| [8] | Dalai B, Zhou R, Wan Y, et al. Histone-like protein H-NS regulates biofilm formation and virulence of Actinobacillus pleuropneumoniae. Microb Pathog, 2009, 46(3): 128-134. DOI:10.1016/j.micpath.2008.11.005 |
| [9] | 陈盛, 余加林, 何念海, 等. 巯乙磺酸钠对大肠杆菌生物膜早期黏附及胞外聚合物的影响. 第三军医大学学报, 2011, 33(24): 2554-2557. |
| [10] | Copeland N G, Jenkins N A, Court D L. Recombineering: A powerful new tool for mouse functional genomics. Nat Rev Genet, 2001, 2(10): 769-779. DOI:10.1038/35093556 |
| [11] | Atlung T, Ingmer H. H-NS: a modulator of environmentally regulated gene expression. Mol Microbiol, 1997, 24(1): 7-17. |
| [12] | Arold S T, Leonard P G, Parkinson G N, et al. H-NS forms a superhelical protein scaffold for DNA condensation. 1Proc Natl Acad Sci U S A, 2010, 107(36): 15728-15732. DOI:10.1073/pnas.006966107 |
| [13] | Fang F C, Rimsky S. New insights into transcriptional regulation by H-NS. Curr Opin Microbiol, 2008, 11(2): 113-120. DOI:10.1016/j.mib.2008.02.011 |
| [14] | Ali S S, Xia B, Liu J, et al. Silencing of foreign DNA in bacteria. Curr Opin Microbiol, 2012, 15(2): 175-181. DOI:10.1016/j.mib.2011.12.014 |
| [15] | Navarre W W, McClelland M, Libby S J, et al. Silencing of xenogeneic DNA by H-NS-facilitation of lateral gene transfer in bacteria by a defense system that recognizes foreign DNA. Genes Dev, 2007, 21(12): 1456-1471. DOI:10.1101/gad.1543107 |
| [16] | Bertin P, Hommais F, Krin E, et al. H-NS and H-NS-like proteins in Gram-negative bacteria and their multiple role in the regulation of bacterial metabolism. Biochimie, 2001, 83(2): 235-241. |



