数字PCR(digital PCR)是一种新型的DNA分子定量检测技术,无需标准物质即可直接测量样品中DNA靶分子拷贝数[1]。目前的数字PCR技术基于芯片式(chip digital PCR,cdPCR)和微滴式(droplet digital PCR,ddPCR)两种平台。微滴数字化PCR(ddPCR)可通过微滴发生器对PCR反应体系进行微滴化处理,将DNA样本随机分装进20 000个独立的纳升级油包水液滴,每个微滴都是独立的PCR微反应器;乳液PCR反应结束后,使用微滴检测器逐个检测每个微滴的荧光信号,根据荧光信号的强弱,将微滴分为阳性微滴和阴性微滴,阴性微滴不含靶标DNA分子,阳性微滴中含有至少1个拷贝的靶标DNA分子。阳性微滴的数量跟靶标DNA含量正相关,两者的关系符合泊松分布,即:P=1-e(-λ)。其中P为阳性微滴的比例,e为自然常数,λ为平均每个微滴中的靶标DNA拷贝数。ddPCR软件根据阳性微滴的比例自动计算出目标DNA分子的拷贝数浓度(copies/μL)[2]。ddPCR已被广泛运用到科学研究及临床应用,如突变检测,拷贝数变异分析以及特定核酸种类的量化[3]。ddPCR有助于肿瘤潜在基因突变的识别,尤其是具有细胞异质性的临床样品;还可用于分析体细胞突变的等位基因分数(allelic fraction),判断等位基因是否具有差异表达[4]。等位基因分数是指带有突变等位基因在样本中的比例,取决于肿瘤纯度,肿瘤局部绝对拷贝数及多重性,以及癌细胞的比例[5]。由于其高度敏感性及对靶分子的绝对定量能力,ddPCR成为替代定量PCR (qPCR) 的选择之一[6]。ddPCR可区分10%或更低的基因表达差异,而对于小于两倍的基因表达差异,qPCR的检测结果准确性明显降低。另外,ddPCR可在1/10 000的频率下识别等位基因,而qPCR难以检测频率小于1%的等位基因。
在前期研究中,我们对临床收集的13例乙型病毒性肝炎相关性肝细胞肝癌患者的肝癌组织、癌旁硬化组织以及外周血进行了全外显子组测序。在观察肝癌与非癌癌组织的非沉默突变率(prevalence of non-silent mutations)时发现1例患者的MSH2基因第10位密码子发生无义突变,从而导致MSH2蛋白翻译提前终止,引起该患者基因组错配修复 (DNA mismatch repair)缺陷。由于下一代测序本身存在测序错误(sequencing error),需要对新识别的体细胞突变进行验证。目前,利用ddPCR对肿瘤样本中通过下一代测序数据所识别的体细胞突变位点的分析工作报道较少。因此,本研究利用ddPCR技术(Bio-radQX100TM)验证该基因的无义突变,并对该患者的癌组织以及癌旁正常组织的基因组DNA进行了分析,评估突变等位基因在肝癌组织中的等位分数。ddPCR作为DNA绝对定量检测技术在肿瘤基因组变异的绝对定量检测方面应用潜力较大。本研究基于ddPCR建立了体细胞突变的验证方法,为临床肿瘤患者体细胞突变研究提供新方法。
1 材料与方法 1.1 临床病例该患者为2013年4月中国医学科学院北京协和医院肝胆外科诊断为原发性肝细胞肝癌,有乙型肝炎史;对该患者实施了原发性肝细胞肝癌切除术,并从该患者身上提取了肝脏组织标本,包括肝癌组织和邻近的肝正常组织。两名病理学家 (北京协和医院病理科) 确认了切除样品的组织学诊断,即肝细胞癌和肝硬化组织。患者签订了知情同意书,该研究获得了重庆医科大学第二附属医院IRB委员会的认可。
1.2 DNA提取使用QIAamp提取试剂盒(Qiagen®,巴伦西亚,CA)从肝癌及肝硬化组织中提取了基因组DNA,具体步骤见试剂盒说明书。
1.3 MSH2引物和ddPCR MGB(minor groove binder) 探针设计对于MSH2中的无义突变,我们设计了相应的引物和探针,并利用UCSC基因组浏览器验证了其特异性,本研究分析位点的扩增片段长度为112 bp。上游引物序列:5′-CGCGCATTTTCTTCAACCA-3′,下游引物序列:5′-CCTGAAAGAAGCGCACGAA-3′。引物扩增片段长度为112 bp,对应的TaqMan-MGB探针包括由突变型探针:5′-FAM-AGGAGACGCTGTAGTT-MGB-3′,用于识别突变等位基因(T);野生型探针5′-VIC-AAGGAGACGCTGCAGT-MGB-3′,用于识别野生型等位基因(C)。引物探针均由Life Technologies公司合成。
1.4 ddPCR检测ddPCR检测在Bio-rad QX100TM微滴式数字PCR 仪上进行。实验流程按照厂家说明书。首先配制 20 μL 的ddPCR反应混合物,包含10 μL ddPCR反应预混液(Bio-rad® ddPCR supermix for probe),0.8 μL MSH2引物混合物(终浓度为900 nmol/L),1 μL探针混合物(各探针终浓度为500 nmol/L)和2 μL样品。随后将整个反应体系混合物以及70 μL液滴生成油(Bio-rad®) 分别加入到微滴发生卡(Bio-rad®)的相应孔内,放入到微滴发生器中制备微滴,耗时约2 min至2 min 30 s。微滴制备结束后,每个样品生成约20 000个微滴,将生成的油包水液滴转移到96孔PCR板 (Eppendorf) 中,在PX1热封仪(Bio-rad)上以金属膜封闭。然后利用T100热循环仪 (Bio-rad®) 进行PCR扩增,PCR程序为95 ℃预变性10 min,94 ℃变形3 s,58 ℃退火延伸60 s,共40个循环,最后98 ℃ 10 min,12 ℃保温。
PCR扩增完成后,将96孔板放到微滴分析仪(Bio-rad®),仪器会自动顺序读取分析每个样本的液滴。有荧光信号的微滴为阳性,记数为1,无荧光信号的为阴性,记数为0。软件记录每个样品里阳性和阴性微滴数。最终根据两种微滴的比例和泊松分布(Poisson)概率密度函数,QuantaSoft软件 (Bio-rad®)自动计算出样品中野生型DNA分子和突变型DNA分子的拷贝数浓度(copies/μL)和等位基因分数。
1.5 ddPCR计算拷贝数的方法对于双重ddPCR反应体系,单阳性微滴中含有且仅含有一种靶标分子,双阳性微滴即同时含有两种靶标分子,阴性微滴不含任何靶标DNA分子。在反应体系中,两种靶标核酸分子(比如野生型和突变型)彼此独立地随机分装进若干微滴中去。两种靶标分子的随机分布使得有些微滴仅含一种靶标分子,有些微滴仅含有另外一种靶标分子,有些微滴同时包裹到两种核酸分子,而有些微滴是空的,不含任何靶标分子。因此野生型DNA和突变型DNA的检测是彼此独立的。系统可以自动计算出野生型和突变型DNA的含量,等位基因的计算公式为:A/(A+B),其中A为突变型DNA分子的拷贝数,B为野生型DNA分子的拷贝数。在计算A和B的时候,就要把双阳性的微滴计算在内,比如FAM单阳性的微滴有 M个,VIC单阳性的微滴有N个,双阳性的微滴有L个,那么FAM阳性的微滴就是M+L个,VIC阳性的微滴为N+L个。
2 结果 2.1 1例原发性肝细胞肝癌患者MSH2基因一个新的无义突变的鉴定通过全外显子测序发现该患者的肝癌组织MSH2基因发生了一个无义突变(rs63751099,g.chr2:47630358 C>T,c.28 C>T,and p. Q10*)(图 1)。MSH2基因包括16个外显子,该突变位于第1外显子,导致密码子CAG (编码谷氨酰胺Q) 变成了终止密码子,从而导致该基因翻译提前终止。该突变仅在癌组织而不是肝硬化组织中检测出。NCBI dbSNP数据库显示该变异位点为三等位基因的单核苷酸变异(rs63751099,C>A/T)。
我们定义“等位基因分数”(allelic fraction,被观察基因座已突变的碱基数除以该基因座碱基总数) 量化在异质性肿瘤样本中突变等位基因的相对数量 [即:AF=(突变型等位基因的数目)/(突变型等位基因的数目+野生型等位基因的数目)]。全外显子组测序发现该患者肝癌组织基因组中MSH2基因无义突变的等位基因分数大小约为0.45(AF=14/(14+17),即含有突变型等位基因T的测序reads数为14,含有野生型等位基因C的测序reads数为17)。利用第一代Sanger测序确认了该无义突变仅发生在癌组织基因组而非癌旁正常组织。
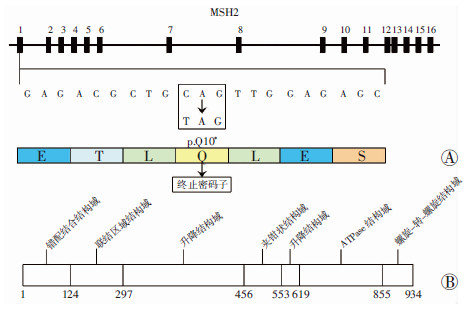 |
| A:MSH2基因结构;该无义突变位于第一外显子,导致第10位的CAG该突变的坐标如图所示;B:该无义突变对应的MSH2编码蛋白结构域的位置图 1 发生在原发性肝细胞肝癌患者癌基因组MSH2基因一个新的无义突变 |
利用ddPCR在基因组水平定性及定量地验证无义突变。图 2为肝癌组织和肝硬化组织中基因组DNA样本中各个液滴的二维图,图中绘制出荧光强度和液滴数的对比。从通道一(FAM,突变等位基因)和通道二(VIC,野生型等位基因)分别观察了各个液滴的荧光。结果确认了肝癌组织中的基因组DNA中存在突变等位基因,而未发生在肝硬化组织。在微滴产生过程中,由于同一个液滴中加入了两个靶基因(野生型等位基因和突变等位基因),可见肝癌组织基因组DNA中存在两种类型的阳性液滴。肝癌组织中基因组DNA样本中的液滴数经量化为:VIC(+)FAM(+)75,VIC(+)FAM(-)1496,VIC(-)FAM(+)973,VIC(-)FAM(-)12709;癌旁硬化组织中基因组DNA样本中的液滴数经量化为:VIC(+)FAM(+)0,VIC(+)FAM(-)2899,VIC(-)FAM(+)0,VIC(-)FAM(-)11384。估计了癌基因组突变等位基因 的等位分数为0.4 [AF=(973+75)/(973+75+1496+75),FAM阳性液滴数973,VIC阳性液滴数1496,FAM和VIC双阳性液滴数75]。我们比较了通过全外显子测序和ddPCR估计的突变等位基因分数,发现二者没有显著差别 (P=0.69)。ddPCR扩增形成的微滴见图 3,可见ddPCR系统生成的阴性微滴和阳性微滴区分明显,系统可准确判读阳性微滴和阴性微滴数目。
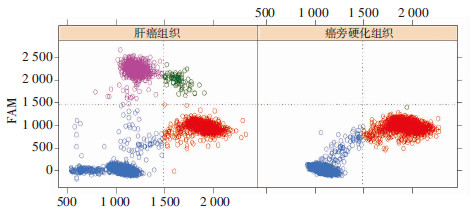 |
| 蓝色代表FAM和VIC呈双阴性;红色代表VIC阳性;粉色代表FAM阳性;绿色代表FAM和VIC呈双阳性图 2 肝癌组织和癌旁硬化组织中基因组DNA液滴情况 |
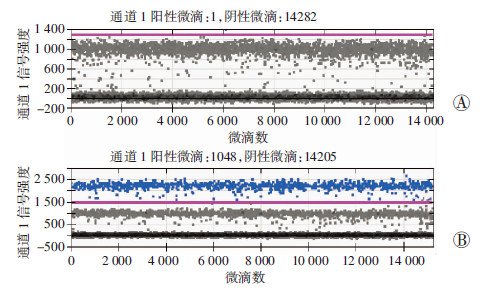 |
| A:ddPCR对癌旁硬化组织的检测结果;B:ddPCR对癌组织的检测结果 蓝色信号点代表发生PCR扩增的微滴,系统判读为阳性;黑色的信号点代表未发生PCR扩增的微滴,系统判读为阴性图 3 微滴数字PCR测定MSH2无义突变 |
随着下一代测序(next generation sequencing,NGS)技术的广泛应用,如何准确、精确地验证NGS所获得的结果,包括基因组改变,如SNP、突变和拷贝数变异,已经成为现今基因组改变检测技术研究的一个关键点[7]。在基因组突变的验证中,传统的qRT-PCR是公认的经典方法,因此在基因组变异的验证分析中应用广泛。在本研究中,我们也使用qRT-PCR技术定量验证了从原发性肝细胞肝癌病人外显子组测序数据识别的DNA错配修复基因MSH2的无义突变 (rs63751099,g.chr2:47630358 C>T)。在实验的过程中,发现qRT-PCR的分析步骤还是较为烦琐,该技术以相对浓度量化癌组织及癌旁硬化组织中突变的拷贝数,存在灵敏度不强,不能绝对量化拷贝数目。目前,qRT-PCR的这些缺点表明其可能不是理想的基因组变异的验证分析方法[8]。
鉴于传统方法存在的劣势,本研究使用ddPCR技术定性以及定量验证了该原发性肝细胞肝癌病人外显子组测序数据识别的DNA错配修复基因MSH2的无义突变 (rs63751099,g.chr2:47630358 C>T)。比较ddPCR与常规定量PCR检测方法,观察到常规定量PCR依赖于标准曲线来进行定量,通过标准曲线计算出待检测样品中目标基因的量,并不是真正意义上的定量;而ddPCR是不依赖于标准曲线的真正的定量,它可以精确的计数待检测样品中目标基因的量。常规定量PCR在目标基因含量很低的时候可能检测不出来,而ddPCR的检测下限是1/100 000,所以ddPCR可用于肿瘤的早期筛选,胎儿的畸形筛查等。ddPCR结果的精确度,准确性和灵敏度更佳[9],适合应用于拷贝数变异(CNV)、突变检测和基因相对表达研究等[3]。本研究使用的QX100 ddPCR平台,其油包水微滴体积为0.86 nL,直径125 μm。尽管是纳米级微滴,但是仍然比生物大分子大很多,甚至可以包裹进完整的细胞,因此在ddPCR平台上,样本基因组DNA的片段化并不是必需的。例如运用ddPCR开展chromosomal phasing实验,就要求gDNA样本保持完整不降解不断裂,使得长达200 kb的gDNA链直接包裹进W/O微滴中[10]。本研究使用的ddPCR体系微滴生成稳定,能够用于MSH2无义突变的分析。ddPCR在结果分析时仅是有/无两种扩增状态,不需要检测荧光信号与设定阈值线的交点,即完全不需要依赖于Ct值的鉴定,因此ddPCR的反应效率受扩增效率的影响大大降低,对PCR反应抑制物的耐受能力也大大提高。
由于ddPCR的绝对定量检测,可直接统计得出验证的MSH2无义突变位点(rs63751099,g.chr2:47630358 C>T)的突变率。在绝对计数时如果直接计数阳性微滴的个数来代表样本DNA的含量,会因为多个靶标DNA分子进入一个微滴,而使得阳性微滴的个数不等于模板含量。要使95%的阳性微滴中都仅含有1个拷贝的靶标DNA分子,则必须控制阳性微滴的比例小于10%。即使是这样,直接计数阳性微滴个数来代表靶标DNA拷贝数,仍然具有5%的误差。此外还会大大降低数字PCR的动态范围,增加检测成本。因此任何现代数字PCR平台都不是直接计数阳性微滴的个数来代表靶标DNA的拷贝数的,并不需要确保微滴中不含或仅含有一个拷贝的靶标DNA分子,就是说阴性微滴中不含靶标DNA分子,而阳性微滴中含有至少1个拷贝的靶标DNA分子。此外本研究使用的ddPCR体系,引物探针的终浓度远高于qPCR,引物终浓度高达900 nmol/L,探针终浓度为450 nmol/L,就是为了保证引物和探针在各微滴反应器中均匀分布。ddPCR技术所需样本量很少,对于临床研究来说具有极其重要的意义,尤其是当临床病理样本量太少时,具有极高拷贝数分辨精度的ddPCR可以进行突变检测,辅助诊断对药物的敏感性。在肿瘤异质细胞内,存在少量突变,这种突变的特定等位基因是否能够被准确检测到,直接影响到肿瘤靶向治疗的有效性,而ddPCR在稀有变异检测过程中发挥着重要作用,是临床肿瘤样本靶向药物分子检测的首选技术。
MSH2为DNA错配基因家族的一员,可修复DNA复制及逃避校对过程中出现错配[11]。MSH2在Lynch综合征(遗传性非息肉性结肠直肠癌)中突变率较高。Lynch综合征患者其他器官,包括肝脏的癌症风险也会增加。癌症体细胞突变数据库中未报道该MSH2基因的无义突变(COSMIC,version 69,http://cancer.sanger.ac.uk/cancergenome/projects/cosmic/)[12]。细胞DNA容易遭受细胞内活性物质以及环境因素的影响从而造成损伤,通过不同修复机制,可将DNA损伤引起的中毒和突变的影响降到最低[13]。当前研究表明,DNA修复能力降低与患癌风险增加有关[14]。
近年来,不断有新方法用于基因突变的分析。如之前我们应用高通量测序的方法分析得到了MSH2中rs63751099的无义突变,这与ddPCR的分析结果保持一致。但是高通量测序分析成本较高,对数据的分析要求也高。从便捷性和经济性的角度来看,ddPCR技术是一种快速和准确的分析验证突变的方法。
| [1] | Vogelstein B, Kinzler K W. Digital PCR[J]. Proc Natl Acad Sci USA, 1999, 96:9236-9241. |
| [2] | Hindson B J, Ness K D, Masquelier D A, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Anal Chem, 2011, 83(22):8604-8610. |
| [3] | Sedlak R H, Jerome K R. Viral diagnostics in the era of digital polymerase chain reaction[J]. Diagn Microbiol Infect Dis, 2013, 75(1):1-4. |
| [4] | Chen R, Mias G I, Li-Pook-Than J, et al. Personal omics profiling reveals dynamic molecular and medical phenotypes[J]. Cell, 2012, 148(6):1293-1307. |
| [5] | Carter S L, Cibulskis K, Helman E, et al. Absolute quantification of somatic DNA alterations in human cancer[J]. Nat Biotechnol, 2012, 30(5):413-421. |
| [6] | Bustin S A, Nolan T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction[J]. J Biomol Tech, 2004, 15(3):155-166. |
| [7] | Zhang D, Guo J. The development and standardization of testing methods for genetically modified organisms and their derived products[J]. J Integr Plant Biol, 2011, 53(7):539-551. |
| [8] | Hayden R T, Gu Z, Ingersoll J, et al. Comparison of droplet digital PCR to real-time PCR for quantitative detection of cytomegalovirus[J]. J Clin Microbiol, 2013, 51(2):540-546. |
| [9] | Baker M. Digital PCR hits its stride[J]. Nat Methods, 2012, 9(6):541-544. |
| [10] | Regan J F, Kamitaki N, Legler T, et al. A rapid molecular approach for chromosomal phasing[J]. PLoS One, 2015, 10(3):e0118270. |
| [11] | Kunkel T A, Erie D A. DNA mismatch repair[J]. Annu Rev Biochem, 2005, 74:681-710. |
| [12] | Forbes S A, Bindal N, Bamford S, et al. COSMIC:mining complete cancer genomes in the Catalogue of Somatic Mutations in Cancer[J]. Nucleic Acids Res, 2011, 39(Database issue):D945-D950. |
| [13] | Wood R D, Mitchell M, Lindahl T. Human DNA repair genes, 2005[J]. Mutat Res, 2005, 577(1/2):275-283. |
| [14] | Hiyama T, Yoshihara M, Tanaka S, et al. Genetic polymorphisms and esophageal cancer risk[J]. Int J Cancer, 2007, 121(8):1643-1658. |



