酪酸梭菌(Clostridium butyricum,CB)是一种革兰阳性厌氧杆菌,可以抵抗高热和强酸[1],被用于调节肠道菌群的平衡,发酵葡萄糖变为乙酸和丁酸,提高人体免疫力。酪酸梭菌不仅对便秘有很好的效果[2],还可以作为临床药物,治疗常见腹泻及抗生素相关性腹泻[3]。
长链非编码RNA(lncRNAs),是非编码RNA中长度超过200个核苷酸的一个亚组。lncRNAs作为基因调节因子,作用方式多种多样,可通过表观遗传学修饰、RNA-蛋白质相互作用、染色体转位和基因融合等多种方式参与细胞生长及增殖等调控过程[4, 5]。新进研究[6, 7]揭示lncRNAs在转录和翻译后的水平中发挥复杂和重要的作用,通过调节癌细胞生长和增殖的方式,对癌症的发生、发展起抑制作用。lncRNAs在结肠癌、肝癌等肿瘤发生的各个环节,包括早期癌变事件,肿瘤的生长、侵袭、迁移和血管生成等[8, 9, 10, 11]发挥或促进或抑制的调节作用。
人体益生菌酪酸梭菌与结肠癌[12, 13]细胞共培养能否刺激结肠癌细胞的lncRNAs表达发生变化,能否调节结肠癌的发生、发展过程仍然不清楚。本研究分析益生菌酪酸梭菌刺激结肠癌细胞后表达发生变化的 lncRNAs和mRNAs,探索酪酸梭菌对结肠癌细胞的作用。
1 材料与方法 1.1 细胞株、菌种、试剂和主要仪器结肠癌细胞株SW480(#CCL-228,ATCC)保存于重庆医科大学检验医学院临床检验诊断学教育部重点实验室,酪酸梭菌保存于本实验室,RPMI1640、胎牛血清(美国HyClone公司),TRIzol(美国Gibco公司),流式细胞仪、Annexin V-FITC/PI试剂盒(美国BD公司),倒置显微镜(德国LEICA公司),Hoechst 33258(中国碧云天公司)。
1.2 方法 1.2.1 细菌培养采用液体培养基厌氧培养酪酸梭菌(Clostridium butyricum,JX297227 Genbank)[14],取对数生长期的酪酸梭菌并经2 mL RPMI1640培养基稀释为1.5×107CFU/mL后,用于后续实验。
1.2.2 细胞培养和酪酸梭菌共培养SW480人结肠癌细胞用含有10%胎牛血清的RPMI1640培养基(100 U/mL青霉素和100 U/mL链霉素),在常规细胞培养箱中,与SW480细胞共培养的酪酸梭菌最适浓度为1.5×107 CFU/mL,处理时间为48 h[14]。SW480+CB组:结肠癌细胞SW480与1.5×107 CFU/mL酪酸梭菌共培养48 h;SW480对照组:结肠癌细胞SW480未加入酪酸梭菌培养48 h。
1.2.3 RNA提取与芯片分析采用TRIzol提取细胞的总RNA,用安捷伦2100 (Agilent technologies,Santa Clara,CA,US)检测RNA质量合格后(RIN均大于8.5),用RNeasy mini kit纯化total RNA。按照Agilent芯片配套提供的杂交标准流程和配套试剂盒,进行逆转录和单色荧光标记后,与安捷伦的人lncRNAs芯片和全基因芯片Agilent Human lncRNA 4*180K芯片(design ID:060228)进行杂交。使用安捷伦芯片扫描仪(Cat#G2565CA)扫描得到芯片荧光照片,并用配套的信号提取软件得到原始数据。芯片原始数据经Gene Spring Software 11.0采用四分位数法归一化处理后,比较两组差异lncRNAs和mRNAs。
1.2.4 lncRNAs靶标预测通过两种方法预测表达差异lncRNAs的潜在靶基因:顺式或反式[15]。在DNA水平上,lncRNAs顺式靶基因是包括lncRNAs的 10 kb窗口上游或下游的基因[根据UCSC注解(http: //genome.ucsc.edu/)]。在RNA水平,筛选lncRNAs反式作用的靶基因,利用BLAST软件分析lncRNAs与靶基因mRNA序列互补性,同时使用RNAplex软件预测两者结合的稳定性[16]。
1.2.5 信号通路富集度分析差异表达的mRNA及lncRNAs的预测靶基因由数据库注释,用功能注释聚类工具(DAVID v6.7)进行可视化和集成的信号通 路富集度分析[17],P<0.05认为是有差异的信号通路。
1.2.6 实时荧光定量PCR(qRT-PCR)检测使用两种方法得到目的基因的特异性引物:①根据NCBI的Primer-Blast在线软件设计引物并验证其特异性[18]。②从Primer bank里面找到对应引物并Blast验证其特异性[19]。
1.2.7 凋亡小体染色和流式细胞仪检测采用双苯并咪唑Hoechst 33258(bisbenzimlde)和Annexin V-FITC/PI染色分别检测细胞凋亡:①根据碧云天公司的说明书处理细胞,并用Hoechst 33258染色溶液染核,然后在荧光显微镜下拍照。②按照1.2.1所述处理细胞,并采用流式细胞仪检测[20]。
1.3 统计学分析采用GraphPad Prism 6.0统计软件,计量资料用x±s 表示,行t检验。P<0.05表示差异有统计学意义。
2 结果 2.1 lncRNAs芯片检测SW480共培养酪酸梭菌后lncRNAs 的变化与SW480对照组相比,SW480+CB组中表达差异大于5倍的上调lncRNAs有50个,下调lncRNAs有152个(图 1)。这些表达差异的lncRNAs可能参与SW480对共培养酪酸梭菌的应答。
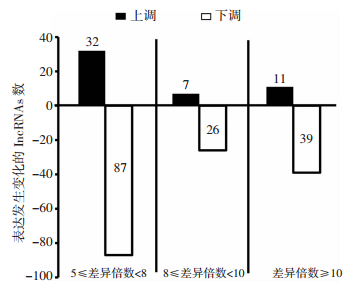 |
| 图 1 lncRNAs芯片检测出的表达变化的lncRNAs数 |
分析安捷伦芯片原始数据,发现738个上调、1 088个下调的mRNA基因。为了验证基因芯片结果,采用实时荧光定量qRT-PCR检测随机选择的6个表达变化的基因(CIDEA、ATM、IL8、CDKN1A、PGF、GADD45A)。qRT-PCR数据显示与芯片数据一致(图 2),证明芯片数据可靠。
 |
| 1:CIDEA;2:ATM;3:IL-8;4:CDKN1A;5:PGF;6:GADD45A 图 2 实时荧光定量PCR验证芯片结果 |
为检测这些表达变化基因及其相关信号通路的功 能,用DAVID v6.7对表达变化的基因进行信号通路富集度分析。显著改变的信号通路(P<0.05,图 3)包括MAPK信号传导、细胞因子-细胞因子、JAK-STAT信号通路、mTOR信号通路、NF-κB信号通路、VEGF信号通路和p53信号通路。
 |
| 图 3 表达差异的mRNAs信号通路富集度分析后差异显著的信号通路 |
通过预测和分析发现多个信号传导途径(图 4),如MAPK信号通路、细胞因子-细胞因子、mTOR信号通路、VEGF信号通路与表达变化的mRNAs的聚集相关信号通路一致,表明发生改变的信号通路可能是SW480对共培养的酪酸梭菌应答的关键信号通路。这些信号传导通路与早期癌发生,肿瘤生长、侵袭、迁移和血管生成有关。表明表达发生变化的lncRNAs可通过调节预测靶基因及其相关的信号通路来调控SW480对共培养酪酸梭菌的应答。
 |
| 1:MAPK信号通路;2:细胞因子与细胞因子受体的相互作用;3:系统性红斑狼疮;4:GnRH信号通路;5:病毒性心肌炎;6:mTOR信号通路;7:VEGF信号通路;8:造血细胞谱系;9:ErbB信号通路;10:移植物抗宿主病;11:Ⅰ型糖尿病;12:血管平滑肌收缩;13:心脏肌肉收缩;14:粘着斑 图 4 表达变化的lncRNAs靶基因信号通路富集度分析后 差异显著的信号通路 |
通过试剂双苯并咪唑Hoechst 33258(bisbenzimlde) 染色和荧光显微镜观察,SW480对照组细胞呈现圆形、细胞核均匀着色,而SW480+CB组与酪酸梭菌共培养24 h的SW480细胞呈现致密浓染或碎块状致密浓染,培养48 h后,发现更多凋亡小体(图 5)。流式细胞检测结果显示与SW480对照组相比,SW480+CB组凋亡率显著提高(P<0.01,图 6)。
 |
| 图 5 荧光显微镜观察凋亡小体染色共培养酪酸梭菌24、48 h后的SW480细胞及其对照 |
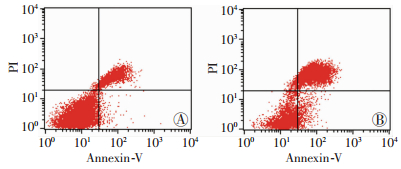 |
| A:SW480+CB组;B: SW480对照组 图 6 流式细胞仪检测2组SW480细胞凋亡率 |
酪酸梭菌是存在于人体肠道中的益生菌,在调节肠道菌群平衡和治疗抗生素性腹泻等方面已多有报道[21]。酪酸梭菌活菌制剂广泛用于临床中肠道相关疾病的治疗或辅助治疗。然而,酪酸梭菌与结肠癌相关的报道鲜见,酪酸梭菌能否通过调节结肠癌细胞表达层面lncRNAs和mRNAs的变化从而阻碍结肠癌的发生、发展鲜为人知。因此我们设计了本实验,以期找到酪酸梭菌有利于结肠癌防治的基础理论依据。
提取与酪酸梭菌共培养结肠癌SW480细胞与对照组SW480细胞的总RNA,利用安捷伦最新的lncRNAs/mRNAs芯片,系统分析SW480+CB组和SW480对照组的差异表达lncRNAs 和mRNAs,最终确定了lncRNAs中表达差异倍数大于5倍的50个上调和152个下调的lncRNAs,以及mRNA中表达差异倍数大于2倍的738个上调和1 088个下调的mRNA。这些基因中的部分基因和Arimochi等[21]报道的与细胞凋亡相关的基因高度相关。他们发现酪酸梭菌条件培养基里含有新的诱导细胞凋亡的物质,并证实其能诱导结肠癌细胞死亡。
DAVID v6.7被用来评估表达差异的lncRNAs和mRNA影响的信号通路。其结果显示,有多条信号通路受到影响,其中一些明显受到影响(P<0.05)的信号通路,与目前已报到的文献相一致,如NF-κB通路、ATM通路和mTOR途径已被发现在人体对细菌感染的应答中起着关键作用;NF-κB通路被证实通过激活TLR4促进TNF-α、IL-6和IL-12p40的免疫应答[22];
Fedor等[23]发现ATM-CHK2信号通路诱导细胞凋亡;Tattoli等[24]发现抑制mTOR活性,能激活宿主的关键防御机制-自噬,进而诱导侵袭性细菌的降解。在这些受到显著影响的信号通路中,我们还发现了p53信号通路、ATM信号通路和mTOR途径等与细胞凋亡高度相关的信号通路,因此我们提出假设,即酪酸梭菌与结肠癌SW480细胞共培养,能促进结肠癌SW480细胞的凋亡。采用荧光染色和流式细胞术两个功能试验验证SW480细胞凋亡的存在,其结果与芯片数据的信号通路分析结果相符。
综上所述,本研究初步证实酪酸梭菌能通过调节结肠癌SW80细胞lncRNAs水平的表达变化,从而引起结肠癌细胞凋亡,为进一步分析酪酸梭菌引起结肠癌细胞凋亡的关键基因和主要机制提供了依据。
| [1] | Collins M D, Lawson P A, Willems A, et al. The phylogeny of the genus Clostridium: proposal of five new genera and eleven new species combinations[J]. Int J Syst Bacteriol, 1994, 44(4): 812-826. |
| [2] | Imase K, Takahashi M, Tanaka A, et al. Efficacy of Clostridium butyricum preparation concomitantly with Helicobacter pylori eradication therapy in relation to changes in the intestinal microbiota[J]. Microbiol Immunol, 2008, 52(3): 156-161. |
| [3] | McFarland L V. Meta-analysis of probiotics for the prevention of antibiotic associated diarrhea and the treatment of Clostridium difficile disease[J]. Am J Gastroenterol, 2006, 101(4): 812-822. |
| [4] | Cesana M, Cacchiarelli D, Legnini I, et al. A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J]. Cell, 2011, 147(2): 358-369. |
| [5] | Xing Z, Lin A, Li C, et al. lncRNA directs cooperative epigenetic regulation downstream of chemokine signals[J]. Cell, 2014, 159(5): 1110-1125. |
| [6] | Liu Q, Huang J, Zhou N, et al. LncRNA loc285194 is a p53-regulated tumor suppressor[J]. Nucleic Acids Res, 2013, 41(9): 4976-4987. |
| [7] | Lau E. Non-coding RNA: Zooming in on lncRNA functions[J]. Nat Rev Genet, 2014, 15(9): 574-575. |
| [8] | Peter S, Borkowska E, Drayton R M, et al. Identification of differentially expressed long noncoding RNAs in bladder cancer[J]. Clin Cancer Res, 2014, 20(20): 5311-5321. |
| [9] | Kallen A N, Zhou X B, Xu J, et al. The imprinted H19 lncRNA antagonizes let-7 microRNAs[J]. Mol Cell, 2013, 52(1): 101-112. |
| [10] | Yuan J H, Yang F, Wang F, et al. A long noncoding RNA activated by TGF-beta promotes the invasion-metastasis cascade in hepatocellular carcinoma[J]. Cancer Cell, 2014, 25(5): 666-681. |
| [11] | 郭勤浩,赵岩,陈洁静,等.BRAF激活的长链非编码RNA在结直肠癌中的表达及功能[J].中华消化外科杂志,2014,13(5):376-380. |
| [12] | Allemani C, Weir H K, Carreira H, et al. Global surveillance of cancer survival 1995-2009: analysis of individual data for 25, 676, 887 patients from 279 population-based registries in 67 countries (CONCORD-2)[J]. Lancet, 2015, 385(9972): 977-1010. |
| [13] | 陈万青, 郑荣寿, 曾红梅, 等. 2011年中国恶性肿瘤发病和死亡分析[J]. 中国肿瘤, 2015, 24(1): 1-10. |
| [14] | 杨金梅, 张德纯, 李科, 等. 酪酸梭菌诱导结肠癌 SW-480 细胞凋亡及作用机制[J]. 第三军医大学学报, 2013, 35(22): 2448-2451. |
| [15] | Hu P, Yang J, Hou Y, et al. LncRNA expression signatures of twist-induced epithelial-to-mesenchymal transition in MCF10A cells[J]. Cell Signal, 2014, 26(1): 83-93. |
| [16] | Tafer H, Hofacker I L. RNAplex: a fast tool for RNA-RNA interaction search[J]. Bioinformatics, 2008, 24(22): 2657-2663. |
| [17] | Huang-da W, Sherman B T, Lempicki R A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44-57. |
| [18] | Sayers E W, Barrett T, Benson D A, et al. Database resources of the National Center for Biotechnology Information[J]. Nucleic Acids Res, 2012, 40(Database issue): D13-D25. |
| [19] | Wang X, Spandidos A, Wang H, et al. PrimerBank: a PCR primer database for quantitative gene expression analysis, 2012 update[J]. Nucleic Acids Res, 2012, 40(Database issue): D1144-D1149. |
| [20] | Peng Q, Zhao L, Hou Y, et al. Biological characteristics and genetic heterogeneity between carcinoma-associated fibroblasts and their paired normal fibroblasts in human breast cancer[J]. PLoS One, 2013, 8(4): e60321. |
| [21] | Arimochi H, Morita K, Nakanishi S, et al. Production of apoptosis-inducing substances from soybean protein by Clostridium butyricum: characterization of their toxic effects on human colon carcinoma cells[J]. Cancer Lett, 2009, 277(2): 190-198. |
| [22] | Hildebrand D, Sahr A, Wolfle S J, et al. Regulation of Toll-like receptor 4-mediated immune responses through Pasteurella multocida toxin-induced G protein signalling[J]. Cell Commun Signal, 2012, 10(1): 22. |
| [23] | Fedor Y, Vignard J, Nicolau-Travers M L, et al. From single-strand breaks to double-strand breaks during S-phase: a new mode of action of the Escherichia coli Cytolethal Distending Toxin[J]. Cell Microbiol, 2013, 15(1): 1-15. |
| [24] | Tattoli I, Sorbara M T, Vuckovic D, et al. Amino acid starvation induced by invasive bacterial pathogens triggers an innate host defense program[J]. Cell Host Microbe, 2012, 11(6): 563-575. |



